DNA fragments detected in monovalent and bivalent Pfizer/BioNTech and Moderna modRNA COVID-19 vaccines from Ontario, Canada: Exploratory dose response relationship with serious adverse events.
David J. Speicher1、Jessica Rose2、L. Maria Gutschi3、David Wiseman4、Kevin McKernan5
4米国テキサス州ダラス、サイネキオン社 ORCID 0000-0002-8367-6158
コレスポンディング・オーサー デヴィッド・J・シュパイヒャー博士 ゲルフ大学
2023年10月19日
キーワード
COVID-19、ワクチン、DNA汚染、不純物、残留DNA、modRNA、mRNA、有害事象
要旨
背景
SARS-CoV-2ワクチン用のヌクレオシド修飾RNA(modRNA)の作製に用いられる試験管内試験転写(IVT)反応は、現在、DNA鋳型から転写するRNAポリメラーゼに依存している。オリジナルのファイザー無作為化臨床試験(RCT)で使用されたmodRNAの製造には、PCRで生成されたDNA鋳型が使用された(プロセス1)。数十億のワクチン用量を生成するために、このDNAを細菌のプラスミドベクターにクローニングし、大腸菌で増幅してから直鎖化した(プロセス2)。モデルナは、臨床試験用ワクチンと臨床試験後使用ワクチンの両方に、同様のプラスミドベースの工程を使用していたようである。最近、DNAシークエンシング研究により、ファイザー・バイオエヌテックワクチンとモデルナmodRNAワクチンの両方において、このプラスミドDNAが有意なレベルで存在することが明らかになった。これらの研究は限られたロットを調査したものであり、国際的に観察された残存DNAのばらつきについては疑問が残る。
方法
すでに公表されているプライマーとプローブの配列を用い、カナダで入手した12ロット(モデルナ小児/成人一価ワクチン5ロット、モデルナ成人二価ワクチンBA.4/5 1ロット、モデルナ成人二価ワクチンBA.4/5 1ロット)のmRNAバイアル27本について、定量ポリメラーゼ連鎖反応(qPCR)とQubit®蛍光測定を行った。 /5、モデルナ小児・成人2価BA.1 1ロット、モデルナXBB.1.5 1価1ロット、ファイザー成人1価3ロット、ファイザー成人2価BA.4/5 1ロット)。ワクチン有害事象報告システム(VAERS)データベースは、試験した各ロットについて報告された有害事象(AE)の数と分類について照会した。ファイザー社製COVID-19ワクチンの1バイアルについて、DNA断片のサイズ分布を調べるため、Oxford Nanoporeシークエンシングを行った。このサンプルはまた、残存DNAが脂質ナノ粒子(LNP)内にパッケージされDNaseIに耐性があるのか、あるいはDNAがLNPの外側に存在しDNaseIに不安定なのかを決定するために使用された。
結果
プラスミド複製起点(ori)およびスパイク配列の定量サイクル(Cq)値(1:10希釈)は、ファイザーではそれぞれ18.44~24.87および18.03~23.83、モデルナではそれぞれ22.52~24.53および25.24~30.10であった。これらの値は、qPCRで測定したオリおよびスパイクのそれぞれについて、0.28~4.27ng/doseおよび0.22~2.43ng/dose(ファイザー)、0.01~0.34ng/doseおよび0.25~0.78ng/dose(モデルナ)に相当し、Qubit®蛍光光度計で測定したファイザーおよびモデルナのそれぞれについて、1,896~3,720ng/doseおよび3,270~5,100ng/doseに相当する。SV40プロモーター-エンハンサー-オリは、Cqスコアが16.64~22.59のファイザーバイアルでのみ検出された。探索的分析において、1回投与あたりのDNA量と重篤な有害事象(SAE)の頻度との用量反応関係の予備的証拠を発見した。この関係はファイザー社とモデルナ社で異なっていた。サイズ分布解析の結果、DNA断片の平均長は214塩基対(bp)、最大長は3.5kbであった。プラスミドDNAはLNP内部に存在し、ヌクレアーゼから保護されていると考えられる。
結論
これらのデータは、これらのワクチンに1回あたり数十億から数千億のDNA分子が含まれていることを示している。フルオロメトリーを用いると、全てのワクチンはFDAとWHOが設定した残留DNAのガイドラインである10ng/用量を188~509倍超えている。しかしながら、qPCRによる残留DNA量はすべてのワクチンでこのガイドラインを下回っており、定量的ガイドラインを解釈する際の方法論の明確さと一貫性の重要性が強調された。qPCRによる残存DNA量とSAEの用量反応効果に関する予備的な証拠は、確認とさらなる調査の必要性を示している。
我々の知見は、ワクチンの安全性に関する既存の懸念を拡大し、LNPを用いた効率的なトランスフェクションが導入される前に考案されたガイドラインの妥当性に疑問を投げかけるものである。いくつかの明らかな限界があるが、われわれの研究を法医学的条件下で再現し、高効率DNAトランスフェクションおよび累積投与を考慮したガイドラインの改訂を強く要望する。
はじめに
一般用に大量の改変RNA(modRNA)ワクチンを製造するため、ファイザー社は無作為化臨床試験(RCT)1用の材料を製造するために使用していた製造工程(プロセス1)を、すでにモデルナ社が使用しているものと同様の工程(プロセス2)に変更した。SARS-CoV-2のスパイク配列は、大腸菌で活性のある細菌複製起点(一般にoriと呼ばれる)を含むプラスミドにクローニングされた。このプラスミド(ファイザー社では7,824塩基対(bp)、モデルナ社では6,777bp)はアミノグリコシドホスホトランスフェラーゼ遺伝子(Neo/Kan)も含んでおり、カナマイシンを含むブロス中でコスト効率のよい細菌複製を可能にし、37℃で30分ごとにプラスミドコピー数を倍増させる。その後、大腸菌細胞を回収し、溶解する。DNAを抽出し、制限酵素Eam1104Iで直鎖化する。この直鎖DNAは、N1-メチル-シュードウリジン存在下、T7 RNAポリメラーゼの試験管内試験転写(IVT)の鋳型となる。IVTの後、DNAは加水分解され、最終的な医薬品に含まれる割合が減少する。欧州医薬品庁(EMA)からリークされ、British Medical Journalに引用された文書2には、このプロセスで作られたmodRNA産物中の残存DNAが著しく変化する可能性があることが指摘されている3。
McKernanらは、これらのワクチンの次世代RNA配列決定を行ったところ、予想に反して、製造時に使用された発現プラスミドに由来するDNAの証拠を発見した4。さらにMcKernanらは、ファイザーとモデルナの両ワクチンに共通する配列を標的とするプライマーを用いて、DNA混入に対する定量的ポリメラーゼ連鎖反応(qPCR)法を開発した4。これらの知見の他のロットのワクチンへの一般化可能性を調査するため、カナダのオンタリオ州で流通していた期限切れの未開封バイアル24本(ファイザー8本、モデルナ16本)とモデルナXBB.1.5 COVID-19ワクチンの期限内残余バイアル3本を入手し、Qubit®蛍光光度法とスパイク、プラスミド・オリ、SV40プロモーター・エンハンサー・オリを標的としたqPCRで調べた。また、以前の研究(McKernan et al.4)の観察を拡張し、DNA断片のサイズ分布とワクチンのDNasel感度を調べることで、残存DNAがLNPにパッケージされているかどうかを調べた。
本研究では、「不純物」や「コンタミネーション」ではなく、「残存DNA」、「DNA量」(またはそれに類するもの)という用語を用いている。
試験方法
COVID-19ワクチンの試験
Pfizer-BioNTech BNT162b2(n=8)とモデルナ Spikevax mRNA-1273(n=16)の期限切れ未開封バイアルをカナダ、オンタリオ州の様々な薬局から入手した(図1)。また、同じロットのモデルナ XBB.1.5ワクチンの期限内残存バイアル3本も入手した。合計12ロットの27本のmRNAバイアルが調査された: 5ロットのモデルナ小児/成人一価ワクチン、1ロットのモデルナ成人二価ワクチンBA.4/5,1ロットのモデルナ小児/成人二価ワクチンWuhan-BA.1,1ロットのモデルナ XBB.1.5一価ワクチン、3ロットのPfizer成人一価ワクチン、1ロットのPfizer成人二価ワクチンWuhan-BA.4/5である。陰性対照として、パパベリン21.7mg/mLおよびフェントラミン1mg/mLと組み合わせたアルプロスタジル66mcg/mLの未開封注射用バイアル(TriMix)を用いた。未開封のバイアルは、ロット番号と有効期限が印刷された無傷のフリップオフプラスチックキャップが付いていたため、改ざんされていなかった。バイアルは薬局の+2-8℃の専用ワクチンユニットで保管され、冷凍ゲルパックとともに断熱容器に入れられ、5時間以内に試験室の冷蔵庫に入れられた。1つのモデルナバイアルのみ有効期限が印字されておらず、薬剤師によるスキャンが必要なQRコードがあった。モデルナ XBB.1.5のバイアルも同様に薬局で保管された。バイアルは冷蔵庫から取り出され、〜20分間温められ、薬剤師が〜30分かけて患者に投与した。残ったバイアルは冷凍ゲルパックと一緒に断熱容器に入れられ、12時間以内に検査室の冷蔵庫に運ばれた。

図1 カナダ・オンタリオ州のCOVID-19ワクチンバイアル:
(A)Pfizer/BioNTech BNT162b2成人1価および2価;モデルナ Spikevax mRNA-1273(B)成人1価XBB.1.5、(C)小児/成人1価、(D)小児/成人2価Wuhan-BA.1、(E)小児/成人2価Wuhan-BA.1および成人Wuhan-2価BA.4/5。
スパイク、オリ、およびSV40プロモーター-エンハンサー-オリDNAのqPCR分析
各バイアルは、プラスミド由来のSARS-CoV-2スパイク、ori、およびSV40プロモーター-エンハンサー-ori DNAの存在について定量的PCR(qPCR)で検査された。スパイクとプラスミドoriは、モデルナとファイザーの発現プラスミドが共有する配列をターゲットとするPCRプライマーを用いて二重に検査した(表1)。ユニプレックスSV40エンハンサーアッセイは、ファイザーベクター特有の核ターゲティング配列を増幅するように設計された6。qPCRキットはMedicinal Genomics社(Part# 420201, Beverly, USA)から入手した。マスターミックスには、3.8 µLのポリメラーゼ酵素、0.8 µLの反応バッファー、1.0 µLのPrimer-Probe mixからなる8.8 µLの反応液、および12.2 µLのddH20が含まれている。12.5μLの100μM oriプローブ、12.5μLの100μMスパイクプローブ、25μLの100μMスパイクフォワードプライマー、25μLの100μMスパイクリバースプライマー、25μLの100μM oriフォワードプライマー、25μLの100μM oriリバースプライマー、および75μLのddH20を用いてPrimer-Probe mixを組み立てた。
スパイクおよびori qPCRアッセイでは、濃度既知の合成gDNAコントロール(gBlock、Integrated DNA Technologies(IDT)社、米国サンディエゴ)を用いて、10倍連続希釈由来の検量線を作成した。SV40エンハンサーgBlockは最初の合成に失敗し、標準曲線を作成できなかった。
表1 spike、ori、SV40プロモーターを標的としたプライマーとプローブの配列
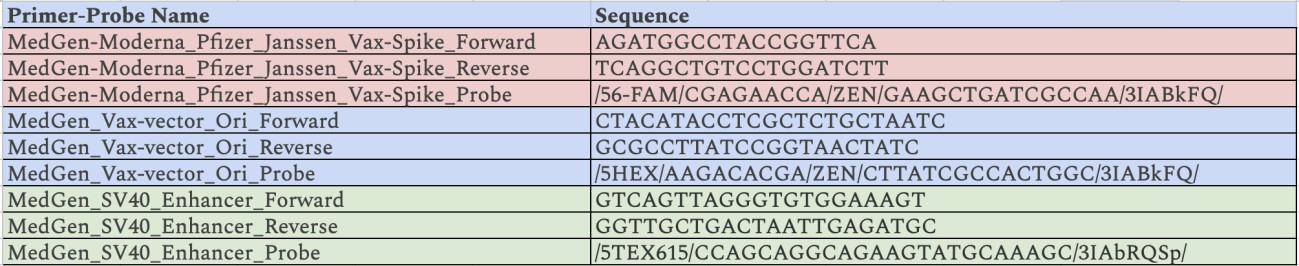
サイクリングはQuantStudio 3(ThermoFisher Scientific, Waltham, USA)を用い、95℃で3分間の初期変性後、95℃で10秒間、65℃で30秒間を35サイクル行った。Cqからng/µLへの変換は、QuantStudioソフトウェアv2.7.0(ThermoFisher Scientific)を用いて計算した。New England BioLabs DNA calculatorで求めたアンプリコンの質量7と長さ(oriは105bp、spikeは114bp)を用いて、プラスミドの長さ(ファイザーは7,824bp、モデルナは6,777bp)を調整し、存在するDNAの総ナノグラム(ng)を推定した。用量あたりのコピー数は、各筋肉内ワクチン注射の容量(ファイザーは300μL、モデルナは500μL)で調整した。残留DNA濃度が最も高かったファイザーの3ロットについて連続希釈を行った。qPCRは処理や抽出を行わずに直接行ったため、LNPによるPCR阻害を調べることができなかった。
Qubit®蛍光定量法
AccuGreen® HS 蛍光測定試薬(AccuGreen #9 9820 および DNA Quantification Buffer #9 9979)と標準物質は、Qubit®分析(ThermoFisher Scientific)用にBiotium社(米国、サンフランシスコ)から入手した。蛍光試薬(995μLのHS Bufferと5μLの200X AccuGreen色素から調製したストック190μL)を10μLのワクチンと一緒にボルテックスした。これらのサンプルを95℃で8分間、4℃で5分間加熱してLNPを破壊し、Fluorometric DyesがDNAにアクセスできるようにした。サンプルはQubit 3.0 Fluorometerを用い、製造者の指示に従って読み取った。Qubit FluorometryとqPCRのデータを比較した。
ワクチン有害事象報告システム(VAERS)データ
VAERS データベースの解析には、Rの Language and Environment for Statistical Computing パッケージ8を使用し、2020年 12月 17日から 2023年 10月 6日までのデータを使用した。VAERS データは、i) 各報告の一般データ、ii) 報告された。AE または「症状」、iii) ワクチン製造業者およびロット番号を含むワクチンデータの 3 つのカンマ区切り値(csv)データファイルでダウンロード可能である5。特定のワクチンに関連する。AEを評価するためには、VAERS IDをリンク変数として 3 つのデータファイルを統合する必要がある。本研究では COVID-19 製品を対象としているため、COVID-19 ワクチンタイプ(COVID19-1(1 価)、COVID19-2(2 価)のみを対象とした。その他の関連変数には、VAERS ID*、ワクチンロット(VAX_LOT)、ワクチン製造業者(VAX_MANU)、入院(HOSPITAL)、死亡(DIED)が含まれた。データはワクチンロットごとにグループ化され、AEおよびSAE報告の総数がカウントされた。SAE 報告には、死亡、入院、救急外来受診、障害報告、出生異常および生命を脅かす報告が含まれ、ロットごとの総死亡数など、MedDRA でコード化された個々の AEも集計された。
VAERSの様々な限界は、例えば、FDA9によって広く認められており、過少報告、誤情報告、自発的報告、因果関係を推論できないことなどが含まれる。それでもなお、残留 DNA 含有量と SAEの間の用量反応関係の可能性を検討するために、毒性学的影響の可能性の代用として、全 AE 数に対する。SAE 報告数の比率(「SAE 報告比率」=SRR)を用いた。この分母は推定が困難であるため、ロット別に報告された全 AE 数を総投与回数の代理とした。この原則は、CDCが、Proportional Reporting Ratio(PRR)10を用いて安全性シグナルを特定する不均衡性シグナル分析(DSA) で用いている。Evans らによって考案された。PRR は、ファーマコビジランスにおいて有用なツールであるが、既知の限界がある11。
VAERS は米国を拠点とするデータベースであるが、世界各国からの報告を受け入れていることに留意する必要がある。米国外の製造業者に報告される特定のカテゴリーの AE は、VAERS データベースに報告されなければならない。過少報告の傾向の違いや、米国内外の製造業者や医療専門家に課された報告義務の違いにより、SRRの推定に交絡が生じる可能性がある。従って、今回の探索的用量反応分析では、この交絡を減らすために、米国外のVAERSデータのみを使用した。さらに、VAERSデータセットのダウンロード版で得られたデータと、VAERS WONDERフロントエンド・ウェブベース・インターフェース(https://wonder.cdc.gov/controller/datarequest/D8)で得られたデータには、いくつかの不一致があった。ダウンロード版の方がウェブ版より詳細であるため、ダウンロード版を使用した。次に、残留DNA濃度と重篤な有害事象の報告頻度との関連を明らかにするために、SRRをバイアルから検出されたDNA濃度に対してプロットした。
ロット中に複数のバイアルが存在する場合は、そのロットの投与量あたりの平均残留DNA質量を使用した。任意のロットについて SRRのゼロ値は、世界中で、1 件以上の AEが確認された場合にのみプロットされた。曲線は対数軸にプロットし、Microsoft® Excelの線形関数を用いて傾向線を引いた。
オックスフォード・ナノポア・シーケンス
以前に配列決定したワクチン4(Pfizer小児用一価Lot# FL8095)を用いた別の実験では、DNA断片のサイズ分布を、Oxford Nanopore Flongle(R.10.4.1, Oxford Nanopore Technologies (ONT), New York, USA)とOxford Nanopore Ligation sequencing kit(SQK-LSK114)を用いて、製造元の指示に従って推定した。リードは、Burrow- Wheeler Aligner with maximum exact matches (BWA-MEM)を用いてNCBI OR134577.1にマッピングした12。ONTシーケンサーのリード長には制限がないが、DNA分離手順によって、ONTライゲーション反応で捕捉される分子の長さに偏りが生じることがある。BWA-MEMを用いて、1分子のリードをカウントし、マッピングされたリード長に従ってビニングした。
ワクチンのヌクレアーゼ感受性
同じバイアル(Pfizer Lot# FL8095)を用いて、混入DNAがLNPにパッケージされDNaseIに耐性があるか、あるいはDNAがLNPの外側に存在しDNaseIに不安定であるかを判定することにより、ワクチンのDNasel感受性を評価した。
ヌクレアーゼで保護されたDNAは、20μLのワクチンを2.5μLのDNaseI-XT(2 units/μL, NEB#M0570S, New England BioLabs Inc, Ipswich, USA)、2.5μLのGrim Reefer 10Xバッファー(Medicinal Genomics #4 20123-125)で処理し、37℃で30分間インキュベートすることで推定した。コントロールには、DNaseI-XTの代わりに2.5μLのddH20を用いた。2.5μLのMGC溶解バッファー(Medicinal Genomics #4 20001)を用いてDNaseI-XT反応を化学的に停止させた。DNaseI化学的停止ステップの後、qPCR増幅可能な内部コントロールDNAをスパイクインし、DNaseI-XTが完全に不活化されたことを確認した(Medicinal Genomics #4 20123-125)。
DNaseI不活性化コントロールをスパイクインした後、54μLのSenSATIVAx磁気ビーズ(Medicinal Genomics社製)を用いて、DNaseI-XTアッセイおよびDNaseI-XT陰性コントロールサンプルからDNAを精製した。磁気ビーズをサンプルとピペットで10回混合し、室温で5分間インキュベートした後、磁気的に分離し、70% v/vエタノールで2回洗浄した。エタノールを除去し、ビーズを室温で2分間乾燥させた。サンプルを30μLのddH20で溶出し、溶出液1μLを18.8μLの反応でスパイクとオリについてqPCRで調べた。DNaseI不活化コントロールプライマーとプローブ(CY5で0.5 µL)を追加し、合計19.3 µLの反応を行った。
結果
8-log連続希釈標準曲線を用いてサンプルのCq値を校正し、スパイクアンプリコンおよびオリアンプリコンについてそれぞれ0.998および0.999のR2値を得た。PCR
効率はスパイクで99.8%、oriで94.7%であった(図2)。すべてのプレートで、陰性コントロールと鋳型なし(ddH20)コントロール(NTC)を3連でテストし、陰性であることがわかった。
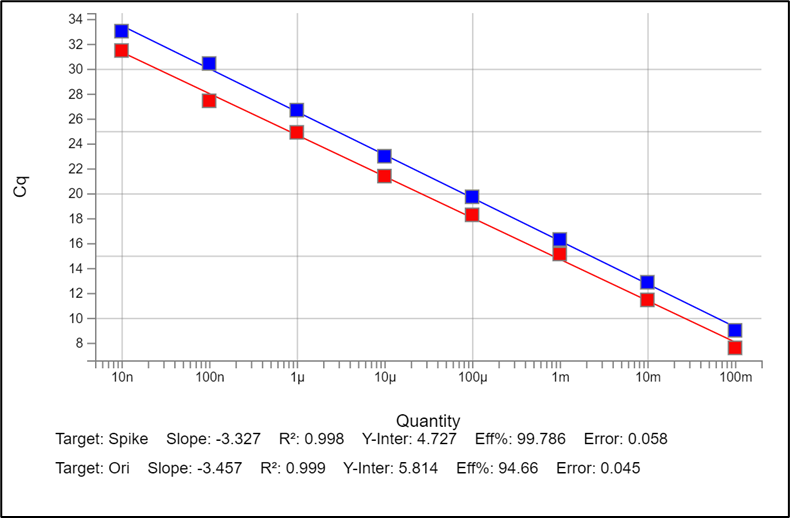
図2 スパイク(赤)とオリ(青)を10倍に希釈し、qPCRで試験した検量線
個々のバイアルについて、ファイザーのqPCRはスパイク、ori、SV40エンハンサー・プロモーター・ori(ΔCq 1.48±0.32)について同様の時間で増幅した(図3)。ファイザーのロットは別: FX4343を除き、ファイザー(スパイクCq 16.91±0.52、オリCq 16.91±1.07、SV40プロモーター-エンハンサー-オリCq 15.46±2.02)とモデルナ(スパイクCq 20.35±0.65、オリCq 25.34±1.47)のバイアル間の差は小さかった(値はバイアル原液に基づく)(表2、図4)。
しかしながら、ロットAS0467Dを除くすべてのモデルナバイアルについて、oriは一貫してCqをスパイクより5-6遅く増幅した。SV40プロモーター-エンハンサー-oriはすべてのファイザーバイアルで検出されたが、モデルナバイアルでは検出されなかった。
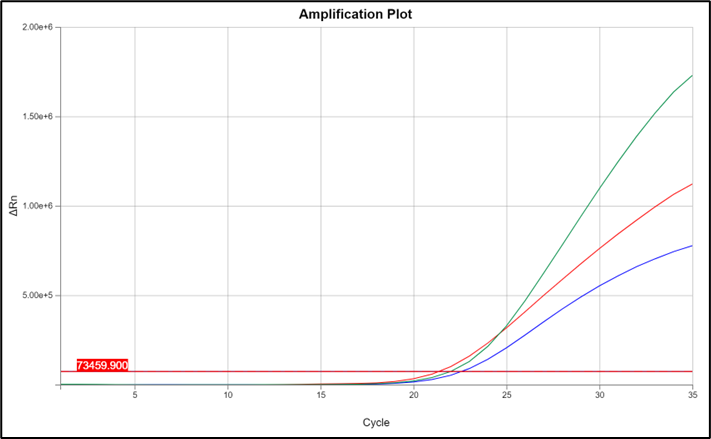
図3 ファイザー(Lot: Fx4343a)の1バイアルで、同じPCRランの2つの異なるウェルから得られたスパイク(赤)、オリ(青)、SV40エンハンサー-プロモーターリ(緑)の増幅曲線
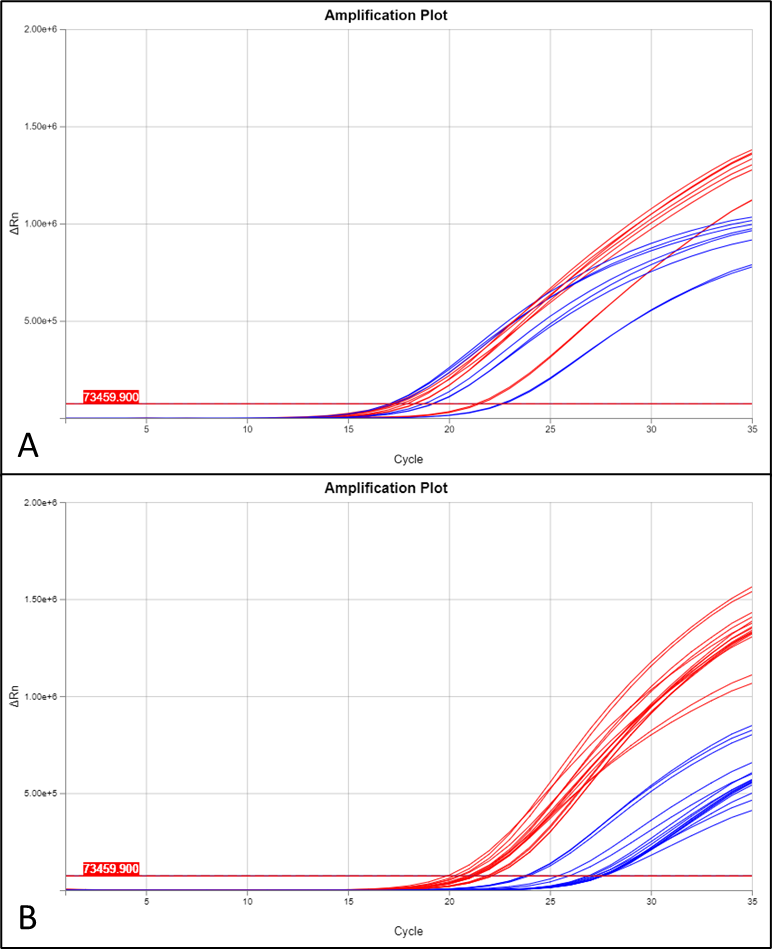
図4 ファイザー(A)とモデルナ(B)の全バイアルの増幅プロットは、ファイザーの個々のバイアルでスパイク(赤)とオリ(青)が同様に増幅したことを示している
モデルナでは、バイアル間の変動は一貫していたが、スパイクはoriよりも早く増幅した(ΔCq~6)。
表2 試験したPfizer-BioNTechおよびモデルナの全バイアルについて、ワクチンバイアルの詳細、確認された有害事象(AE)、SAおよびSV40プロモーター-エンハンサー-オリに関するqPCR試験結果 計算foは、それぞれ0.30mLおよび0.50mLの成人用量に基づいている。モデルナは6-12歳にも適応があり、投与量は0.25mLで、アンプリコンの長さ(105bpのオリ、114bpのスパイク)が6,777bpのモデルナプラスミドのほんの一部であることを考慮して調整した結果、総ng/投与量は成人投与量の半分となった。
*ロット番号の末尾の小文字は、同一ロットの異なるバイアルを示す。SV40。
モデルナおよびファイザーの3ロットのDNA含量は、スパイクまたはプラスミドオリのいずれについても1ng/用量を超えるものはなかった。これら3つのバイアル中のワクチンを10倍に連続希釈し、qPCRでLNP阻害を評価した(図5)。その結果、1:10 希釈後(1:10,1:100,1:1000)に予想される約 3.3 Cqの反応が観察されたことから、これらの希釈では DNAの定量に影響する。LNP 阻害があることが示唆された(図 6)。したがって、1:10希釈のデータをさらなる分析に使用した。この希釈は、一部の用量が使用前に希釈されるように設計されていたことと同様に、計算で考慮された。
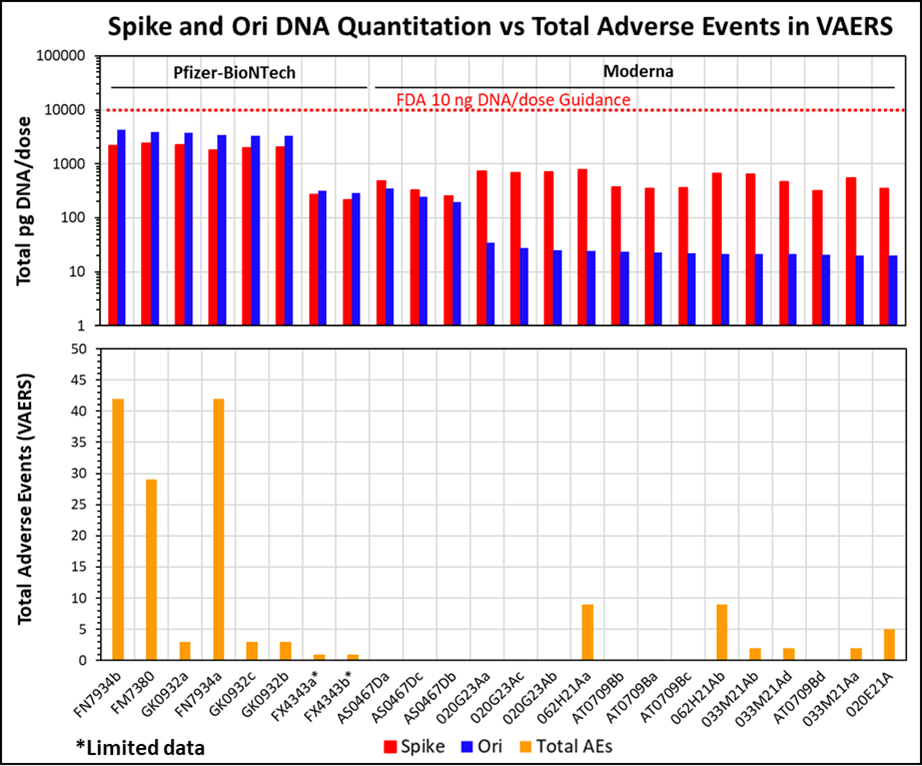
図5 スパイク(赤)とオリ(青)の残存DNA量とVAERSに報告された有害事象の総数(オレンジ)の比較
FDAおよびWHOの残留DNAに関する規制ガイドライン10 ng/dose1314を赤い点線で示す。バイアルはプラスミドオリのDNA負荷量の降順でソートされている。ロット番号の末尾の小文字は、同一ロットの異なるバイアルを示す。AEの総数はロットごとに決定し、同一ロットの各バイアルについて再現した。
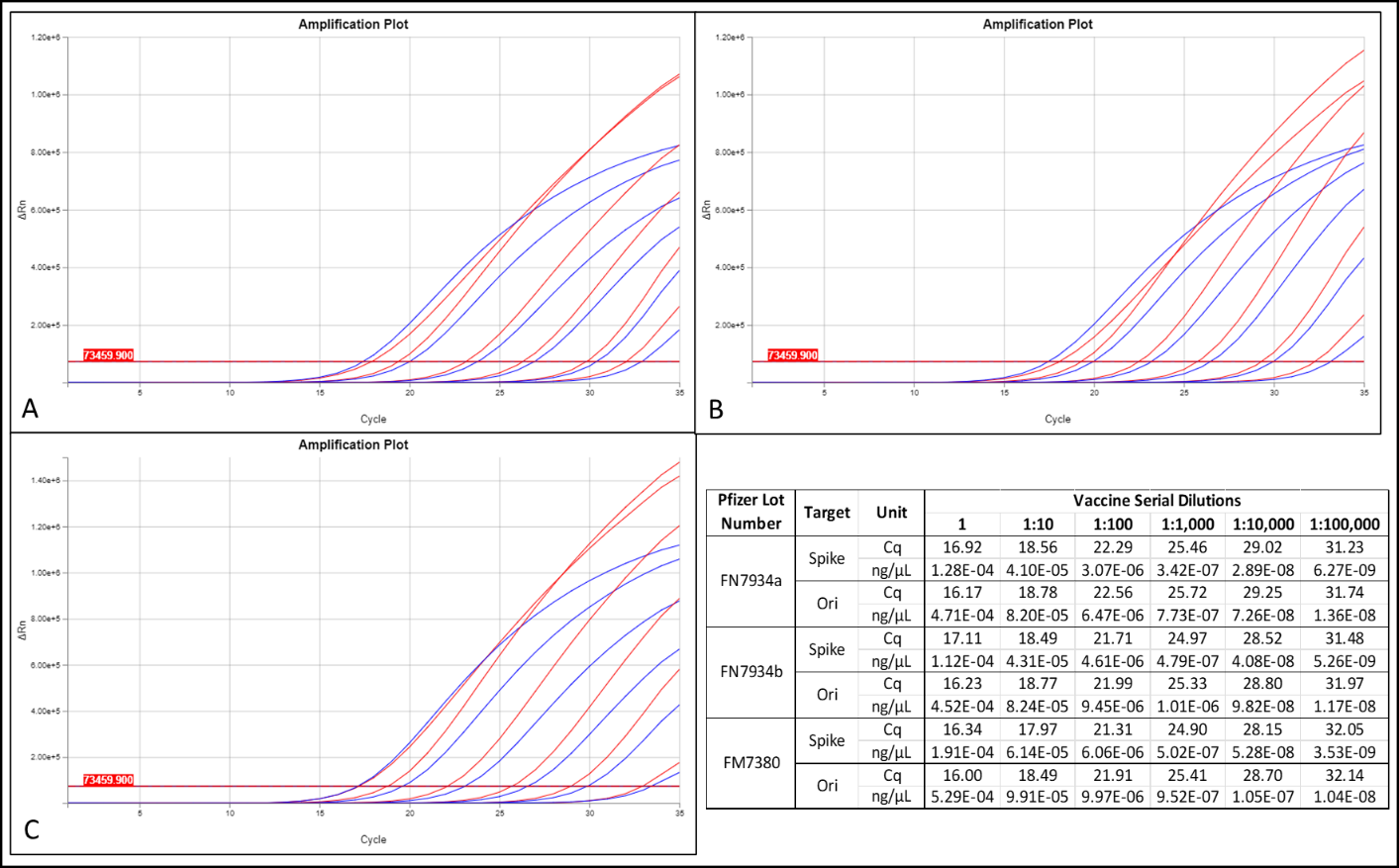
図6 DNA 負荷が最も高かった
3 ロット(ファイザー社ロット:A, FN7934a; B, FN7934b; C, FM7380)の連続希釈液(10 倍)から得られた。qPCR 増幅プロファイル。
残存DNA量は、qPCRで検査した場合、ロット間で大きく異なっていた(ファイザーオリ0.28~4.27ng/dose、ファイザースパイク0.22~2.43ng/dose、モデルナオリ0.01~0.34ng/dose、モデルナスパイク0.250.78ng/dose)。ワクチンの蛍光光度計を用いた測定(例:Qubit®)では、ファイザーで2,567±618 ng/dose(範囲:1,896~3,720 ng/dose)、モデルナで4,280±593 ng/dose(範囲:3,270~5,100 ng/dose)を示し、DNAの高い割合がqPCRアンプリコンのサイズ範囲内にあることが示唆された。
Qubitフルオロメトリーで得られた残存DNA値をqPCRで得られた値に対してプロットした(図7)。ファイザー社の製品では、オリ推定値とスパイク推定値の傾向線はいずれも正の傾きを示した。モデルナ製品のグラフはPfizer製品のグラフと異なり、どちらの軸でも値の重なりがほとんどなく、傾きがかなり浅くなっている。モデルナのプロットを詳細に見ると、ori値は負の傾きを示しているが、このトレンドラインは3つの外れた値の影響を受けている可能性がある。これらの値はモデルナ BA.1-Wuhan二価ワクチンのバイアルから得られた。
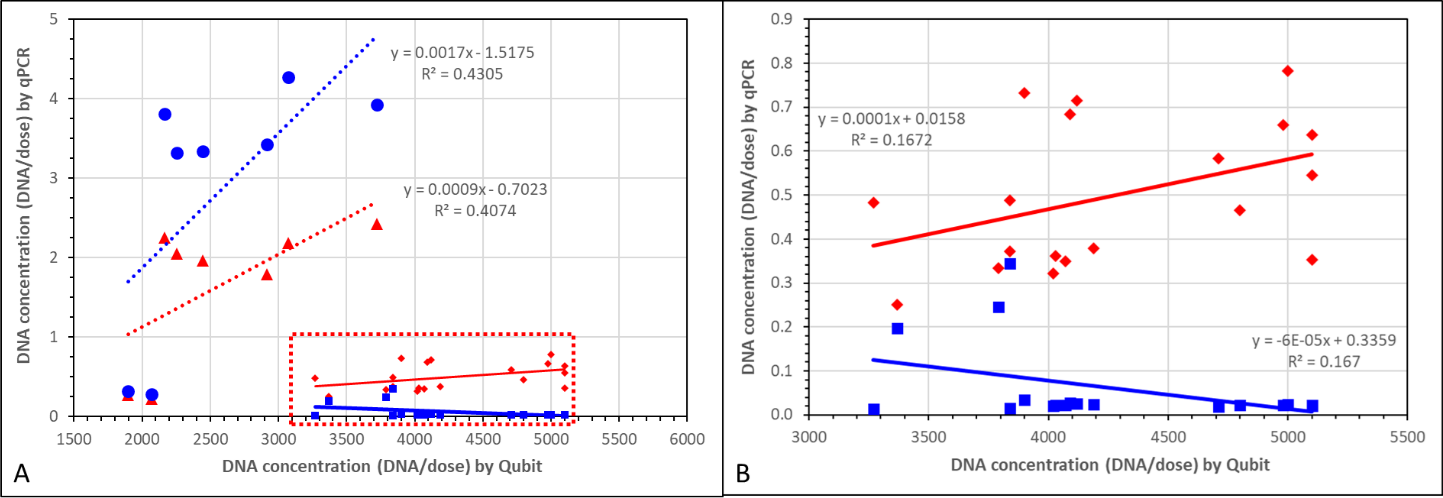
図7 qPCRで測定されたスパイク(赤)およびオリ(青)の残存DNA濃度と、Qubitで測定された個々のバイアル中の総残存DNA濃度のグラフ比較
パネルAでは、ファイザーとモデルナのデータが同じスケールでプロットされている。モデルナのデータは赤枠で囲み、パネルBでは詳細を表示するためにスケールを拡大して個別に表示している。
モデルナのロットAS0709D、AS0467D、020G23A以外では、本研究で調査したすべてのロットでVAERSレポートが見つかった(図5)。調査した12ロットのうち、世界のVAERSに報告された数が最も多かったロットはFM7380とFN7934で、それぞれ29件と42件であった。ロットFM7380の場合、15人(52%)がSAEを報告したが、ロットFN7934の場合、1人が死亡、2人が障害を報告、18人が入院を報告し、21人(50%)がSAEを報告した。ロット062H21A、020J21A、020E21A、GK0932,033M21Aおよび055K21Aについては、それぞれ9件、7件、5件、3件、2件および2件の報告があった。これらのロットのうち、モデルナのロット020J21Aでは5/7(71%)が入院を伴う報告であり、モデルナのロット020E21Aでは1/5が死亡を伴う報告であった。また、モデルナ 020E21A では、1/5の死亡例が報告された。VAERSに報告されたこれらのロットの AE は合計 100 件であり、このうち 48 件(48%)が、SAE であった。これらの AE(n=92)および SAE(n=44)のほとんどは米国外から報告され、その割合はほぼ同じであった。これらの 92 件の AEのうち、70 件(76%)がカナダに由来するものであり、さらに 5 件(5.4%)がカナダに由来するものであった。
探索的分析として、ファイザー(上段)およびモデルナ(下段)のバイアル瓶から検出されたスパイク(赤色)およびプラスミドオリ(青色)の DNA 量を SAE 報告比率(SRR)に対してプロットし、用量反応曲線を作成した(図 8)。ファイザー製品のoriおよびスパイク曲線は互いに類似しており、正の用量反応関係を示している。モデルナロットの対応する曲線は、1~2桁左にシフトしている。しかし、オリ曲線とスパイク曲線は位置と傾きが異なっている。
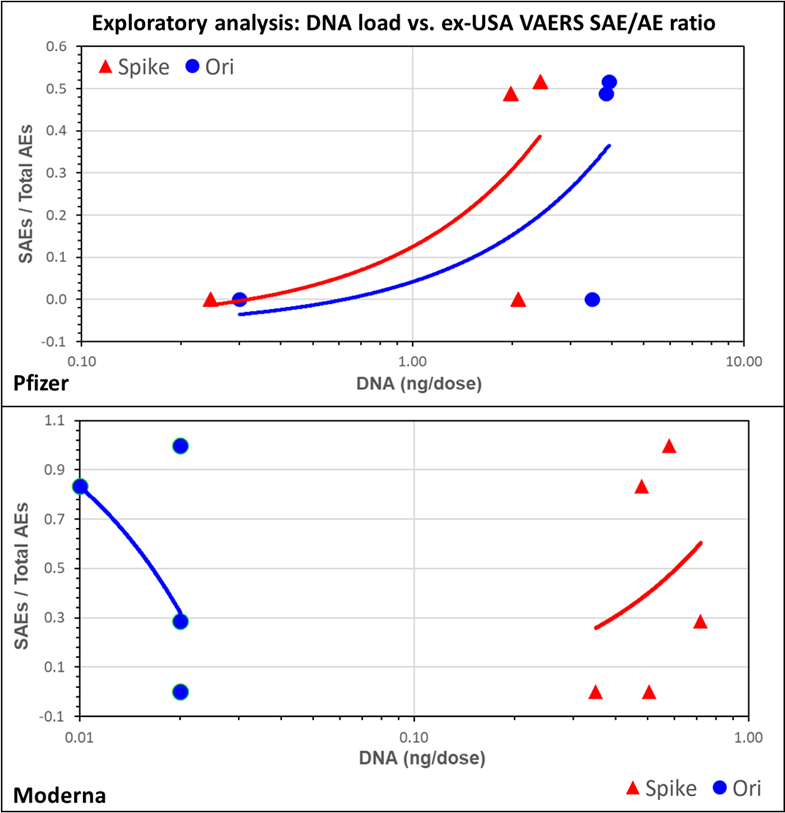
図8 ファイザー(A)ロットおよびモデルナ(B)ロットで検出されたスパイク(赤)およびプラスミドori(青)についてqPCRで測定された残留DNA濃度を、米国以外の国からの各ロットのSRR(SAEの報告数/VAERSに報告された全有害事象の総数)に対してプロットしたものを比較した探索的用量反応解析。投与量あたりの残存DNA量を対数スケールでプロットした。1:10 希釈のデータを使用した。
フルオロメトリーを用いて推定された残存 DNA 量に対応するプロット(図 9)では、ファイザーとモデルナの両製品で負の傾きを持つ曲線が得られた。
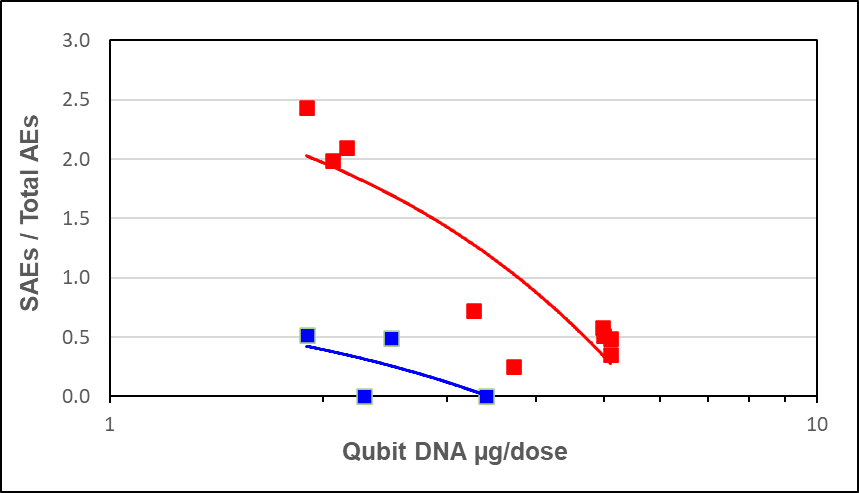
図9 ファイザー(青)とモデルナ(赤)のワクチンロットについて、Qubit蛍光光度法で測定した残存DNA濃度を、米国以外の国のSRR(SAEの報告数/VAERSに報告された全有害事象の総数)に対してプロットした、探索的用量反応解析。投与量あたりの残存DNA量を対数スケールでプロットした。
McKernanら4が記載したPfizer小児用一価(Lot FL8095)の塩基配列をOxford Nanopore(ONT)で決定し、リードをNCBIのプラスミドの参照配列にマッピングした後、リード長分布を評価した(図8)。865リードの中で検出された最長のリードは3.5 kbで、リードはプラスミドバックボーンのほとんどにマッピングされた(図9)。
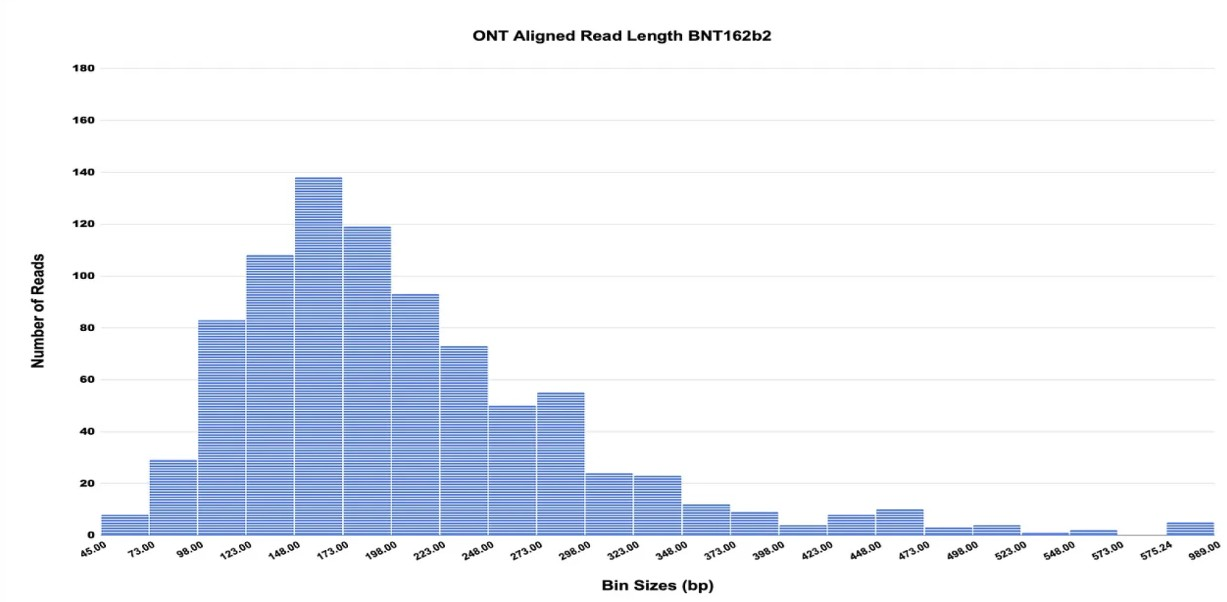
図10 ベクター配列(NCBI OR134577.1)にマッピングされた866リードのOxford Nanopore(ONT)リード長分布
平均214 bp。最大3.5 kb。
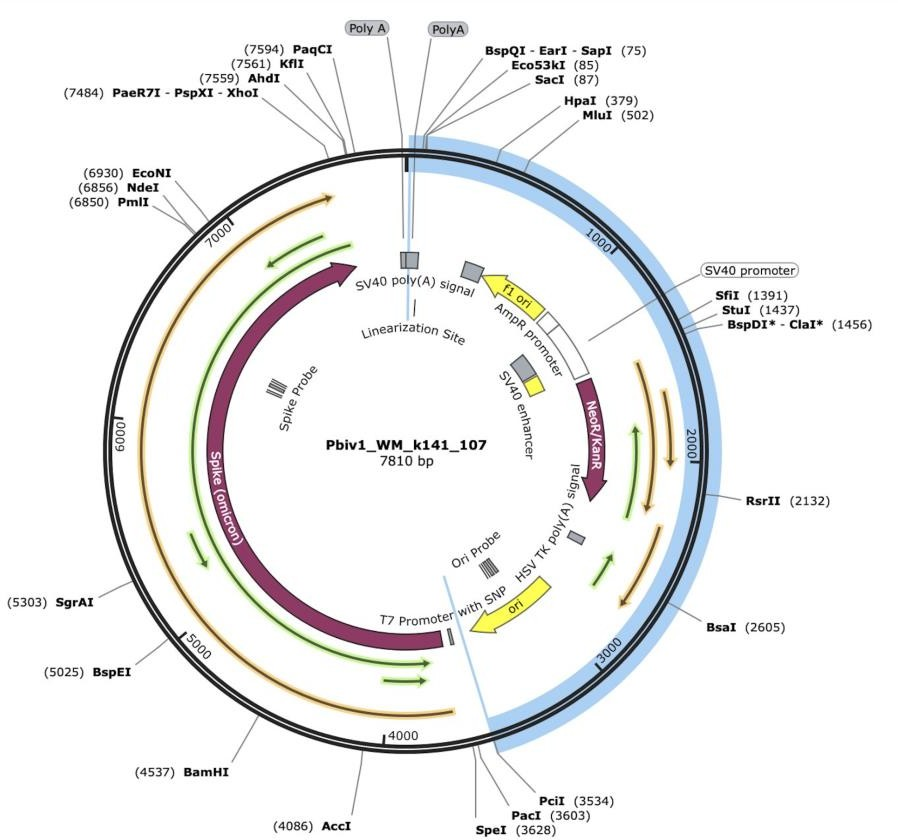
図11 最も長いOxford Nanopore (ONT)リードは、青色で示したベクター領域にアライメントしている
オープンリーディングフレーム(ORF)は金と緑の矢印で注釈されている。カナマイシン耐性遺伝子は、ワクチンのごく浅いシークエンス調査で検出された。
ファイザーワクチンのヌクレアーゼ感受性はDNaseI-XTを用いて評価した。このDNAヌクレアーゼはRNA:DNAハイブリッドが豊富なIVT反応に最適化されている。この処理では
<1Cqのオフセットを示した。一方、LNPにスパイクした裸のDNAコントロールは、同じ条件下で15Cqから検出不能まで減少した。このことは、ワクチンに含まれるDNAがLNPへのカプセル化によって保護されていることを示している(図10、図11)。
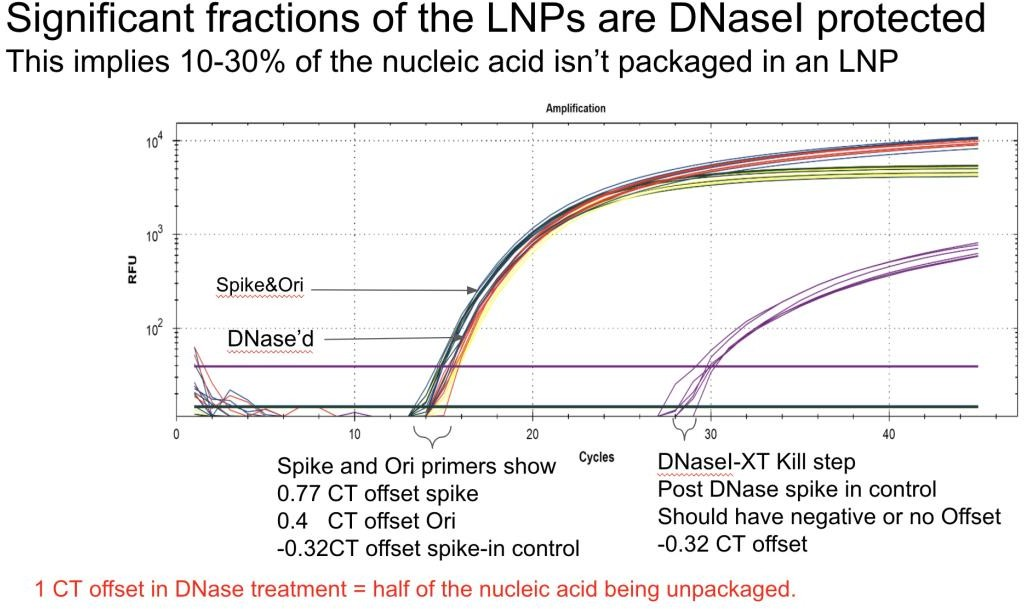
図12 ファイザー社製ワクチンのDNase I-XT処理により、ワクチン中のDNAのヌクレアーゼ耐性が証明された
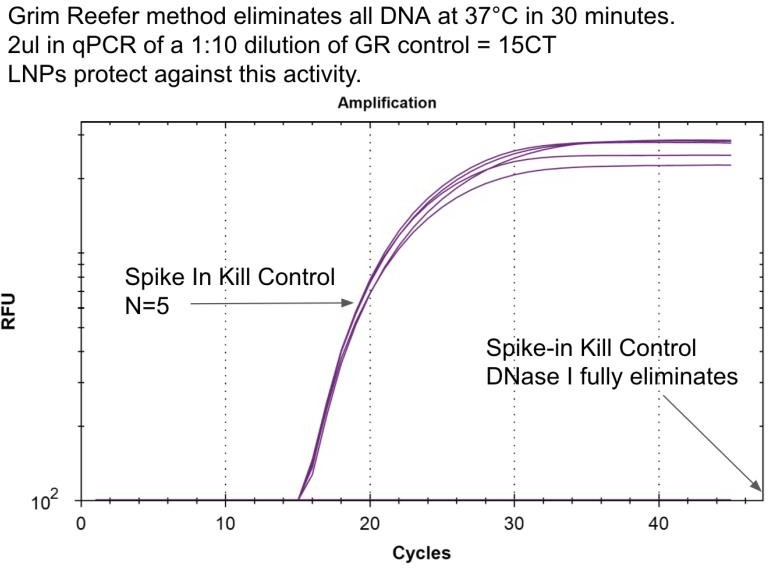
図13 DNaseI-XT陽性対照は、ワクチンのヌクレアーゼ感受性を評価するために使用されたのと同じ条件下で、消化アッセイによりスパイクされたDNAがすべて除去されることを示す
考察
残留DNAは調査した27本すべてのワクチンバイアルから検出された。同一ロットの複数のバイアルから、すべてのターゲットに対して非常に類似した負荷量が検出され、アッセイの信頼性、再現性、およびロット内での一貫性が示された。カナダで配布されたワクチンバイアルに関するこれらのデータは、modRNAワクチンにDNAが混入しているという非ピアレビューのいくつかの報告(McKernan、Buckhaults、Konig)と一致している4 15。
モデルナはqPCRで最もDNA濃度が低かったが、Qubitでは最も濃度が高かった。モデルナのバイアルは、バイアル間でDNA濃度が最も一定しており、より強固で標準化された製造工程であることが示唆された。ロットAS0467Dを除くモデルナ製品の各バイアルにおいて、oriはスパイクよりも低い濃度を示し、ベクターDNAがより効果的に除去されていることが示唆された。相同修飾RNAがハイブリダイゼーションによる鋳型DNAの消化を防いでいる可能性がある17。
DNA濃度が最も高いバイアルは、リン酸緩衝生理食塩水(PBS)製剤のファイザー社製一価紫色トップバイアルの2ロットであり、投与前に希釈が必要である。2021年10月29日、米国FDAは製剤をトリス/スクロース緩衝液に変更することを承認した。この変更は、安定性を高め、保管要件を簡素化し、すぐに使用できる製剤を提供するために行われた18 19。これらの紫色トップのファイザーのロットは、試験されたすべてのロットの中で、VAERSで報告されたAEおよびSAEの数が最も多かった。各ロットの実際の投与回数は不明であるため、毒性学的/薬理学的影響を推定するために、投与回数の代理として AEの総数を SAEの数の分母として使用した。これは、CDC10 11が不釣り合いシグナル分析(DSA)で用いているのと同じ原理である。
残留DNA含量とVAERSに報告されたSAEとの関係についての我々の探索的分析は、予備的なものであり、サンプル数も限られているが、より多くのロットおよびバイアルを調査することによって確認する必要がある。残留DNAのqPCR推定値に基づくと、ファイザーのロットで正の用量反応関係が観察された。
modernaのロットでは、ファイザーとmodernaの両方のロットで、qPCRデータおよびフルオロメトリーにより推定された残存DNAに基づくプロットにおいて、異なる関係が観察された。これらの観察結果は、DNAの量、DNA断片のサイズ分布、プラスミドベクターの組成と配列、脂質ナノ粒子の組成など、2つの製品間の相違を反映している可能性がある。2つの製品間および各製品の異なるロット間のその他の違いも、今回の観察結果に寄与している可能性がある。これらの違いには、汚染物質や不純物のレベルのばらつきが含まれる。不純物の主な原因のひとつは断片化したmRNAで、miRNAプロセスへの影響など、多くの毒性学的メカニズムが提唱されている20。dsRNAも不純物の一種で、T7 RNAポリメラーゼのプロモーターに二次的に発生する。dsRNAは炎症性サイトカイン21を誘導する可能性があり、心筋炎などの免疫炎症反応に関与しているという仮説がある22。エンドトキシンによる細胞内のリポ多糖は、スパイクプロテインのS1サブユニットとS2サブユニットの両方に結合し、炎症反応の亢進をもたらす可能性がある23。
より広範なサンプリングにより、死亡などの事象の種類や、様々なワクチンのロット番号24と AEの相関を報告した。Schmeling ら24のような他の研究結果との比較など、より詳細な情報が明らかになると思われる。現在研究されているワクチンのロットはSchmelingの研究には含まれておらず、このDNA汚染がAEとどのように関連しているかを理解するためには、さらなる研究が必要である。
SV40エンハンサーは核局在を促進するが、COVID-19 modRNA産物ではDNA断片のゲノムへの統合はまだ証明されていない26。しかし、DNAの混入は不当な自然免疫反応を引き起こす可能性があり、特にGC(グアニン・シトシン)含量の高い断片では血栓症を引き起こす可能性があることが知られている27。dsDNAはまた、脳卒中を含む虚血性疾患における重要な因子である可能性がある28。高濃度のDNA汚染とSAEとの間には相関関係があるように見えるが、サンプルサイズを拡大し、潜在的なメカニズムを解明するための研究が必要である。
qPCRではアンプリコンのサイズ(105-114bp)より小さい分子は定量できないため、qPCRでは各ワクチンに含まれる総DNA量が過小評価されることを強調しておきたい。このことは、Qubit蛍光法と比較してqPCRによって推定された残存DNA量が、特にファイザー社とモデルナ社の製品間で大きく異なることを説明する。モデルナ製品で得られた値が非常に大きいことから、Pfizer製品よりも断片化された小さな残存DNAの割合が高いことが示唆される。これは、より徹底したヌクレアーゼ消化ステップと一致する。このことは、FDAが推奨するDNA汚染ガイドラインが、DNAを定量するための方法に大きく依存していることを示している。高濃度のN1-methylpseudouridine modRNAを含むサンプルに使用する場合、DNAトロピック蛍光色素の特異性が不明であることが、高濃度の蛍光測定値を説明する別の仮説である。
EMAの文書3に記載されているように、ファイザー社のCOVID-19ワクチンに含まれるRNAの定量にはフルオロメトリーとUVスペクトロフォトメトリーが使用され、DNAの定量にはqPCRが使用されたため、このフルオロメトリーの評価は特に興味深い。RNA/DNA比を定量するために、このように異なる方法を選択的に使用することは、EMAで実施されている比測定ガイドラインに対して、大きく異なる結果をもたらす可能性がある。
qPCR定量に比べてフルオロメトリー定量が高いことは、DNAの一部がアンプリコンサイズより小さい可能性を示唆するONTリード長分布とも一致する。ONTシークエンサーは100bpより短い分子を検出するが、ONTのライブラリー構築法は、0.7X Ampure DNA精製ステップを使用する。その結果、ONTのリード長分布は150 bp以上の断片に偏り、存在する可能性のある小さな断片を完全に反映したものではなく、ONTでもqPCRでもカウントが不十分となる。
現在、米国FDAはウイルスワクチン製造業者に対し、非経口接種の場合、最終製品中の残留DNA量を 10ng/dose以下に、DNAのサイズを機能的遺伝子サイズ、すなわち200塩基対以下に制限することを推奨している13。1986年のWHOの研究グループは、100pg/線量まではリスクは無視できると結論づけ、1996年にWHOはさらに10ng/線量までレベルを引き上げた14。
FDAとWHOのワクチン中のDNA許容量ガイドラインは、FDAの科学者であるSheng-Fowlerら30が発表した研究の影響を受けている。このようなナノグラムあたりのコピー数が多い汚染物質では、フェムトグラムからアトグラムのDNAが、ナノグラムの細胞基質ゲノムDNAに相当すると考えられる。modRNAワクチンの断片サイズが短いことから、1回の投与量に含まれる分子数は1,000億分子以上に達する可能性がある。これらのワクチンに残存するDNAはコピー数が多く、プロモーター、ORF、核標的配列が豊富である。FDAとWHOのガイドラインは、脂質ナノ粒子にDNAを封入することを考慮しておらず、その結果、DNAの残存期間が長くなり、トランスフェクション効率が高まる可能性が高い。さらに、ガイドラインはLNPベースのmodRNAの累積投与を考慮していない。COVID-19ワクチンを5回以上投与し、ブースター投与の間隔を2カ月と短くした例もある。さらに、同じプラスミドとLNPベースのmodRNAプラットフォームを使用しているが、他の感染症を対象としたワクチンによる累積投与のリスクは、残存DNAガイドラインの設定において考慮されていない。
FDAのガイドラインはまた、200bp以上のDNA断片のみを定量するように書かれているが、その理由の一つは、これより小さな断片は機能的遺伝子を産生することができないと考えられていたからである。しかし、Klinmanら31は、7bpという小さな断片が統合のリスクをもたらす可能性があることを示唆している。さらに、このガイドラインは、200bpより短い裸のDNA断片は、より大きな分子よりも宿主のヌクレアーゼ活性によって急速に加水分解されることも考慮に入れている可能性がある32。
Klinmanら31はまた、「プラスミド統合の潜在的な害を評価する際には、強力な制御領域を持つプラスミドを宿主ゲノムに導入するリスクは、ランダムな点突然変異に関連するリスクをはるかに上回ることに注意すべきである」と述べている。
Vacikらは、ファイザー社のベクターに存在するSV40エンハンサーが、遺伝子治療に有望な強力な核標的配列であることを示した25。
結論
これらのデータは、試験したmodRNA COVID-19製品中に、1用量当たり数十億から数千億のDNA分子が存在することを示している。フルオロメトリーを用いると、試験したすべての製品はFDAとWHOが設定した残留DNAのガイドラインである10ng/用量を188-509倍超えていた。しかし、qPCRで検出された残留DNA量は、試験した全製品でこのガイドラインを下回っており、定量的ガイドラインを解釈する際には、方法論の明確さと一貫性が重要であることが強調された。最新のXBB.1.5 モデルナワクチンのCqスコアは、DNA残存量が以前のワクチンバージョンから減少していないことを示唆している。
qPCRで測定された残留DNAとSAEの用量反応効果に関する予備的証拠は、確認とさらなる調査が必要である。我々の知見は、ワクチンの安全性に関する既存の懸念を拡大し、LNPを用いた効率的なトランスフェクションが導入される前に考案されたガイドラインの妥当性に疑問を投げかけるものである。いくつかの明らかな限界があるが、われわれの研究を法医学的条件下で再現し、高効率DNAトランスフェクションと累積投与量を考慮したガイドラインの改訂を強く要望する。
この研究は、規制当局と産業界が予防原則を遵守し、製品が安全で有効であるという十分かつ透明な証拠を提供し、その組成と製造方法の詳細を開示する必要性を強調している。
データの入手
マッピングされたONTシーケンスデータのFastqファイル: mega.nz/file/UZhkiTBQ#8 vjDK5JV5N5Dj2On34B6zdRObEKGBy3ZC7w8q2t9U Vc
謝辞
我々の取り組みに寄付してくださったすべての薬剤師に感謝する。
著者貢献
DJ Speicher:サンプル管理、試験デザイン、qPCR、データ解析、原稿作成
J Rose: J・Rose:VAERS解析、原稿作成 LM・Gutschi:データ解析、原稿作成 D・Wiseman:データ解析、原稿作成
K McKernan:qPCRアッセイデザイン、DNAseIおよびONT実験、原稿作成
利益相反声明
Kevin McKernanはMedicinal Genomics社に雇用されており、qPCR試薬を無償で提供した。他の著者は利益相反がないことを宣言している。
改訂履歴
2023-10-19 – バージョン1.0
文献
-
Guetzkow JA. Letters to the editor: Effect of mRNA Vaccine Manufacturing Processes on Efficacy and Safety Still an Open Question. BMJ 2023;378:o1731.
-
Tinari S. The EMA covid-19 data leak, and what it tells us about mRNA instability.
BMJ 2021;372:n627. doi: 10.1136/bmj.n627 [published Online First: 20210310]
-
EMEA/H/C/005735/RR. Rapporteur Rolling Review Critial Assessment Report: Quality Aspects. 2020 [Available from: covidvaccinereactions.com/ema- pfizer-leak/.
-
McKernan K, Helbert Y, Kane LT, et al. Sequencing of bivalent Moderna and Pfizer mRNA vaccines reveals nanogram to microgram quantities of expression vector dsDNA per dose. OSFio 2023 doi: 10.31219/osf.io/b9t7m
-
Shimabukuro TT, Nguyen M, Martin D, et al. Safety monitoring in the Vaccine Adverse Event Reporting System (VAERS). Vaccine 2015;33(36):4398-405. doi: 10.1016/j.vaccine.2015.07.035 [published Online First: 20150722]
-
Dean DA, Dean BS, Muller S, et al. Sequence requirements for plasmid nuclear import. Exp Cell Res 1999;253(2):713-22. doi: 10.1006/excr.1999.4716
-
New England Biolabs. NEBioCalculator 2023 [Available from: nebiocalculator.neb.com/#!/dsdnaamt2023.
-
R: The R Project for Statistical Computing [program]: R Foundation for Statistical Computing, 2010.
-
Funk PR, Yogurtcu ON, Forshee RA, et al. Benefit-risk assessment of COVID-19 vaccine, mRNA (Comirnaty) for age 16-29 years. Vaccine 2022;40(19):2781-89. doi: 10.1016/j.vaccine.2022.03.030 [published Online First: 20220328]
-
VAERS Team. Vaccine Adverse Event Reporting System (VAERS) Standard Operating Procedures for COVID-19 (as of 29 January 2021) 2021 [Available from: www.cdc.gov/vaccinesafety/pdf/VAERS-v2-SOP.pdf.
-
Evans SJ, Waller PC, Davis S. Use of proportional reporting ratios (PRRs) for signal generation from spontaneous adverse drug reaction reports. Pharmacoepidemiol Drug Saf 2001;10(6):483-6. doi: 10.1002/pds.677
-
Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform. Bioinformatics 2009;25(14):1754-60. doi: 10.1093/bioinformatics/btp324 [published Online First: 20090518]
-
Food and Drug Administration. Chemistry, Manufacturing, and Control (CMC) Information for Human Gene Therapy Investigational New Drug Applications (INDs): U.S. Department of Health and Human Services; 2020 [Available from: www.fda.gov/media/113760/download.
-
Shin J, Wood D, Robertson J, et al. WHO informal consultation on the application of molecular methods to assure the quality, safety and efficacy of vaccines, Geneva, Switzerland, 7-8 April 2005. Biologicals 2007;35(1):63-71. doi: 10.1016/j.biologicals.2005.12.005 [published Online First: 20060220]
-
South Carolina Senate. SC Senate Hearing – USC Professor Dr. Phillip Buckhaults 2023 2023/10/19
-
Nelson J, Sorensen EW, Mintri S, et al. Impact of mRNA chemistry and manufacturing process on innate immune activation. Sci Adv 2020;6(26):eaaz6893. doi: 10.1126/sciadv.aaz6893 [published Online First: 20200624]
-
Milano G, Gal J, Creisson A, et al. Myocarditis and COVID-19 mRNA vaccines: a mechanistic hypothesis involving dsRNA. Future Virol 2021 doi: 10.2217/fvl-2021- 0280 [published Online First: 20211206]
-
Samsudin F, Raghuvamsi P, Petruk G, et al. SARS-CoV-2 spike protein as a bacterial lipopolysaccharide delivery system in an overzealous inflammatory cascade. J Mol Cell Biol 2023;14(9) doi: 10.1093/jmcb/mjac058
-
Schmeling M, Manniche V, Hansen PR. Batch-dependent safety of the BNT162b2 mRNA COVID-19 vaccine. Eur J Clin Invest 2023;53(8):e13998. doi: 10.1111/eci.13998 [published Online First: 20230409]
-
Vacik J, Dean BS, Zimmer WE, et al. Cell-specific nuclear import of plasmid DNA.
Gene Ther 1999;6(6):1006-14. doi: 10.1038/sj.gt.3300924
-
Lim S, Yocum RR, Silver PA, et al. High spontaneous integration rates of end- modified linear DNAs upon mammalian cell transfection. Sci Rep 2023;13(1):6835. doi: 10.1038/s41598-023-33862-0 [published Online First: 20230426]
-
Gaitzsch E, Czermak T, Ribeiro A, et al. Double-stranded DNA induces a prothrombotic phenotype in the vascular endothelium. Sci Rep 2017;7(1):1112. doi: 10.1038/s41598-017-01148-x [published Online First: 20170425]
-
Ma X, Xin D, She R, et al. Novel insight into cGAS-STING pathway in ischemic stroke: from pre- to post-disease. Frontiers in Immunology 2023;14
-
World Health Organization. Annex 3: Requirements for the use of animal cell cultures as substrates for the manufacture of biologic medicinal products and the characterization of cell banks. Replacement of Annex 1 of WHO Technical Report Series, No. 878. 2013 [Available from: cdn.who.int/media/docs/default- source/biologicals/documents/trs_978_annex_3.pdf?sfvrsn=fe61af77_3&downloa d=true.
-
Sheng-Fowler L, Lewis AM, Jr., Peden K. Issues associated with residual cell- substrate DNA in viral vaccines. Biologicals 2009;37(3):190-5. doi: 10.1016/j.biologicals.2009.02.015 [published Online First: 20090314]
-
Klinman DM, Klaschik S, Tross D, et al. FDA guidance on prophylactic DNA vaccines: analysis and recommendations. Vaccine 2010;28(16):2801-5. doi: 10.1016/j.vaccine.2009.11.025 [published Online First: 20091124]
-
André M, Reghin S, Boussard E, et al. Universal real-time PCR assay for quantitation and size evaluation of residual cell DNA in human viral vaccines. Biologicals 2016;44(3):139-49. doi: 10.1016/j.biologicals.2016.03.002 [published Online First: 20160328]
