Contents
SARS–CoV–2 Spike Impairs DNA Damage Repair and Inhibits V(D)J Recombination In Vitro
Hui Jiang 1,2,* andYa-Fang Mei 2,* 著
1Department of Molecular Biosciences, The Wenner-Gren Institute, Stockholm University, SE-10691 Stockholm, Sweden
2Department of Clinical Microbiology, Virology, Umeå University, SE-90185 Umeå, Sweden
*
受け取った。2021年8月20日 / 改訂:2021年9月8日 / 受理:2021年10月13日 2021年10月8日 / 公開日:2021年10月13日
概要
重症急性呼吸器症候群新型コロナウイルス(SARS-CoV-2)は、コロナウイルス感染症2019(COVID-19)のパンデミックを引き起こし、公衆衛生と世界経済に深刻な影響を与えている。適応免疫は、SARS-CoV-2感染症との戦いにおいて重要な役割を果たし、患者の臨床転帰に直接影響を与える。しかし、SARS-CoV-2が適応免疫を阻害するメカニズムはまだ明らかになっていない。今回、我々は、SARS-CoV-2のスパイクタンパク質が、適応免疫におけるV(D)J組み換えの有効性に必要なDNA損傷修復を著しく阻害することを、試験管内試験の細胞株を用いて報告した。そのメカニズムは、スパイクタンパク質が核内に局在し、DNA修復の鍵となるタンパク質であるBRCA1と53BP1の損傷部位へのリクルートを阻害することで、DNA損傷修復を阻害することを明らかにした。今回の発見は、スパイクタンパク質が適応免疫を阻害する潜在的な分子メカニズムを明らかにするとともに、全長スパイクワクチンの潜在的な副作用を強調するものである。
キーワード SARS-CoV-2; スパイク; DNA損傷修復; V(D)J組換え; ワクチン
1. はじめに
重症急性呼吸器症候群新型コロナウイルス(SARS-CoV-2)は、230万人以上の死亡者を出した現在進行中のコロナウイルス感染症2019(COVID-19)のパンデミックの原因となっている。SARS-CoV-2は、構造タンパク質と非構造タンパク質からなるエンベロープ型シングルポジティブセンスRNAウイルスである[1]。感染後,これらのウイルスタンパク質は,宿主の細胞機構をハイジャックして制御不能にし,子孫ウイルスを複製,組み立て,拡散させる [2].最近の臨床研究では、SARS-CoV-2感染がリンパ球の数と機能に特別な影響を与えることが示されている[3,4,5,6]。軽度および中等度の生存者と比較して、重度のCOVID-19患者は、総T細胞数、ヘルパーT細胞数、サプレッサーT細胞数が有意に少ないことが明らかになっている[3,4]。さらに、COVID-19では、症状が出てからIgGおよびIgMのレベルが遅れている[5,6]。これらの臨床観察結果を総合すると、SARS-CoV-2が適応免疫系に影響を与えていることが示唆される。しかし、SARS-CoV-2が適応免疫を抑制するメカニズムはまだ不明である。
免疫系とDNA修復系は、高等生物が多様な脅威からの防御や組織の恒常性維持のために利用する重要なモニタリングシステムである。この2つのシステムは、特にリンパ球の発生と成熟の過程において、相互に依存していることが明らかになっている[7]。主要な二本鎖DNA切断(DSB)修復経路の1つである非相同末端結合(NHEJ)修復は、リンパ球特異的な組換え活性化遺伝子エンドヌクレアーゼ(RAG)を介したV(D)J組換えにおいて重要な役割を果たしており、その結果、B細胞では抗体、T細胞ではT細胞受容体(TCR)のレパートリーが非常に多様化している[8]。例えば、ATM、DNA-PKcs、53BP1などの主要なDNA修復タンパク質の機能が失われると、NHEJ修復に不具合が生じ、機能的なB細胞やT細胞の産生が阻害され、免疫不全に陥る[7,9,10,11]。一方、ウイルス感染は、通常、活性酸素種(ROS)の産生や宿主細胞の複製ストレスの誘導など、さまざまなメカニズムでDNA損傷を引き起こす[12,13,14]。DNA損傷が適切に修復されなければ、ウイルス感染によって誘発される病態の増幅に寄与することになる。そこで我々は、SARS-CoV-2タンパク質がDNA損傷修復システムをハイジャックし、それによって適応免疫に影響を与えるかどうかを試験管内試験で調べることを目的とした。
2. 材料と方法
2.1. 抗体および試薬
DAPI (Cat #MBD0015), doxorubicin (Cat #D1515), H2O2 (Cat #H1009), β-tubulin抗体 (Cat #T4026)はSigma-Aldrich社から購入した。Hisタグ(Cat #12698)、H2A(Cat #12349)、H2A.X(Cat #7631)、γ-H2A.X(Cat #2577)、Ku80(Cat #2753)、Rad51(Cat #8875)に対する抗体は、Cell Signaling Technology (Danvers, MA, USA)から購入した。53BP1(Cat #NB100-304)およびRNF168(Cat #H00165918-M01)抗体は、Novus Biologicals(Novus Biologicals, Littleton, CO, USA)から入手した。Lamin B(Cat #sc-374015)ATM(Cat #sc-135663)DNA-PK(Cat #sc-5282)およびBRCA1(Cat #sc-28383)抗体は、Santa Cruz Biotechnology(Santa Cruz, CA, USA)から購入した。XRCC4(Cat #PA5-82264)抗体はThermo Fisher Scientific(Waltham, MA, USA)から購入した。
2.2. プラスミド
pHPRT-DRGFPおよびpCBASceIは,Maria Jasinから寄贈された(Addgene plasmid #26476および#26477)[15]。pimEJ5GFPはJeremy Starkから寄贈された(Addgene plasmid #44026)[16]。NSP1, NSP9, NSP13, NSP14, NSP16, スパイクおよびヌクレオカプシドタンパク質は,まずコドン最適化を行って合成し,C末端に6xHisタグを付けて哺乳類発現ベクターpUC57にクローニングした。また,V(D)Jレポーターベクターには,12スペーサーのRSS-GFP逆相補鎖配列-23スペーサーのRSSを合成した。この配列をpBabe-IRES-MRFPベクターにクローニングして,pBabe-12RSS-GFPi-23RSS-IRES-MRFPレポーターベクターを作製した。12-スペーサーRSS配列。5′-cacagtgctacagactggaacaaaaacc-3′.23-スペーサーRSS配列.5′-cacagtggtagtactccactgtctggctgtacaaaaacc-3′.RAG1およびRAG2の発現コンストラクトは、Martin Gellert氏から惜しみなく提供された(Addgene plasmid #13328および#13329)[17]。
2.3. 細胞および細胞培養
ATCC(American Type Culture Collection)から入手したHEK293T細胞およびHEK293細胞を、10%(v/v)ウシ胎児血清(FCS、Gibco)1%(v/v)ペニシリン(100IU/mL)およびストレプトマイシン(100μg/mL)を含むダルベッコ改変イーグル培地(DMEM、高グルコース、GlutaMAX)(Life Technologies、Carlsbad、CA、USA)中、37℃で5%CO2下で培養した。HEK293T-DR-GFPおよびHEK293T-EJ5-GFPレポーター細胞は,前述の方法で作製し,5%CO2,37℃の環境下で,上記の培養液を用いて培養した。
2.4. HRおよびNHEJレポーターアッセイ
HEK293T細胞におけるHRおよびNHEJ修復は、DR-GFPおよびEJ5-GFP安定細胞を用いて前述のように測定した。0.5×106個のHEK293T安定レポーター細胞を6ウェルプレートに播き、2μgのI-SceI発現プラスミド(pCBASceI)とSARS-CoV-2タンパク質発現プラスミドを一緒にトランスフェクトした。トランスフェクションおよびアスピリン処理の48時間後に細胞を採取し、フローサイトメトリー分析によりGFPの発現を解析した。平均値は、3回の独立した実験から得られたものである。
2.5. 細胞分画およびイムノブロッティング
細胞分画アッセイには、メーカーの説明書に従ってSubcellular Protein Fractionation Kit(Thermo Fisher社)を使用した。BCA試薬(Thermo Fisher Scientific, Rockford, IL, USA)を用いてタンパク質ライセートを定量した。ドデシル硫酸ナトリウム-ポリアクリルアミドゲル電気泳動(SDS-PAGE)でタンパク質を分解し,ニトロセルロース膜(Amersham protran, 0.45μm NC)に転写し,特異的な一次抗体とHRP標識二次抗体を用いてイムノブロットした。タンパク質のバンドは,SuperSignal West Pico or Femto Chemiluminescence kit (Thermo Fisher Scientific)を用いて検出した。
2.6. コメットアッセイ
細胞を異なるDNA損傷試薬で処理した後、指示した時点で採取して分析した。細胞(1×105 cells/mL、冷えたリン酸緩衝生理食塩水[PBS])を1:3 vol/volの割合で40℃の1%低融点アガロースに再懸濁し、コメットスライドにピペッティングした。その後、あらかじめ冷やしておいた溶解バッファー(1.2 M NaCl, 100 mM EDTA, 0.1% Sodium lauryl sarcosinate, 0.26 M NaOH pH > 13)に浸し、4℃の暗所で一晩(18-20時間)溶解させた。その後、スライドを注意深く取り出し、リンスバッファー(0.03M NaOHおよび2mM EDTA、pH > 12)に室温(RT)で20分間、暗所で浸した。この洗浄ステップを2回繰り返した。スライドをリンスバッファーを含む水平電気泳動室に移し,0.6V/cmの電圧で25分間分離した。最後に、スライドを蒸留水で洗浄し、10μg/mLのヨウ化プロピジウムで染色し、蛍光顕微鏡で分析した。各サンプルの約100個の細胞を含む20フィールドを評価し、フィジーソフトウェアを用いて定量化し、尾の長さ(テールモーメント)を決定した。
2.7. 免疫蛍光法(Immunofluorescence
12ウェルプレートのガラスカバースリップ上に細胞を播種し、指示したプラスミドで24時間トランスフェクションした後、実験設定に従ってDNA損傷試薬を用いて、または用いずに細胞を処理した。細胞をPBSに溶解した4%パラホルムアルデヒド(PFA)で20分間固定した後,0.5% Triton X-100で10分間伝染させた。スライドを5%正常ヤギ血清(NGS)でブロッキングし、1%NGSで希釈した一次抗体と4℃で一晩インキュベートした。その後、1% NGSで希釈したAlexa Fluor 488または555(Invitrogen社)で標識した指示の二次抗体とRTで1時間インキュベートし、その後、DAPIでRTで15分間染色した。カバースリップをDako Fluorescence Mounting Medium(Agilent)を用いてマウントし,Nikon共焦点顕微鏡(Eclipse C1 Plus)を用いて画像化した。すべてのスコアリングは、ブラインド条件下で行った。
2.8. V(D)Jリコンビネーションの解析
V(D)Jレポータープラスミドには,逆GFPと,連続的に発現するRFPを駆動するIRESが含まれている。継続的に発現するRFPは内部トランスフェクションのコントロールとなる。組換え活性化遺伝子1/2(RAG1/2)を細胞に共導入すると、RAG1/2がRSSを切断してDSBの誘導を仲介し、V(D)J組換えが起こると、逆GFPはNHEJ修復により正順にライゲーションされる。そして、細胞は機能的なGFPを発現することになる。つまり、GFPとRFPの二重陽性細胞がV(D)Jレポーターアッセイのリードアウトとなるわけだ[18]。70%コンフルエントになった293T細胞に,V(D)J GFPレポーターを単独で(バックグラウンド),あるいはRAG1およびRAG2発現コンストラクトと組み合わせて,1 µg V(D)J GFPレポーター:0.5 µg RAG1:0.5 µg RAG2の割合でトランスフェクトした。翌日、培地を交換し、さらに48時間後に細胞を採取し、フローサイトメトリーでGFPおよびRFPの発現を解析した。
2.9. 統計解析
すべての実験は、独立して収集または準備したサンプルを用いて、少なくとも3回繰り返した。データは、GraphPad 8を用いて、Studentのt検定またはANOVAにTukeyの多重比較検定を加えて解析した。
3. 結果
3.1. 核内に局在するSARS-CoV-2ウイルスタンパク質のDNA損傷修復への影響
DNA損傷修復は、ゲノムの安定性を確保するために主に核内で行われる。SARS-CoV-2のタンパク質は細胞質で合成されるが[1]、Nsp1, Nsp5, Nsp9, Nsp13, Nsp14, Nsp16など、一部のウイルスタンパク質は核内でも検出される[19]。我々は,これらの核内に局在するSARS-CoV-2タンパク質が,宿主細胞のDNA損傷修復システムに影響を与えるかどうかを調べた。そこで,これらのウイルスタンパク質の発現プラスミドと,一般的に細胞質に局在するタンパク質と考えられているスパイクや核タンパク質の発現プラスミドを一緒に構築した。これらのタンパク質の発現と局在を、免疫ブロット法と免疫蛍光法によって確認した(図1Aと図S1A)。
Nsp1,Nsp5,Nsp9,Nsp13,Nsp14,Nsp16は確かに核に局在しており,核タンパク質は主に細胞質に局在していることが判明した。意外なことに、スパイクタンパク質は核に豊富に存在することがわかった(図1A)。NHEJ修復と相同組換え(HR)修復は、2つの主要なDNA修復経路であり、ゲノムの完全性を継続的にモニタリング・確保するだけでなく、適応免疫細胞の機能にも不可欠である[9]。これらのウイルスタンパク質がDSB修復経路を阻害しているかどうかを評価するために,HRとNHEJのレポーターシステムとして,direct repeat-green fluorescence protein(DR-GFP)とtotal-NHEJ-GFP(EJ5-GFP)をそれぞれ用いて,I-SceIエンドヌクレアーゼによって誘導された部位特異的DSBの修復を調べた[15,16].
Nsp1,Nsp5,Nsp13,Nsp14,スパイクタンパク質を過剰発現させると、HRとNHEJの両方の修復の効率が低下した(図1B-E、図S2A,B)。さらに、Nsp1,Nsp5,Nsp13,Nsp14を過剰発現させると、他のタンパク質に比べて増殖が劇的に抑制されることもわかった(図S3A,B)。したがって、Nsp1,Nsp5,Nsp13,Nsp14によるDNA損傷修復の抑制効果は、成長停止や細胞死などの二次的な作用によるものと考えられる。興味深いことに、スパイクタンパク質を過剰発現させても、細胞の形態や増殖には影響を与えないが、HR修復とNHEJ修復の両方を有意に抑制した(図1B-E、図S2A,BおよびS3A,B)。
図1 重症急性呼吸器症候群新型コロナウイルス(SARS-CoV-2)の核内に局在するタンパク質がDNA損傷修復に及ぼす影響
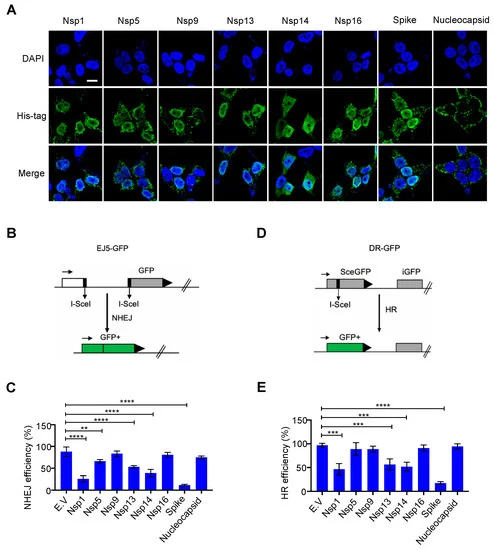
(A)SARS-CoV-2タンパク質の細胞内分布。ウイルスタンパク質を発現させたプラスミドをHEK293T細胞にトランスフェクションしてから24時間後に免疫蛍光法を行った。スケールバー。10μm。(B)非相同末端結合(NHEJ)のモニタリングに用いたEJ5-GFPレポーターの模式図。(C)空のベクター(E.V.)およびSARS-CoV-2タンパク質がNHEJ DNA修復に及ぼす影響。値は、3回の独立した実験の平均±標準偏差(SD)を表す(図S2Aの代表的なFACSプロットを参照)。(D)相同組換え(HR)のモニタリングに用いたDR-GFPレポーターの模式図。(E)HRのDNA修復に対するE.VおよびSARS-CoV-2タンパク質の効果。数値は、3回の独立した実験の平均±SDを表す(図S2Bの代表的なFACSプロットを参照)。統計的有意性は、(C,E)の一元配置分散分析(ANOVA)を用いて判断した。** p < 0.01, *** p < 0.001, **** p < 0.0001. 3.2. SARS-CoV-2のスパイクタンパク質はDNA損傷修復を阻害する スパイクタンパク質は、ウイルスが宿主細胞に侵入する際に重要な役割を果たしており、ほとんどのワクチン戦略の焦点となっている[20,21]。そこで、DNA損傷修復とそれに伴うV(D)J組み換えにおけるスパイクタンパク質の役割をさらに調べた。スパイクタンパク質は、通常、粗面小胞体(ER)上で合成されると考えられている[1]。スパイクタンパク質は、グリコシル化などの翻訳後修飾を受けた後、他のウイルスタンパク質とともに細胞膜装置を介して輸送され、成熟したビリオンを形成する[1]。スパイクタンパク質には,S1とS2という2つの主要なサブユニットのほか,いくつかの機能ドメインやリピートが存在する[22](図2A)。スパイクタンパク質は,本来,不活性な完全長のタンパク質として存在する.ウイルス感染時には,宿主細胞のプロテアーゼであるフーリンプロテアーゼがSタンパク質をS1とS2のサブユニットに切断して活性化し,これがウイルスの標的細胞への侵入に必要となる[23]。さらに,DNA修復阻害に必要な機能的特徴を明らかにするため,スパイクタンパク質のさまざまなサブユニットを調べた。その結果、完全長のスパイクタンパク質だけが、NHEJとHRの両方の修復を強く阻害した(図2B-Eおよび図S4A,B)。次に、スパイクタンパク質がDSB修復を阻害することでゲノムの不安定性に直接寄与しているかどうかを調べた。コメットアッセイを用いてDSBのレベルをモニターした。γ線照射、ドキソルビシン処理、H2O2処理など、さまざまなDNA損傷処理を行ったところ、スパイクタンパク質の存在下では修復が少なくなった(図2F,G)。これらのデータは、スパイクタンパク質が核内のDNA修復に直接影響を与えることを示している。
図2 重症急性呼吸器症候群新型コロナウイルス(SARS-CoV-2)のスパイクタンパク質はDNA損傷修復を阻害する
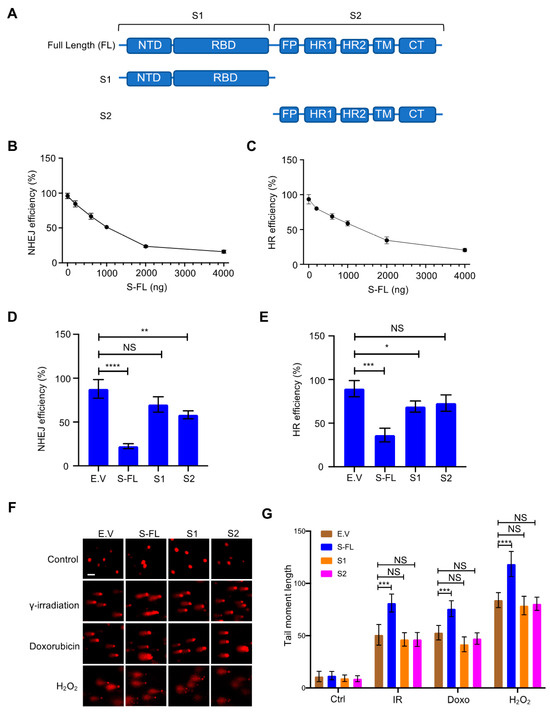
(A)SARS-CoV-2のスパイクタンパク質の一次構造の模式図。S1サブユニットは、N末端ドメイン(NTD、14-305残基)と受容体結合ドメイン(RBD、319-541残基)を含む。S2サブユニットは、融合ペプチド(FP,788-806残基)ヘプタペプチド反復配列1(HR1,912-984残基)HR2(1163-1213残基)TMドメイン(TM,1213-1237残基)細胞質ドメイン(CT,1237-1273残基)から構成されている。(B,C) HEK-293T細胞におけるDNA修復に対するスパイクタンパク質の滴定発現の影響。(D,E) 完全長のスパイクタンパク質のみが、非相同末端結合(NHEJ)および相同組換え(HR)のDNA修復を阻害する。値は、3回の独立した実験の平均±SDを示す(図S4A,Bの代表的なFACSプロットを参照)。(F)完全長スパイク(S-FL)タンパク質を導入したHEK293T細胞は、異なるDNA損傷条件において、空のベクター、S1およびS2を導入した細胞よりも多くのDNA損傷を示した。ドキソルビシンは4μg/mL、2時間、γ線照射は 10Gy、30分。H2O2の場合 100 µM, 1 h. スケールバー: 50µm。(G) 3回の独立した実験で得られた20の異なるフィールド(n > 200コメット)のコメットテールモーメントの対応する定量化。統計的有意性は、二元配置分散分析(ANOVA)を用いて評価した。NS(Not Significant)。* p > 0.05, ** p < 0.01, *** p < 0.001, **** p < 0.0001.
3.3. スパイクタンパク質はDNA損傷修復チェックポイントタンパク質のリクルートを阻害する
スパイクタンパク質が核内に存在することを確認するために、細胞内画分解析を行ったところ、スパイクタンパク質は細胞膜画分に豊富に存在するだけでなく、核内画分にも豊富に存在し、クロマチン結合画分でも検出可能な発現量であることがわかった(図3A)。また、スパイクには3つの異なる形態があることを確認した。上のバンドは高度にグリコシル化されたスパイク、中のバンドは完全長のスパイク、下のバンドは切断されたスパイクのサブユニットである。また、コメットアッセイと同様に、DNA損傷マーカーであるγ-H2A.Xの発現が、DNA損傷条件下でスパイクタンパク質を過剰発現させた細胞で確認された(図3B)。最近の研究では,スパイクタンパク質が小胞体ストレスと小胞体関連タンパク質の分解を誘導することが示唆されている[24]。スパイクタンパク質がDNA修復タンパク質の分解を促進することでDNA修復を阻害している可能性を排除するために、NHEJおよびHR修復経路に必須のいくつかのDNA修復タンパク質の発現を確認したところ、これらのDNA修復タンパク質はスパイクタンパク質の過剰発現後も安定していた(図3C)。スパイクタンパク質がNHEJとHRの両方の修復経路を阻害する仕組みを明らかにするために、HRとNHEJの修復にそれぞれ重要なチェックポイントタンパク質であるBRCA1と53BP1のリクルートを解析した。その結果、スパイクタンパク質は、BRCA1と53BP1の両方の病巣形成を顕著に阻害することがわかった(図3D-G)。これらのデータを総合すると、SARS-CoV-2の完全長スパイクタンパク質は、DNA修復タンパク質のリクルートを妨げることで、DNA損傷修復を阻害することがわかる。
Viruses 13 02056 g003 550図3 重症急性呼吸器症候群新型コロナウイルス(SARS-CoV-2)のスパイクタンパク質は、DNA損傷修復チェックポイントタンパク質のリクルートを阻害する。(A)SARS-CoV-2スパイクタンパク質をトランスフェクトしたHEK293T細胞の膜画分(MF)細胞質画分(CF)可溶性核画分(SNF)クロマチン結合画分(CBF)を、His-tagスパイクと指示されたタンパク質についてイムノブロットした。(B)左:空のベクター(E.V.)およびスパイクタンパク質を発現させたHEK293T細胞における10Gyγ線照射後のDNA損傷マーカーγH2AXのイムノブロット。右:左のイムノブロットの対応する定量化。数値は平均±SD(n=3)を示す。統計的有意性はStudent’s t-testを用いて判定した。**** p < 0.0001. (C) スパイクタンパク質を発現したHEK293T細胞におけるDNA損傷修復関連タンパク質のイムノブロット。(D) 10Gyのγ線を照射したE.V.細胞およびスパイクタンパクを発現させたHEK293細胞における53BP1病巣形成の代表画像。スケールバー。10μm。(E)核あたりの53BP1病巣の定量的解析。数値は平均±SEM、n=50を示す。(F) 空のベクターおよびスパイクタンパク質を発現させたHEK293細胞を10Gyのγ線に曝した際のBRCA1病巣形成。スケールバー。10μm。(G). 核ごとのBRCA1病巣の定量的解析。数値は平均±SEM、n=50を示す。統計的有意性はStudent’s t-testを用いて判定した。**** p < 0.0001.
3.4. スパイクタンパク質によるV(D)J組換えの阻害(試験管内試験
DNA損傷修復、特にNHEJ修復は、B細胞やT細胞の免疫の中核をなすV(D)J組み換えに不可欠である[9]。現在までに、mRNAワクチンやアデノウイルス-COVID-19ワクチンなど、承認されている多くのSARS-CoV-2ワクチンは、完全長のスパイクタンパク質に基づいて開発されている[25]。SARS-CoV-2がリンパ球前駆体に直接感染するかどうかは議論の余地があるが[26,27]、感染した細胞がエクソソームを分泌し、SARS-CoV-2のRNAやタンパク質を標的細胞に送達できることを示す報告もある[28,29]。さらに,スパイクタンパク質がNHEJを介したV(D)J組み換えを減少させるかどうかを検証した。そのために,以前の研究[18]に従って,試験管内試験 V(D)J組換えレポーターシステムを設計した(図S5).空のベクターと比較して、スパイクタンパク質を過剰発現させると、この試験管内試験レポーターシステムにおいてRAGを介したV(D)J組み換えが阻害された(図4)。
図4 スパイクタンパク質は試験管内試験でV(D)J組換えを阻害する
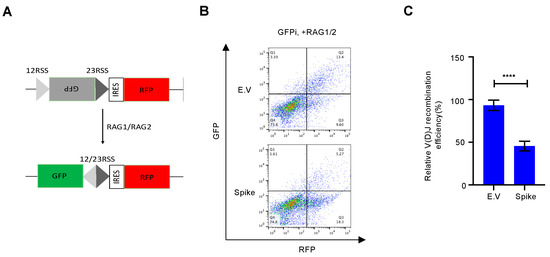
(A) V(D)Jレポーターシステムの模式図。(B) SARS-CoV-2のスパイクタンパク質が試験管内試験でのV(D)J組み換えを阻害することを示すフローサイトメトリーの代表的なプロット。(C) 相対的なV(D)J組み換えの定量的分析。統計的有意性はStudent’s t-testで判定した。**** p < 0.0001.
4. 考察
今回の研究成果は、スパイクタンパク質がDNA損傷修復機構や適応免疫機構をハイジャックしている証拠を示している。我々は、スパイクタンパク質がDNA損傷修復を阻害することで適応免疫を損なう可能性のあるメカニズムを提案する。SARS-CoV-2が胸腺細胞や骨髄リンパ系細胞に感染するという証拠は発表されていないが、試験管内試験のV(D)Jレポーターアッセイでは、スパイクタンパク質がV(D)J組み換えを激しく阻害することが示された。我々の結果と一致するように、臨床観察でもCOVID-19による重症化や死亡のリスクは年齢とともに増加し、特に高齢者が最もリスクが高いことが示されている[22]。これは、SARS-CoV-2のスパイクタンパク質が高齢者のDNA修復システムを弱め、その結果、V(D)J組み換えや適応免疫を阻害するためであると考えられる。対照的に、我々のデータは、DNA損傷修復におけるスパイクタンパク質サブユニットの関与に関する貴重な詳細を提供しており、完全長スパイクベースのワクチンがB細胞におけるV(D)Jの組み換えを阻害する可能性を示している。これは、完全長スパイクベースのワクチンがRBDベースのワクチンと比較して低い抗体価を誘導するという最近の研究とも一致する[28]。このことは、スパイクの抗原性エピトープをSARS-CoV-2ワクチンとして使用することは、全長スパイクよりも安全で効果的である可能性を示唆している。以上のように、我々はSARS-CoV-2が宿主の適応免疫機構を抑制する重要なメカニズムの1つを明らかにした。さらに、今回の発見は、全長スパイクを用いたワクチンの潜在的な副作用も示唆している。本研究は、COVID-19の病原性に関する理解を深めるとともに、より効率的で安全なワクチンを設計するための新たな戦略を提供するものである。
以下はオンラインでhttps://www.mdpi.com/article/10.3390/v13102056/S1,図S1:核に局在するSARS-CoV-2タンパク質のヒト細胞での発現、図S2:核に局在するSARS-CoV-2タンパク質のNHEJ-およびHR-DNA修復経路への影響、図S3: Nsp1, Nsp5, Nsp13, Nsp14は細胞増殖を抑制するが、スパイクは抑制しない、図S4: SARS-CoV-2のスパイク変異株がNHEJ-およびHR-のDNA修復経路に及ぼす影響、図S5:試験管内試験 V(D)J組み換えアッセイ。
著者の貢献
H.J.は、本研究の構想と設計を行った。H.J.とY.-F.M.が研究を監督し、実験を行い、データを解釈した。執筆-原案作成:H.J.; 執筆-査読・編集:H.J. and Y.-F.M.; 資金調達:Y.-F.M. 全著者が本稿の公開版を読み、同意したものとする。
資金調達
本研究は,ウメオ大学医学部のCOVID-19に対する企画助成金(研究プロジェクト番号:3453 16032からY.F.M.へ),ウメオ大学ライオン癌研究財団(助成金:LP 17-2153,AMP 19-982,LP 20-2256からY.F.M.へ),北部医療地域の学術医療機関および大学医療機関での研究に対する拠点ユニットのALF資金(ALF-Basenheten:2019,2020,2021からY.F.M.へ)から支援を受けた。
