Deep sequencing of the Moderna and Pfizer bivalent vaccines identifies contamination of expression vectors designed for plasmid amplification in bacteria | Kevin McKernan
注:未編集、誤訳多いです。
2023年3月1日|米国の大学では、COVIDに感染するリスクが限定的な学生に対して無責任注射(COVIDワクチン)を義務付け続けているため、これらの実験的ワクチンの成分について、より多くの公開情報を入手することが不可欠になっています。EMAとTGAの両機関は、RNAの断片化やウェスタンブロットの汚れについて言及し、ワクチンの製造工程が忠実で透明性に欠けることを示唆しています。TGAのデータが発表された直後、Patelら(Pfizer)はこれらの懸念を払拭しようとする論文を発表しました。
今回の技術発表とhttps://anandamide.substack.com/p/curious-kittens の記事は、この発見を調査したものです。
Kevin McKernanはMedicinal GenomicsのCSO兼創業者で、大麻と麻のゲノミクスを開拓し、大麻ベースの治療法の研究のためのより強い科学環境(http://Kannapedia.net)と大麻遺伝学の追跡と検証のためのブロックチェーン技術を構築しています。
それ以前は、Courtagen Life Sciences, Inc.のCSOを務め、Life Technologiesの副社長兼研究開発部長として、Life Technologiesの次世代シーケンサーSOLiD技術の開発を統括していました。SOLiDの研究開発プロセスにおいて、次世代シーケンサーによる新たな生物学的フロンティアを探求する100以上の共同研究を監督し、特にヒト腫瘍シーケンサーにおける興奮と牽引力を実感しました。ケムFET半導体を用いたDNAシーケンスの研究開発プロジェクトを立ち上げ、DNAシーケンス企業Ion Torrentを3億5000万ドルで買収するプロセスの指揮を執りました。これらの共同研究により、何百もの出版物を生み出し、Science Translational MedicineからNatureまで7つのジャーナルをカバーしました。
2005年に共同設立した新興企業Agencourt Personal Genomicsでは社長兼CSOを務め、ヒトゲノムのシーケンスのコストを3億ドルから3,000ドルに引き下げる革命的なシーケンサー技術を発明しました。2000年、Agencourt Biosciences Corporationを共同設立し、Beckman Coulterに買収されるまでCSOとして活躍。また、ホワイトヘッド研究所/MITのヒトゲノム・プロジェクトの研究開発を担当し、核酸精製に関する複数の特許を取得しました。エモリー大学で生物学の学士号を取得し、ノルエピネフリントランスポーターのクローニングと発現に焦点を当てた。DNAの解読や大麻薬の謎を解明していないときは、ボート、スキー、ガーデニングを楽しんでいます。
PANDAが毎週開催しているオープンサイエンスセッションは、科学、研究、政策について、様々な分野の国際的な第一人者によるプレゼンテーションの機会を提供するものです。これらのセッションは、理解を広げ、新たな洞察力を刺激することを目的としています。
オープンサイエンス・セッションの様子はこちらからご覧いただけます: www.pandata.org/open-science-sessions/
私たちの活動を支援する: www.pandata.org/donate/

ドミニ 0:13
本日のスピーカーはKevin Mckernonさんです。ケヴィンはDNAシークエンス技術の専門家で、特に大麻ゲノム、Psilocybe cubensisゲノム、ポリメラーゼ連鎖反応技術に詳しい。合法大麻の品質と安全性を検査するための遺伝子ベースのプラットフォームを販売するライフサイエンス企業、メディカル・メディシナル・ゲノミクスの創設者兼最高科学責任者です。エモリー大学で生物学の理学士号を取得し、ノルエピネフリントランスポーターのクローニングと発現に焦点を当てました。
以前は、ヒトゲノムプロジェクトの一環として研究チームのリーダーを務めていました。ここ数年、ケヴィンは、OSの咳の存在検査におけるPCRの使用とmRNAワクチンについて広く研究しており、彼はサブスタックのアナンダミドの研究を発表しています。
今日は、ケヴィンがモデルナとファイザーのVaillantワクチンの深い配列決定、バクテリアにおけるプラスミド増幅のために設計した発現ベクターの汚染の特定について話すために来てくれました。それでは、ケビンさん、ありがとうございました。
ケビン・マッカーナン 1:17
わかりました。さて、ご紹介ありがとうございました。この研究はちょっとした偶然で、実は私はこれを扱っていて、大麻畑を実際に壊滅させているホットプレートとウイルス感染を理解するために、RNA配列決定ライブラリーを作っている最中だったんです。
そこで、このRNA配列決定を大量に行い、この生物の病原性の様式は何なのか?そして、なぜこれほどまでに植物に破壊的な影響を与えるのか。ところが突然、よくわからない理由でライブラリーが失敗し始めたんです。
mRNAとは思えない、ゲノムのノンコーディング領域の一部をカバーするような塩基配列が出てきたのです。これは、何かがうまくいかないという兆候でした。そこで、私のネットワークを通じて、この問題を解決するためのコントロールとして使用できる、おそらく換気されたmRNAを持っている人がいないか、募集をかけたのです。
驚いたことに、私のネットワークの誰かが、4本のバイアント(ワクチン)を送ってくれました。しかし、RNAシーケンスライブラリの中身を分解するには、完璧なmRNAになります。おそらく換気されていて、4KBの長さがあり、キャップが付いているからです。
ですから。そこで私は、ライブラリで何が起こっているのかをデバッグするために、これらを使用しました。もちろん、DNAのステップがうまく動作していないことを突き止めるのにも役立ちました。しかし、その過程で、過剰な配列決定が行われることになり、論理的に考えても、100万回以上の配列決定が行われることになりました。
つまり、ワクチンにするための配列の深さは、とてつもなく大きいのです。そうすることで、他の汚染物質が大量に存在することがわかるのですが、これはこれから説明します。
さて、その前に、スライドを進めるかどうか確認させてください。さて、このようなことが始まったのは、過去にmRNAに転写や翻訳の忠実性に問題があるのではないかという文献が多数出てきたことに興味を持ったからです。
このヌクレオチドに関する文献では、ポリメラーゼがこのヌクレオチドを取り込もうとするとエラーが生じ、リボソームが読み込もうとするとエラーが生じることが示唆されています。その結果、ウェスタンブロットには複数のバンドがあり、BansalやJiangなどの論文に見られるような、非常に不鮮明なウェスタンブロットになってしまうのです。
多くの場合、「滑空星座が発生しているため、バンドが本来の姿を現していない」という言い訳がなされます。そこで話は終わってしまうのです。しかし、タンパク質の分野では、この答えに納得がいかない人が多いようです。というのも、一般的には、タンパク質の塩基配列を決定してバンドを特定したり、pngaseのような酵素を使ってグリコシレーションを除去し、ウェスタンブロットで再測定したりするものです。
ジェシカ・ローズとウィキリークスは、このブロックゲートを追跡するために多くの研究を行ってきました。それは、この修飾ヌクレオチドの使用と関係があると思います。この修飾ヌクレオチドは塩基対を変化させることが知られており、その結果、RNAに変換する際の転写エラー率が高くなり、先ほど述べたようにリボソームRNAが増加するのです。
このことについては、非常に優れた論文がありますので、ぜひご覧いただきたいと思います。また、この論文には少し極論があります。私はこの著者たちを知っていますが、彼らはとても良い仕事をしています。しかし、査読を受けると、正直な著者はどうなるかというと、タイトルが変わってしまうのです。

この論文の査読名は、「ヨーロッパのアナログ結合の忠実度を向上させる」です。試験管内試験転写の際、査読を通過すると、編集者は、メチルSUTAウレタンがセキュリティよりも高い忠実度で組み込まれた場合に、に変更します。
彼らは、あなたがすでにタイトルにあるよりも、それらの両方が悪化していることを教えていません。だから、人を油断させるんです。この論文に書かれているのは、T7ポリメラーゼとSP6ポリメラーゼを使ったウレタンのエラーレートは、100万分の1強ということです。
この下にも腱鞘炎の6が見えますね。そして、高学年に切り替えると、やはりこれは300以上に跳ね上がります。そして、また1年に1回メタルスーツに切り替えると、250くらいまで上がります。つまり、この論文の当初のタイトルは、「金属スーツにウレタンを入れると、転写エラー率が2倍になるため、懸念がある」ということを示すものでした。
その代わりに、擬似ヨーロッパとユーロの違いや、1メチル都市編集の違いを見ることにしました。繰り返しますが、人々の注意を、このヌクレオチドを使って実際にワクチンになるmRNAを作る過程で、2~3倍高いエラー率を受け入れているという事実から遠ざけるのです。
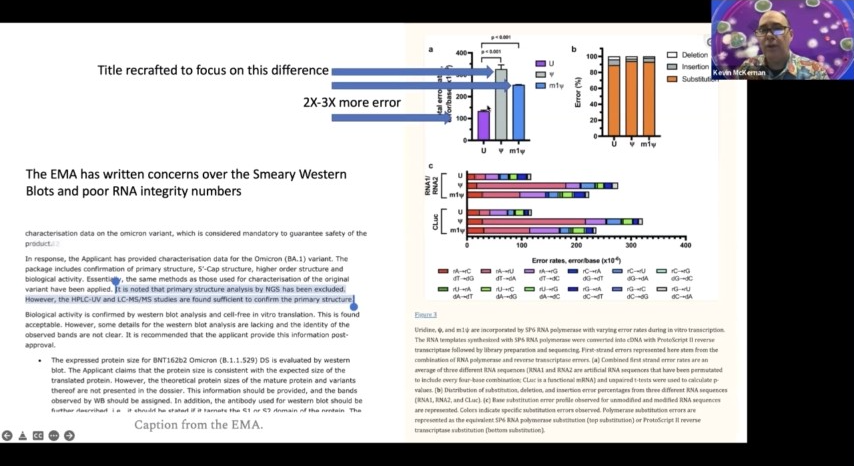
EMAは、RNAの完全性が低いこと、ウェスタンブロットが非常に多いことを指摘しています。また、ファイザーがNGSデータ(NGSとは次世代シーケンサーのデータ)を持っているにもかかわらず、それを除外したことも指摘されています。
なぜ除外したかというと、このデータを見れば、誰もが汚染されていることに気づくからです。なるほど。これが、2021年2月のEMAの文書です。ウェスタンブロット用のウェブページの身元について、いくつかの懸念があることがわかると思います。
十分に正当化されておらず、いくつかの明確化が要求されています。この文書には、臨床バッチの製造工程を実際に生産されるものに切り替えたことに懸念を抱いていることが示されています。DNAテンプレートを合成したものから、プラスミドで作られたものへと変更したのですが、この直線化がどの程度うまくいくかどうかが重要なトピックになりそうです。

公表されていないのですが。また、磁気ビーズによる精製をプロテイナーゼKや私たちが使っている他の精製システムに置き換えています。効率的にスケールアップするための製造方法ですが、IDTから入手できるような従来のCarruthers合成では、十分なDNAテンプレートを合成することができなかったのだと思います。
そこで、プラスミドにDNAを入れることにしたのです。それで、それを増幅することができるようになったのです。大腸菌のDNAを使う場合、様々なリスクがあります。プラスミドDNAは、MRAを作った後にmRNAの中に残留する可能性がありますし、大腸菌を使い始めると、局所多糖類による汚染が心配になり、大腸菌ベースのプラスミドベクターに他の汚染物質が混入する可能性があります。

しかし、ウェスタンブロットについては、より多くの懸念があることがおわかりいただけると思います。ここで興味深いのは、RNAの切断は問題ではない、あるいは無視すべきだという理論が出始めていることです。この言葉の多くは、非常に理論的あるいは仮説的なもので、「これらは急速に分解されることが予想され、ポリアデニル化されるべきではありません」と述べていることに気づきます。
なぜなら、今日お見せするシークエンスからわかることは、これらのベクターの内部にはポリAテールは存在せず、mRNAを合成した後にポリAテールを付加しているのです。ですから、もしmRNA合成後に切断された種があれば、それらもポリアデニル化されることになります。
私は、切断された分子はポリアデニル化されないという主張には、いささか疑問を感じています。配列決定の結果、代替のポリアデニル化部位を見つけることができたというデータもありますが、まだ公表していませんし、そのデータを精査している最中でもあります。
しかし、ここでも前提となっているのは、mRNAが切断されているため、おそらくタンパク質に変化しないだろうということです。そして、当時目の前にあった臨床データでは、これらのmRNAはきれいなものであったため、これらの汚いmRNAは実際には安全であろうと考えたのです。
この潜伏梯子声明が誤りであったことは、今となっては周知の通りです。この文書が書かれたのが2021年2月ですから。ファイザーはこの試験の最初の90日間に12人の123人の死亡者を出しており、安全性がなかったことが分かっています。
安全性が隠されていたにもかかわらず、切り捨てられた種が安全であると仮定するのは、彼らの論理が破綻していると言わざるを得ません。さて、もう1つ知っておくべきことは、これらの文書の中で、EMAが製薬会社に「この指標はどうあるべきか」と尋ねている場面がしばしば見られることです。

これは新薬です。これは新薬で、これまで扱ったことがありません。だから、RNAインテグリティはどうあるべきかを教えてください」ファイザーは「80%であるべきだ」と返しました。なぜ、その数字をどこから持ってきたのかは誰にもわかりませんが、そのスペックを達成できなかったときに、ゴールポストを250%達成できるものへとシフトさせたのかもしれませんね。
つまり、なぜこのような仕様にしたのか、その理由には何の根拠もないのです。次のスライドで非常に重要になるのですが、二本鎖DNAの混入はどの程度まで許容されるのか、EMAは私の鑑賞用として何もないところから数字を出してきましたが、私はEMAは想定していなかったのではないかと感じています。
EMAは大腸菌のバックグラウンドDNAを想定していたのでしょうが、大腸菌の宿主の中で自己増殖する複製起点のあるDNAを想定していなかったのでしょう。大腸菌の細胞内に入り込むと、少量でも大量に増殖する可能性があるため、このような汚染は別の形で懸念されるでしょう。
では、これまで誰がこれらの配列を決定してきたのでしょうか?また、これらのワクチンから得られた配列はあるのでしょうか?パンデミックの初期に偶然に配列が決定され、研究室で配列が決定されGitHubに投稿されたことがあります。
しかし、私はこの研究室に何度も生のシーケンスリードを提供できるかどうか尋ねています。なぜなら、コンセンサスシーケンスは実際には1つのリードを提供するだけで、すべてのリードが表していると思われるものを提供するからです。

コンセンサス配列は1つのリードを提供するだけで、すべてのリードが表していると思われるものを提供することになり、データ内の不一致をすべて隠してしまいます。このような不一致は非常に重要です。もし、切断された種の代替ポリアデニル化部位を探そうとするならば、修飾ヌクレオチドの使用による転写エラーを探そうとするならば、コンセンサス反射ではそれを確認することができません。
コンセンサスが示すのは、データの51%が一致していることですが、稀に存在する可能性のあるエラーはすべて隠されてしまうのです。さて、これが送られてきたファイルです。ロット番号、モデルナとファイザーの価数、ファイザーはこの上にありますね、とても小さいです。
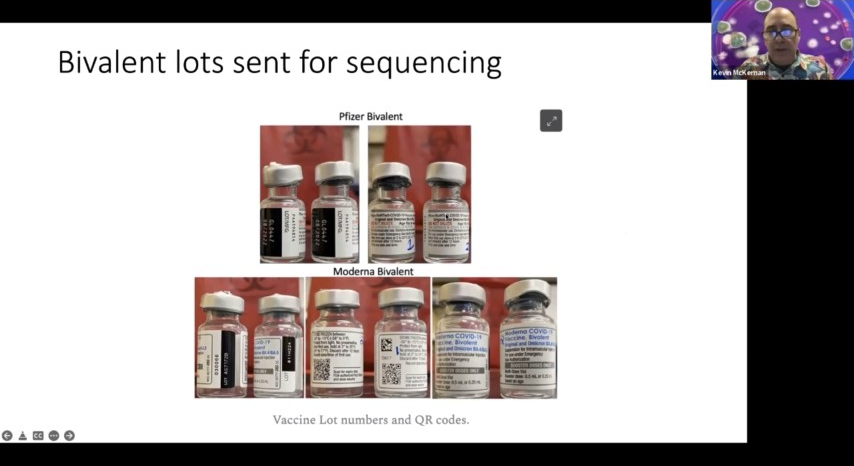
2mlのロットで、6回分の服用が可能です。下のほうにあるのはもっと大きいもので、これだと思うのですが、殺害されたモデルナ社のものは下のほうにあるファイルのほうがずっと大きいです。そこで、まず最初に、これらのファイルを断片分析にかけました。
これはファイザーが以前一価のワクチンで行ったフラグメント分析です。これはキャピラリー電気泳動システムで行われますが、私が使っているものとは少し異なります。アジレントのテープステーションを使用しています。しかし、それでも、このように断片化しているのがわかります。
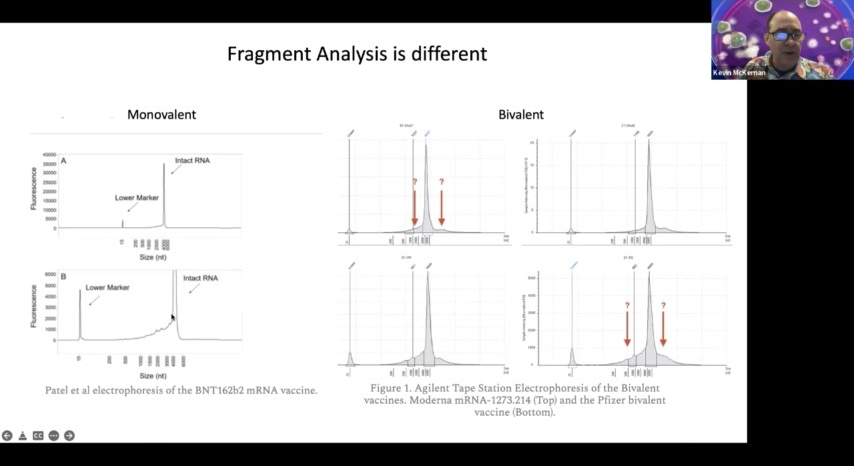
私の矢印は、プレゼンテーションに反映されていますか?つまり、この年は、mRNAがプラスミドのバックボーンから転写されるときに、伸長する過程で失速している可能性が高いのです。メチルSUTAが増殖すると、正しい適合性がないため、どうしたらいいかわからなくなってしまいます。
そのため、そのヌクレオチドを取り込むのに苦労し、前のスライドで示したように、そのヌクレオチドでさらにエラーを起こす傾向があります。酵素が2つほどエラーを起こすと、連続的に3つ目の塩基を作ることができなくなり、結局その鎖から外れてしまいます。
そうすると、短い鎖の配列ができ、その後、ポリテール化するのです。そうすると、その後に生成物ができるわけです。ファイザーやマドンナの場合、RNAの完全性解析の結果、予想されるサイズよりも大きなものが検出されましたが、これは何か別のものです。
それが何であるかは分かりませんが、シークエンスによって、それが何であるかというヒントが得られたと思います。そして、それが最終的にシークエンスデータに反映されたのです。では、どのようにして配列決定を行うのか、これが重要な問題です。
多くの人がナノポアを投入したがりますが、ナノポアはミスが多すぎるため、低発生率を探すのに使うのは無理があります。先ほど紹介したChenの研究でお分かりのように、彼らが示した発生率は100万分の3から400です。

つまり、ワクチン1分子につき1つのミスがあることになりますが、1つのミスが4KBにもなる可能性があります。ですから、もしミスがあったとして、しかも1分子ごとに、それ以上のものを作る1分子シーケンサーを使うのは非常に難しく、機械から実際のエラーレートを見ることができなくなるだけでしょう。
そこで、私たちはイルミナのシーケンサーを使うことにしました。このような方法にはいくつかの妥協点があります。なぜなら、世の中のほとんどのDNA配列はRNAを直接配列するのではなく、まずDNAに変換して、そのDNAを配列します。
そしてもちろん、そのDNAを変換するのです。RNAをDNAに変換する際、先に述べたようなヌクレオチドでつまづくことになります。このn1メチル擬似ウレタンは逆転写酵素にとってあまり良いテンプレートではないので、RNAをDNAに変換する際に、さらに空気が入ってしまいます。
さて、RNAライブラリーを作成する場合、一般的には2種類の重合ステップを経ることになります。私たちは、1回しか行わないものを選びました。なぜなら、転写エラー率を調べるために、エラーの数を制限したかったからです。この方法では、RNAをランダムヘキサマーにします。
ランダムヘキサマーとは、6文字の配列に含まれる4096種類の塩基のすべての組み合わせのことで、これを用いてRNAを逆転写酵素でプライミングします。その過程で。ヘキサマーが自分自身に結合して複製しないようにするために、アクチノマイシンDというものを入れるのです。
そうすると、DNAの増幅は抑制されますが、RTは抑制されません。そうすると、RNA、あるいはRNAからDNAがコピーされ、RNAがDNAに戻り、そのDNAの配列を決定することができます。さて、このDNAの塩基配列を決定するためには、塩基配列決定用アダプターをこの上に置く必要があります。
そこで、第2鎖合成と呼ばれる段階に入り、アダプターを付けることができるようにします。リガーゼは一般に忠実度が高いので、二本鎖DNAを使ってプライマーをリゲートしたい。ステップ6では、ステップ1で作成したDNAが表示されています。
そして、いくつかの酵素を使ってRNAを消去することができます。2本目の鎖を作るために、2本目の鎖の合成が行われるのですが、その際、消去できる塩基を入れるように注意しています。これは重要なことです。なぜなら、これは良いことで、最終的に配列の方向性を理解するのに役立つからです。
さて、このDNAがコピーされたら、再シーケンスプライマーがついた二本鎖のアダプターを接着します。そのためには、DNAの2本目の鎖を作り、T字型のオーバーハングと結合させる必要があります。そして、DNAの塩基自体は破壊しますが、RNAのウラシルは破壊しない酵素で、ユーロの細胞を喜ばせることで、それを消去します。
さて、つまりは、この緑色のDNAと黄色のDNAを一本鎖のDNAに乗せるだけです。一本鎖リガーゼを使えば、このようなことができるかもしれません。しかし、一本鎖リガーゼには他のアーティファクトがあり、シーケンスが不明瞭になる可能性があります。
つまり、重合のステップを1つ経て、ワクチン由来のRNA(メッセンジャーRNA)をDNAに変換し、その配列を決定することができるのです。さて、プラスミドのようにバックグラウンドDNAが存在する場合、コピー数が抑制されることを理解する必要があります。
この方法はRNAを捕捉するように設計されており、通過するDNAの量を減らすために存在しているため、完全にノックアウトすることはできませんが、抑制することは可能です。それでは、イルミナライブラリーを作成しますと、このような感じになります。
さて、ここで見えている赤い点数は、ワクチン製造工程とは関係なく、この方法でRNAシークライブラリーを作るとこうなる、ということです。このような長い断片が含まれることは想定していません。しかし、通常、イルミナでシーケンスされるのは小さな断片がほとんどで、800塩基対以上の分子は、400塩基対以下のものをクラスタリングするようにできています。
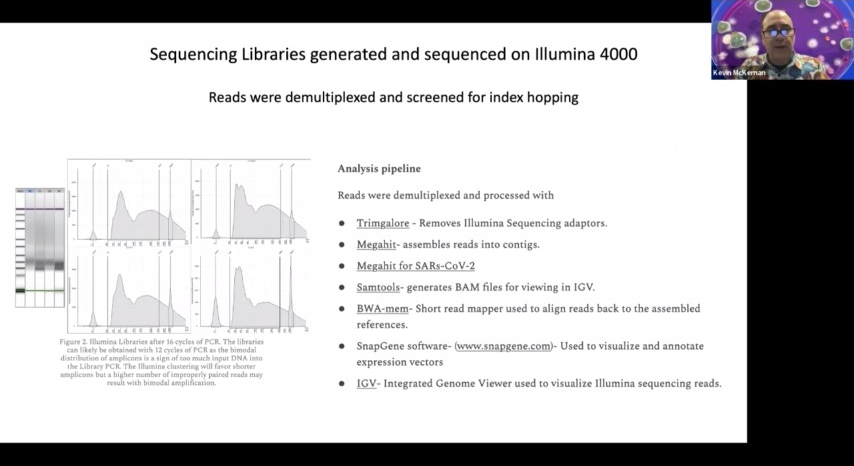
しかし、WindowsをサポートするこれらのmRNAの配列を決定するために、私たちはこのRNAをより小さな断片に断片化し、それを配列し、最後に情報学的な情報をつなぎ合わせます。しかし、4KBのような小さなターゲットであるため、200塩基対のリードは非常に多くのサインを持ち、実際には非常に美しく組み合わされます。
そこで、これらをメガヒットMEGA HITというプログラムでアセンブルしました。メガヒットは、ウイルスゲノム、あるいは少なくともスターゲノムのアセンブルに最適なツールであると発表されています。そして、私たちにとって衝撃的だったのは、これらのプラスミドがアセンブリの中に出現していたことです。
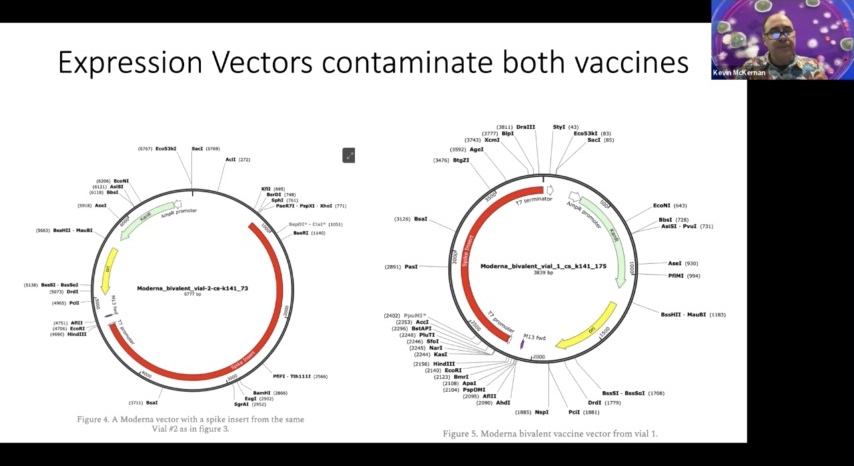
このプラスミドは、スパイクプロテインのインサートがあることを示すものですが、このプラスミドは1,000,000倍でカバーされており、1,000倍以下の範囲にはワクチンやワクチンベクターがありました。複製起点があり、カナマイシン耐性遺伝子が入っていて、app barプロモーター、M 13、プライマーサイエンス、T 7プロモーターがあります。
これは両方のワクチンで同様でしたが、ここではサイズに若干の違いがあります。アセンブラーがオミクロンインサートとウーハンインサートを混同してしまい、スパイクインサートを2倍の大きさにしてしまうことがあるのですが、これは分子の片方がオミクロンインサートを、もう片方がウーハンインサートを組み立てることになり、完全に一致しないため、重複事象のように見えてしまったのです。
他の部分のアセンブリは、それが完全にそのままで、およそ正しい大きさで出てきたのです。ファイザー社のアセンブリは、同一ではありませんが非常によく似た構造をしており、ネオマイシンやアカデミア耐性カセットが共通しています。これはアミノグリコシド耐性カセットで、このWalsallはジェントルミオシンに対する耐性も持っています。
哺乳類の細胞でこれを発現させるためのspプロモーターがここにあります。このf1 oriは、M 13バクテリオファージのコンポーネントのようなもので、必要であれば、実際にこのようなものをToファージにパッケージ化することができます。

他にもいくつかのPollyシグナルがありますが、ワクチンの最後にはシグナルがないことにお気づきでしょうか。つまり、ワクチンはこの方向から始まり、スパイクプロテインはこの方向へと進んでいきます。おっと。そして、SCB恐怖症の切断部位がほぼ真ん中にあるのがわかりますね。しかし、この下にはポリaシグナルがあり、mRNAをコピーしてポリaを得ようとするはずですが、ポリaは重合するのが難しいので、後から重合させるという感じです。
さて、興味深いのは、バクテリアにはこのような高コピー複製起点が存在するということです。さて、私が心配になったのは、ジェシカ・ローズかジャッキー・リークによる生体内分布研究のデータを見たとき、LPSが腸に到達していることがわかったからです。
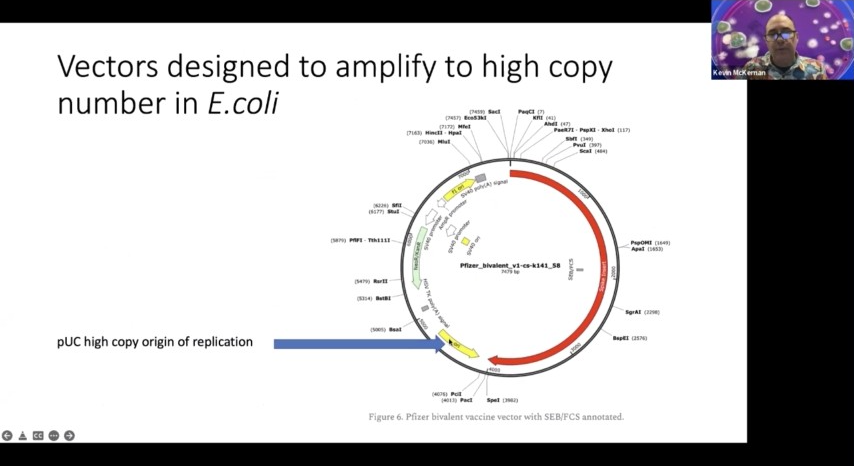
もしLPSが腸に届くなら、プラスミドは必ずしもLPSに含まれている必要はないわけです。LNPが実際にバクテリアの細胞壁をトランスフェクトするのかどうかはわかりません。しかし、腸の温度を上げれば、細菌を実際に形質転換させることができることは知っています。
実際、大腸菌を形質転換する際には、神の体温である37℃に加熱するだけです。ですから、裸のプラスミドDNAは、もし細菌に吸収されるのであれば、そこに持っていくことができるのです。LPSがあれば、途中で消化されることなく、安全に届けることができるかもしれません。
しかし、LPが本当に哺乳類の細胞まで届くように設計されているかはわかりません。しかし、34兆個のmRNA分子を注入する場合、このようなリスクがあります。もし、350個に1個、3000個に1個の割合で、これからお見せする数字がプラスミドだとしたら、何十億個ものプラスミドが注入されることになります。
そのうちの1%が腸に到達し、細菌を形質転換させたとしたら、その細菌はあまり生き残れないかもしれませんね。しかし、患者さんがアミノグリコシドを投与されている場合、生き残るのはプラスミドを投与された菌だけだと思います。というのも、このような抗生物質を使用している場合、腸内細菌をプラスミドに変えてしまう可能性があり、そのプラスミドが大腸菌の内部で生き残り、高いコピー数を複製してしまうからです。
では、これらの比率をどのように測定し、推定すればよいのでしょうか?すでにお話ししたように、私たちはこのための理想的なセットアップを持っていませんし、DNAの塩基配列決定では、DNAよりもmRNAの塩基配列を優先して決定してきました。
しかし、その優先順位がわかっていても、RNAを約220万倍カバーしていることがわかります。これは、T 7の転写が始まる側が分子の中にある場所です。そして、このエリアを超えて、ベクターがある場所にズームインすると、最も高いカバレッジポイントとして約739倍のカバレッジが得られているのです。
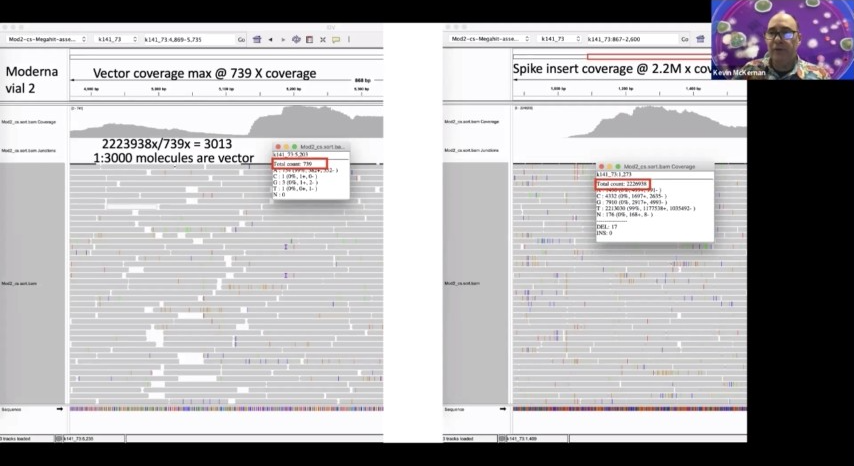
これは3000分の1の割合です。これは、EMAが無造作に選んだスペックから要求されるものにかなり近いものです。ファイザーのバイアルに目を移すと、この数字はもっと悪く、ここでは150万倍、ベクターでは約4,000倍のカバー率で、350分子に1個ということになります。
アクチノマイシンDとRNA配列決定実験によって、このようなことが起こっていることがわかったのですから。つまり、DNAはRNAに比べて抑制されているということです。ですから、これらを測定するためにqPCRアッセイを設計した場合、シーケンシングだけから見た場合よりもはるかに狭い範囲に広がり、実際にはDNAの混入量が多くなることが予想されます。
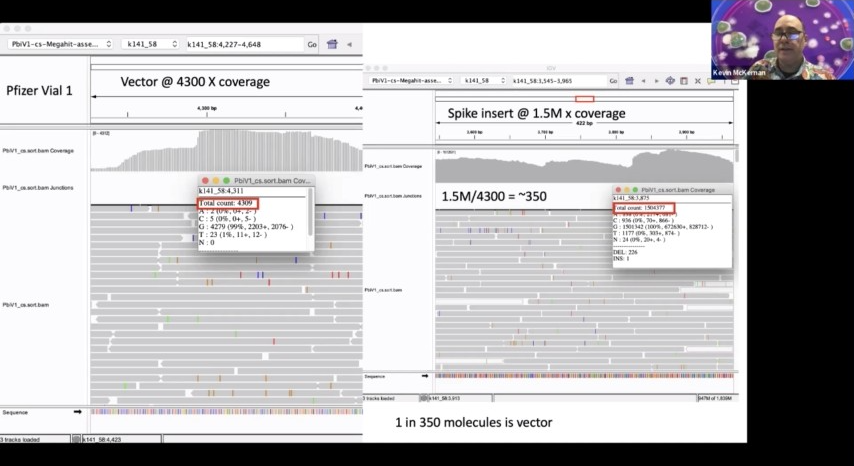
さて、これは彼らの文書に記載されている限界値ですが、ここでは1ミリグラムあたり330ナノグラムとなっており、これは3000分の1に相当します。つまり、両社とも10アドバイザー10に対する失敗を恐れているのです。少なくとも私たちがこれまでやってきたことの次、そして現代はまさにそのライン上にあります。

しかし、このデータがEMAに提出されれば、彼らは都合よく数字を変えてくるでしょうけど、これはこれで、彼らが配置したものです。さて。なぜ、このような規定があるのでしょうか?文献を見ると、二本鎖DNAを注射すると、1型インターフェロン反応を引き起こす可能性があることが分かっています。
しかし、EMAは、このナノグラムがプラスミドのような形で、100万個のプロモーターや細菌の複製成分を含んでいるとは考えていなかったと思います。そうなると、DNAの許容量に対する私の考えも変わってきます。もし、DNAがバクテリアの中で自己増殖し、この薬が腸に届くと知っていたら、もっと厳しい数値になるでしょうね。
では、その他の質問として、得られた配列情報が本当にDNAベクターから得られたものだとどうしてわかるのでしょうか?RNAの塩基配列を決定したのだから?もし、プラスミドから発現したのがRNAだけで、DNAはもう存在しないとしたら?ファイザーは、このようなものを直鎖化して、もしかしたら精製してしまうかもしれないという主張をしていますよね?
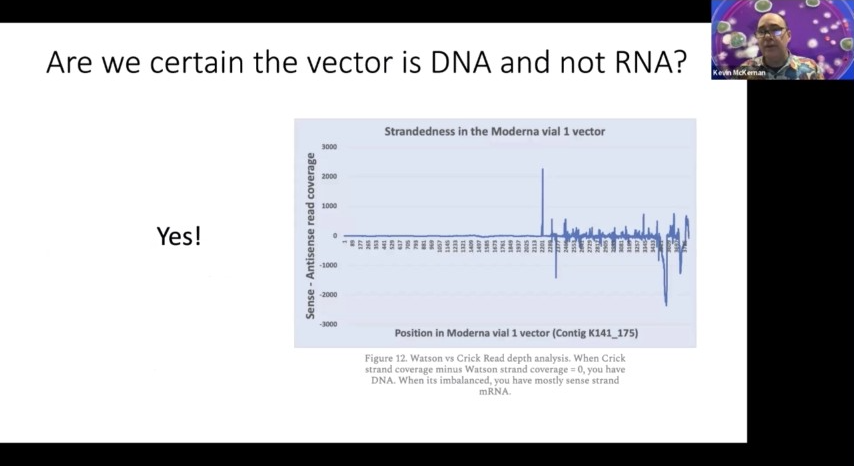
このような方向性のあるRNAシーケンスライブラリーでできることがあります。それは、このDNAを参照ゲノムにアライメントしたときに、これらのDNAがマップする鎖を確認することです。もしワトソンとクリックのアライメントが同量であれば、DNAの空間にいることになります。
ワトソンのアライメントよりもクリックのアライメントが多い領域に入った瞬間、センスRNAを発現している生物であることがわかります。そこで、公開されているシーケンシングリードをスキャンしてみると、ワトソンとクリックの比率はゼロかそれに近い値になっていることがわかります。

そして、T7プロモーターに到達した瞬間、クイックセンス鎖シーケンスが主体となっていることがわかります。つまり、プロモーターに到達するにつれて、ここに大きな偏りがあることがわかります。つまり、これはDNAで、RNAがどれだけあるかを見ることができるのです。
そして、実際にそこにあるのはプラスミドベクターです。このデータから判別できないのは、このデータに敏感なアセンブラを使用していないため、プラスミドが円形なのか線状なのかがまだわからないということです。この問題を解決するには、いくつかの方法があります。
一つは、RNA Cを作り、RNAライブラリーを作って、すべてのRNAを取り除き、DNAの配列だけを非常に深く調べる方法です。そうすれば、1%のリードが実際に円形の状態なのか、原点を越えているのか、あるいは線状化されているのかを数えることができます。
もうひとつは、大腸菌にヒートショックを与えてコロニーを作り、定量性は低いかもしれませんが、少なくとも大腸菌を複製できるものがあるかどうかはわかります。さらに、定量的に分析したい場合は、ベクターバックボーンとmRNAをターゲットにした定量的PCRアッセイを構築し、任意のバイアル内のそれらの比率を監視することができます。DNAとRNAの比率を知るには、DNA塩基やルナーゼを使うのが一番でしょう。RNAシーケンスのようなバイアスはなく、何が起こっているのかを正確に把握することができます。
では、次に何をするかというと、これらのものからRNAを取り出し、それをAST化するのです。RNAsは酵素で、RNase L(この酵素のヒト版)とは異なり、消化に問題があり、メチルSUTAの1つです。尿中RNase aは実際にCとニュースでRNAを切断します。
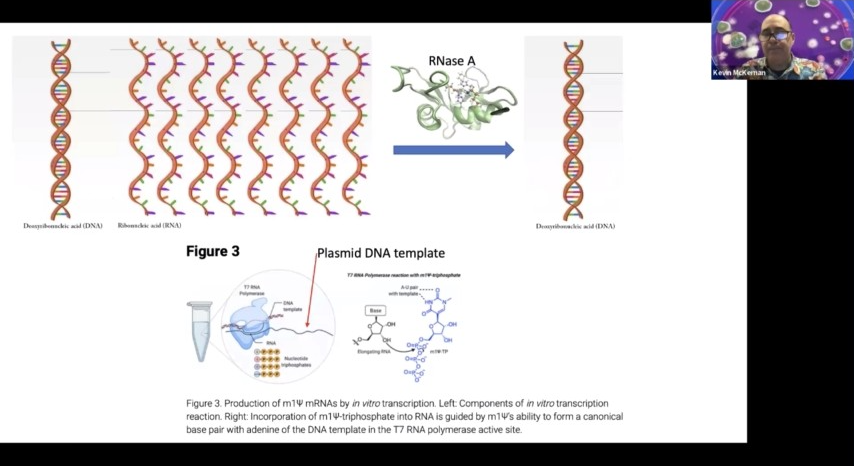
そして、おそらくn one methyl su urienと一緒に使うことで窒息してしまうのでしょう。しかし、Cの部分は詰まらせません。それで、サイズアップを噛み砕くことによって、ワクチン中のRNAをすべて除去するのです。
そこで、DNAを取り出すために、このような方法をとりました。そして、そのDNAを精製したのです。というのも、ご記憶の通り、これはイノシトールが説明するメカニズムなのです。プラスミドを鋳型にして、T7ポリメラーゼがこの鋳型から私たちのmRNAを作るのです。
すみません、この矢印はこっちを向いているはずです。このDNAテンプレートはそのエラーを見逃したのでしょうが、このプロセスでRNAを作っています。このRNAを取り除くと、DNAの塩基配列をより深く理解できるようになります。そして、そのDNAを使って大腸菌に形質転換することもできます。
そこで私は、DHD 10B、あるいは、感受性の高い細胞を作ることができるB Express と言うべきものをいくつか持っていきました。これはカナマイシンに感受性のある細胞で、ワクチンから精製したものをヒートショックさせると、数日後にコロニーが出てきました。
このコロニーを採取して分離してみると、実は大腸菌であることがわかります。大麻産業で何百万回と実施され、サンプルに付着した大腸菌を検出する大腸菌検査は、OACの承認を受けた高品質の検査法です。また、ゲル化すると、ゲノムDNAの他にプラスミドのバンドも確認することができます。
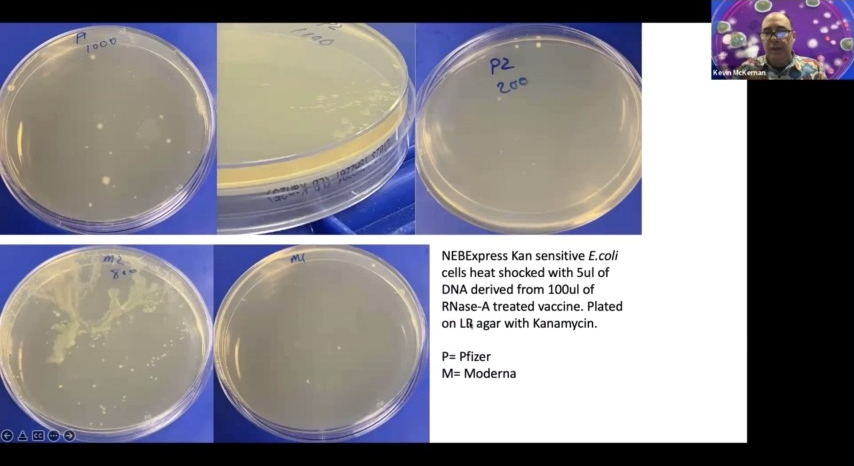
つまり、ワクチンから出たDNAで形質転換する大腸菌の細胞は、間違いなく得られているのです。プラスミドは作られていますが、そのプラスミドがファイザー社の母体血漿であることを証明する塩基配列情報はまだ得られていません。というのも、私は以前、このようなことがうまくいかないのを見たことがあるので、かなり警告を発しておきたいのです。
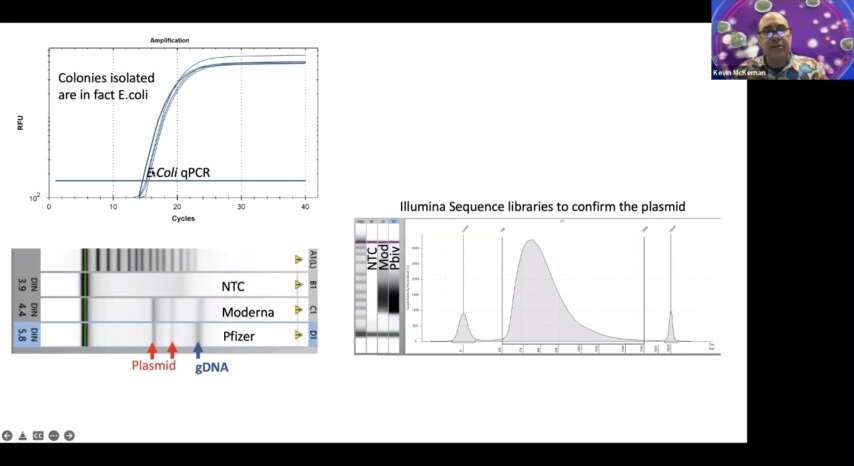
しかし、大腸菌の中にはこのように他のプラスミドが浮遊しており、もしかしたら私たちが形質転換したと思っていたものとは違うかもしれません。しかし、RNA化したワクチンと一緒にヒートショックすれば、カナマイシンで生き残るコロニーができることは分かっているのです。
そこで、現在、このプラスミドの塩基配列を決定するために、塩基配列決定ライブラリーを作成中です。そして、そのプラスミドが実際に大腸菌を通過しているかどうかについては、2週間ほどで情報が得られると思います。このことから、循環型の物質が存在する可能性があり、実際に患者さんのGiを変化させる可能性があることがわかります。

しかし、長期的には、業界全体として、この問題に対処するためのqPCRアッセイを構築することが必要だと思います。母乳中にこのmRNAが存在することを示す論文はいくつかあります。サイバーグリーンアッセイはTaqManアッセイよりも特異性が低く、Plexテストとして1回しか使用できません。
だから、サイバーグリーンアッセイに内部統制を入れるのは難しいんです。私は、業界にとってより良い方法は、モデルナ・ファイザーワクチンをターゲットにしたqPCRアッセイで、2価ワクチンと1価ワクチンの両方にヒットし、C 90にはヒットしないことだと考えています。
これにより、ベクターがどの程度通過し、他の組織(背中や負傷した患者など)に到達しているかどうかを定量化することができます。また、ワクチン瓶のQCにも利用できます。ワクチン瓶のベクターとインサートの比率はどのくらいか。
さらに、C 19のヌクレオカプシドをターゲットにしたものもあります。こうすることで、血清サンプルや組織サンプル、病理学研究室などで「これはワクチンによるものなのか?」定量的PCRを行えば、その組織からVAX RNAの量がわかります。C19のヌクレオカプシドがあれば、この患者はワクチン接種と感染の両方を経験していることになりますね。
同様に、ベクターの骨格がどれくらいあるのか、アミノグリコシド系薬剤の使用で合併症が起きた場合、この後に消化器系の苦痛がある場合などに備えて、このような基準をすべて満たすようなアッセイを構築できないか、試行錯誤しているところです。
スパイク・プロテインやスパイクmRNAの血液供給を望まない人々がいるのです。残念ながら、今現在、すべての血液が汚染されていると思われます。もし他に質問があれば、このサブスタックにもっとたくさんありますし、私にも連絡をください。コメント欄もありますし、Kevin zone mckernon at medicinal genomics.comにも連絡ください。
