SV40 origin of replication in mammalian cells in absence of SV40 Large T-Antigen
https://anandamide.substack.com/p/sv40-origin-of-replication-in-mammalian
SV40 Large T抗原なしでの哺乳類細胞における複製の起源
ワクチン接種後1年経過した腫瘍生検のqPCR
アナンダミド
2024年10月15日

記事の解説
- Pfizerのワクチンに含まれるSV40ウイルスのDNAが、予想外の方法で人体の細胞内で複製される可能性が示唆された。通常、このような複製には特定のウイルスタンパク質(SV40大型T抗原)が必要だが、それなしでも起こり得ることが分かった。
- Pfizerのワクチンには、SV40ウイルスの複製起点(SV40 origin of replication)が含まれているが、SV40大型T抗原は含まれていない。
- これまでは、SV40複製起点を含むDNAが哺乳類細胞内で複製するためには、SV40大型T抗原が必要だと考えられていた。しかし、この研究では、SV40大型T抗原がなくてもワクチンDNAが哺乳類細胞内で複製できる可能性が示唆された。
- この複製は、ワクチンDNAに含まれる特殊な遺伝子配列(ColE1起点、F1起点、ベータグロビンUTRs)によって可能になると考えられる。これらの配列は、細胞にDNAの複製を指示する役割を果たす。
- 研究者たちは、実験室で培養した人間の細胞(OVCAR3細胞)にワクチンDNAを導入したところ、DNAに予想以上の頻度で変異(遺伝子の変化)が見られた。これは、DNAが複製される過程で起こったエラーだと考えられる。
- さらに驚くべきことに、ワクチン接種から1年後の患者の腫瘍サンプルからもワクチンDNAが検出された。その量は、人間の通常のDNAと同等かそれ以上だった。これは、ワクチンDNAが長期間にわたって体内で複製され続けている可能性を示している。
- このDNAは、人間の遺伝子に組み込まれるか、細胞内で独立して複製されている可能性がある。どちらの場合も、予期せぬ健康影響を引き起こす可能性がある。
- これらの発見は、ワクチンの安全性に関する現在の規制(FDAによるDNA量の制限など)が不十分である可能性がある。
- また、この現象は「ワクチンのシェディング」(ワクチン接種者から未接種者への何らかの影響の伝播)という議論の的になっている現象を説明できるかもしれない。
ワクチンの「シェディング」について:
1. シェディングとは:ワクチン接種者から未接種者へ、ワクチンの成分や影響が何らかの形で伝わる現象を指す。これまで多くの専門家がこの可能性を疑問視してきた。
2. 従来の疑問点:著者は、従来のシェディング仮説に懐疑的だった。その理由は、仮に接種者から未接種者に少量のスパイクタンパク質が伝わったとしても、その量が病気を引き起こすほど多いとは考えにくかったためだ。
3. 新たな可能性:しかし、今回の発見により、シェディングのメカニズムに関する新たな可能性が示唆された。ワクチンDNAが自己複製能力を持つことが判明したためだ。
4. 自己複製DNAの影響:もし接種者から未接種者に少量の自己複製可能なDNAが伝わった場合、それが未接種者の体内で増殖する可能性がある。これにより、従来考えられていたよりも大きな影響が生じる可能性がある。
5. 伝播の経路:著者は、スパイクタンパク質を含むエクソソーム(細胞から放出される小胞)にこのDNAが含まれている可能性を示唆している。これが未接種者に伝わる経路となる可能性がある。
6. 従来の理論との違い:この新たな仮説は、従来のシェディング理論とは異なり、少量のDNAでも長期的に影響を及ぼす可能性を示唆している。
SV40 Large T抗原
以前、BNT162b2プラスミドDNA複製の証拠を示すBNT162b2トランスフェクトOVCAR3細胞株について説明した。SV40の複製は通常、SV40 Large T抗原を必要とするため、これは不可解である。また、ファイザー社のワクチンにはSV40は含まれていない。
そこで私は、Consensus.appという新しいLLMツールを試してみることにした。これは、文献検索用に調整されたChatGPTのようなものである。Nick Jikomesが私にこのツールを紹介してくれた。
このLLMはすぐに答えを出した。
SV40複製起点のほ乳類細胞でのColE1による複製
ほ乳類細胞における複製と安定性:
SV40複製起点とf1バクテリオファージ複製起点を含むSV40ベースのシャトルベクターは、ほ乳類細胞内で複製できる。これらのベクターは一本鎖DNAから二本鎖分子を形成し、再配列の低レベルで安定性を維持できる。しかし、UV誘発損傷を持つヘテロ二本鎖分子は鎖損失の割合が高く、複製中に損傷したDNA鎖が選択的に失われることを示している。
複製の細胞周期調節:
SV40複製起点におけるDNA複製の開始は、細胞周期特異的な因子によって調節される。S期細胞の抽出物は、G1期細胞と比較して複製開始が著しく効率的である。SV40 T抗原(ウイルスタンパク質)と共に特定の巻き戻し活動が、SV40複製起点での部位特異的な開始に必要であり、細胞がS期に入る直前に出現する。
結論
SV40複製起点は、ColE1複製起点と併用すると、ほ乳類細胞での効率的な遺伝子発現と安定した複製を促進する。複製過程は細胞周期特異的な因子によって厳密に制御され、S期には開始効率が顕著に増加する。これらのベクターの安定性は一般に高いが、特にヘテロ二本鎖分子では損傷誘発性の鎖損失が起こりうる。これらの知見は、ほ乳類細胞研究におけるSV40ベースのベクターの有用性と複雑さを浮き彫りにしている。
いくつかの参考文献へのリンク。
ファイザー製薬のワクチンに詳しくない人のために説明すると、このワクチンには3つの複製起点が含まれている。SV40 ori、ColE1 ori、F1複製起点である。コンセンサスでは、SV40複製起点がSV40大型腫瘍抗原なしでも哺乳類細胞で複製できることを示すいくつかの論文が見つかった。必要なものは、コロニー化1(ColE1)とF1の複製起点であり、これらはファイザー社のプラスミドに存在する。SV40複製起点の3番目の複製メカニズムは、ベータグロビン配列に存在するS/MARドメインを必要とする。ファイザー社のワクチンにはベータグロビンのUTRも存在し、S/MARドメインを模倣する能力についてさらに調査する必要がある。
複製メカニズムが明らかになったことで、これまで収集してきた証拠をよりよく説明できるようになった。例えば、ヒト細胞株にトランスフェクトした場合にのみワクチンで確認される変異について、その説明が可能になる。裸のワクチンを配列決定しても、プラスミドのバックボーンに変異は見られない。OVCAR3細胞にトランスフェクトした後のワクチンを配列決定すると、複製起点にSNPが確認される。
これは、ポリメラーゼ複製エラーのみから、このような高頻度(任意のSNPで変異を示すリードの10%以上)の変異が見られるとは思われないため、困惑する。ポリメラーゼ複製エラーは通常、1万から100万に1回の頻度であり、それらを確認できるほどシーケンスの深度がない。他の何らかのメカニズムが、このような高頻度(100リード中14リードがSNPを確認)を生み出しているに違いない。Consensus.appで提供された論文は、そのメカニズムを明らかにしている。
この点をしっかりと理解することが重要である。
PCRキットで使用されているpUC ori(ColE1)が、ColE1 ori下のSNPの検出に寄与している可能性があるという指摘もある。これはもっともな指摘であるが、当社のキットでは、ColE1 oriプライマーセットでは通常、NTCシグナルが35 CTを超えている。CT値が28未満(通常は22未満)のサンプルのみを配列決定している。35CTの混入により、IGVで10%を超える対立遺伝子頻度が見られるとは思わない(7deltaCTは128分の1の負荷である)。また、これらのSNPは、ポリメラーゼキットのバックグラウンドに存在しないSV40オリジンおよびF1オリジンでも見られる。
これはエラーが複製によるものであることを示唆しており、これらの論文は複製エラーが最も起こりやすい領域は複製起点でよく見られるヘテロ二重鎖にあることを示唆している。これは、これまで私たちが観察してきたデータに最もよく当てはまる。
まとめると、
1)ワクチンが細胞内に入っていることは明らかであり、そうでなければこのような突然変異は起こりえない。裸のワクチンをコントロールとして配列決定すると、変異は見られない。ワクチンがヒト細胞に導入されると、変異が現れる。
2) DNAが複製されており、Consensus.appでいろいろと試すまでは、SV40 Large T抗原が存在しないのにこのようなことが起こる理由について、納得のいく説明ができなかった。
3) DNAが哺乳類細胞で複製されているのであれば、すでにファイザー社のワクチンで投与されているので、自己増幅型mRNAワクチンは必要ない。
自己複製可能なDNAを注射することの意味とは何だろうか?
何よりもまず、哺乳類の複製起源を持つDNAを含むLNP(リポソーム核酸粒子)であれば、その規制をトラックで通過できるため、FDAのDNA10ngに関する規制はもはや無関係である。
私は常に「シェディング」に関する文献に懐疑的であった。それは、ピエール・コリー博士や他の人々が提示した仮説に関する逸話や優れた説明が不足しているからではなく、
ピエール・コリーの医療に関する考察
ワクチン接種者から未接種者へのコビッドmRNAワクチン成分および製品の「シェディング」 – パート1
1年前 · 610いいね · 261コメント · ピエール・コリー医師、MPAhttps://pierrekorymedicalmusings.com/p/shedding-of-covid-mrna-vaccine-components
しかし、私はその投与量についてどうしても理解できなかった。たとえBansalらが示唆するように、スパイクで覆われたエクソソームを少量排出したとしても、受容者へのスパイクの投与量はごく微量である。それなのに、どうして受容者に病気を引き起こすことができるだろうか? また、ワクチン接種による被害(すべてではないが)の多くは、Marc Girardotが提唱したボーラス理論に関連していると思う。シェディングされた受容者は、このリスク要因にさらされることはない。
これらのエクソソームのいずれかに自己複製DNAまたはプラスミドが含まれている場合、投与量計算は完全に変わる。 少量の放出プラスミドがレシピエント内で増殖し、この現象を説明できるかもしれない。
もしDNAが細胞内で複製されているのであれば、ワクチン接種を受けた患者におけるDNAの持続性に関して重大な影響がある。 研究室で作成されたプラスミドが核カプシドに対するFP qPCRテストを誘発するケースメディックを生み出した例がある。 ファイザー社のプラスミドは、この論文のように大腸菌でも複製するように設計されている。
核カプシドをエンコードするプラスミドが研究室スタッフに定着した興味深いケース
アナンダミド 2023年4月16日
DNase I阻害剤について検索している際に、興味深い論文を見つけた。これは、BSL2の研究室スタッフがNucleocapsid C19 RT-qPCR検査で偽陽性を引き起こした事例を記録したものである。通常であれば、これは見過ごされ、C19の無症候性感染の一連のケースとして片付けられていただろうが、この人々はさらに深く掘り下げた。同じ研究室でプラスミドを扱っていたようである… 全文を読む
この複製能を有するワクチンDNAは患者体内でどのくらい持続するのか?
私たちは現在、ワクチン接種を受けた患者の腫瘍サンプル中のワクチンDNAについてqPCRを実施しているところである。ワクチン接種から1年後でも、RNAPヒトゲノムコントロールと同等またはそれ以上の量のqPCR陽性サンプルが見つかっている。
これは何を意味するのか?RNAPは、ヒト遺伝子を標的とする一般的なqPCRアッセイである。これは、内部コントロールとして一部のCOVID-19 PCRキットで使用されている。このPCRテストでは、鼻腔スワブで採取したヒト細胞の数とワクチンRNAを比較する方法がないため、臨床的に怠慢なテストとなっている。鼻腔スワブで採取するヒト細胞の数は1000倍も異なる可能性があるため、RNAPコントロールなしではウイルス量を算出できない。
もしファイザー社のワクチンからSV40のqPCRを行い、1年後のサンプルでヒトのRNAP遺伝子と同じかそれ以上のコピー数であった場合、SV40 DNAは時間とともに希釈されておらず、ゲノムDNAとともに複製されているように見える。これはSV40 DNAが細胞分裂のたびに統合またはエピソーム複製されていることを示す兆候である。
これは想定される結果ではない。ここで関係する体積について、簡単な希釈計算をしてみよう。
この患者はファイザー社のワクチンを4回摂取した。最後の摂取は生検が行なわれる1年前であった。この生検から約30日後に死亡した。
ワクチン300ul~1200ul(ファイザー製薬の1~4回分)が87,000mlの体積(人間の平均的な体積は87リットル)に希釈された場合、ワクチンが体積全体に均等に分布していると仮定すると、ワクチンDNAは1日目に少なくとも64,000倍に希釈されるはずである。もしDNAが広告通りに腕に留まっているのであれば、結腸腫瘍の生検にはDNAは見られないはずである。
64,000倍の希釈は、300ulのワクチンに見られる元のDNAシグナルと比較して、qPCRで16CT後の(2^16)シグナルとなる組織で明らかになる。
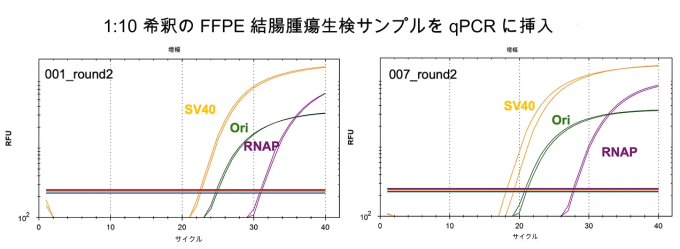
しかし、1年後のワクチンと類似したCT値を示す組織が発見されている。これは、ワクチンDNAが組み込まれ増幅されているか、DNAがエピソームプラスミドとしてこれらの複製起点によって複製されている場合にのみ起こり得る。
もし私たちのラボまたは検体を採取したラボ(INMODIA GmbH)からのワクチン汚染であった場合、スパイク、SV40プロモーター、ColE1 OriのCT値が等しくなるはずである。なぜなら、ほとんどのファイザー製ワクチンには、これら3つの標的がすべて同量含まれているからだ。スパイクアッセイでは、驚くべきことに、この生検ではDNAがまったく検出されなかった。これは、モダナ社のロットによっては、RNA:DNAハイブリッドのDNaseI処理が不完全であるため、スパイクDNAの濃度が6CT(以前)高くなることがあるが、これは異常である。Speicherらはこのアーティファクトを実証しているが、当社がこれまで調査したバイアルにおけるファイザー社の製造プロセスでは、このアーティファクトは認められない。
代わりに、スパイクシグナルのないSV40プロモーターとColE1 Oriオフセットが観察され、NTCはCT 35までクリーンである。
また、実験室でのワクチン汚染も、シーケンスデータで確認されている変異体をもたらすことはない。OVCAR3の研究で示されているように、裸のワクチンDNAをFFPE gDNAと単純に混合してシーケンスしても、細胞が作り出しているSNPは確認できない。このDNAが細胞にトランスフェクションによって改変されたことを示す非常に特異的なシグネチャーがあるが、これは、詐欺を企てるために研究者がでっち上げることができるようなものではない。
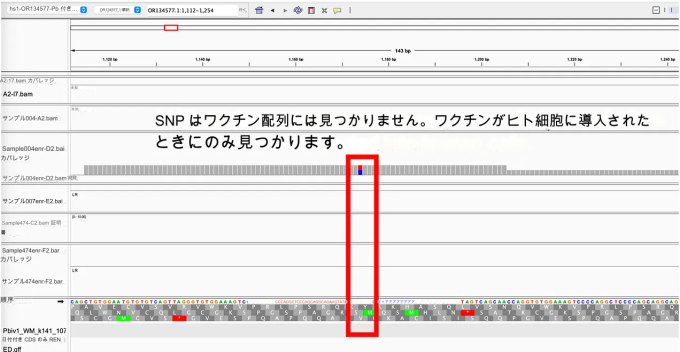
なぜ現在、SV40プロモーターとColE1 Ori qPCRのみが検出されるのか?
一部のバイアルに少量の空ベクターが含まれている可能性がある。これは、スパイクインサートがプラスミドにクローニングされなかったDNAの混入である。また、大きなインサートプラスミドの再配列で悪名高いColE1複製起点によるプラスミド複製により、スパイク配列の一部が削除された可能性もある。データにはスパイク配列がいくらか見られるが、当社のqPCRアッセイが標的とするスパイク配列の領域で何が起こったのかを確かめるには、カバレッジの深さが十分ではない。
結論
ファイザー製薬のワクチン配列は、スパイク陽性の IHC 大腸がん生検において、ワクチン接種から1年後に検出された。ワクチン接種後の核酸の最長検出期間は、Roltgen(60日)によるものである。
これらのサンプルのCT値が非常に低いのは、ワクチンDNAの複製能力またはゲノム統合と増幅によってのみ説明できる。これらのサンプルの予備的な配列決定により、qPCRが標的以外のシグナルではないこと、および変異体がプラスミドに存在することが確認された。これは、ワクチンDNAの複製エラーまたは生体変換を示唆している。ワクチンDNAの統合またはエピソーム複製を解明するために、さらに詳細なシーケンスが行われている。
シーケンス実験の詳細
このサンプルのシーケンスカバレッジを0.6Xに設定し、FFPE DNAのシーケンスの精度を評価した。FFPE DNAは、しばしば架橋結合され、アバシス部位を含むため、シーケンスが困難であることで知られている。
シーケンスでは、統合イベントが見つからない可能性が高い小さな挿入物(断片化DNA)が大量に検出された。それ以来、NEBの最新FFPEライブラリ構築キットを使用して、新しいシーケンスライブラリを構築した。このキットは、イルミナシーケンスライブラリを構築する前にDNAのabasicサイトを修復し、わずか8サイクルのPCRで、患者の死亡前後に採取した生検用の30ulのライブラリに30ng/ulを確保した。


これらのライブラリーのDNAの一部(20ul)をプールし、IDT xGENロックダウンプローブ増強法で使用した。このプロセスでは、PfizerとModernaのプラスミドと配列が共通するDNAをすべて「釣り針で引っ掛ける」ため、シーケンサーはプラスミドのような配列に焦点を当てることができる。これは、ワクチンと配列相同性を共有するサンプル領域に対して、より深いシーケンスカバレッジを提供することを目的としている。
簡単に説明すると、214のビオチン化100マープローブが合成され、ハイブリダイゼーション捕捉試薬として使用された。イルミナライブラリーにハイブリダイズすると、ストレプトアビジン磁気ビーズがビオチンと結合し、磁石を使用してワクチン様配列を回収する。
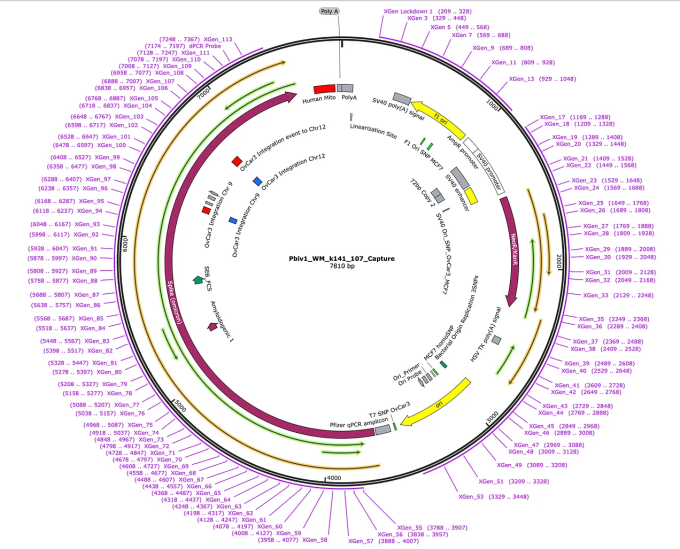
方法
IHC陽性であった結腸腫瘍生検がDNA解析用に選択された。DNAはINMODIA社で精製され、qPCRと配列解析のためにマサチューセッツ州ビバリーにあるMedicinal Genomicsに送られた。
1:10に希釈した2ulのDNAを、前述の18ulのqPCRアッセイで使用した。NEBNext UltraShear™ FFPE DNA Library Prep Kit (NEB #E6655)を使用して、陽性サンプルを優先的に配列決定した。14ulのFFPE DNAを15ulのddH20で希釈し、ライブラリー準備プロセスへの29ulの初期インプットとした。NEBNext® Multiplex Oligos for Illumina® (#E7600S) を使用して最終ライブラリーを増幅するために、8サイクルのPCRを利用した。
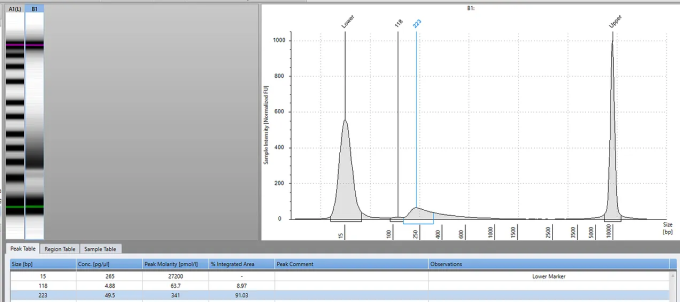
PCRサンプルは0.9X Ampureを使用して精製し、インサートサイズと量を評価するためにAgilent Tape Stationで評価した。
シーケンスは、デュアルインデックスバーコードを使用して、Illumina NovaSeqで2x150bpリードとして実施した。
解析
ReadはTrimmomaticでトリミングし、IlluminaのDRAGEN mapperを使用してGRCh38.p14にマッピングした。ReadマッピングはIGVで可視化した。
MedGen qPCRキットのアップデート
アナンダミド
·
2月8日
MedGen qPCRキットのアップデート
これらのqPCRプライマーセットは、Pfizer、Moderna、Janssenのワクチンに対してスクリーニングされている。スパイクアッセイは、3つのワクチンすべてで陽性である。ベクターオリジンプライマーは、Janssen(異なるアデノウイルスベクター)では約10CTの遅延があるが、PfizerとModernaでは同様に増幅される。