Sequencing of bivalent Moderna and Pfizer mRNA vaccines reveals nanogram to microgram quantities of expression vector dsDNA per dose
ケビン・マッカーナン、イヴォンヌ・ヘルバート、リアム・T・ケイン、スティーブン・マクラフーリン
メディシナルゲノミクス、100 Cummings Center, Suite 406-L, Beverly Mass, 01915
要旨
モデルナとファイザーの二価mRNAワクチンの期限切れバイアル4本の核酸組成を評価するために、いくつかの方法が展開された。各ベンダーの2本のバイアルは、イルミナシーケンス、qPCR、RT-qPCR、Qubit™ 3蛍光測定、Agilent Tape Station™電気泳動で評価した。複数のアッセイで、欧州医薬品庁(EMA)の330ng/mg要件およびFDAの10ng/投与要件を超えるDNA混入が確認された。ワクチンmRNAを標的としたRT-qPCRアッセイは、RNaseまたはDNaseヌクレアーゼ処理なしではDNAとRNAを識別できないため、これらのデータは母乳または血漿中のワクチンmRNAの監視に影響を与える可能性がある。同様に、LINE-1とワクチンmRNAの逆転写酵素活性を評価する研究では、ワクチンに含まれる高レベルのDNA汚染を考慮する必要がある。線状の断片化したDNAと無傷の円形プラスミドDNAの正確な比率は、まだ調査中である。DNA汚染を追跡するために使用される定量的PCRアッセイについて説明する。
はじめに
いくつかの研究で、母乳や血漿中にワクチンのmRNAが長期間存在することが指摘されている(Bansalら2021年、Hannaら2022年、Castruitaら2023)。これは、ワクチンのmRNAにおけるN1-メチルシュードリジン(m1Ψ)の安定性の結果であると考えられる。Nanceらは、ワクチンmRNAの試験管内試験T7ポリメラーゼ合成の前に大腸菌で最初に増幅されるdsDNAプラスミドを利用するワクチンmRNA合成方法を描いている(Nance and Meier 2021)。このDNAの除去に失敗すると、修飾RNAよりも安定なスパイクコード化核酸が注入される可能性がある。EMAは、RNAに対するDNAの330ng/mgでの制限を明言している(Josephson 2020-11-19)。FDAは、ワクチンにおける10ng/dose未満のガイダンスを発表している(Sheng-Fowler et al.2009)。
注入されたDNAが残留すると、I型インターフェロン反応が起こり、DNAの統合の可能性が高まる可能性がある(Ulrich-Lewis et al. 2022)。
結果について
ワクチンの核酸組成を評価するために、2つの異なる方法を用いてワクチンDNAの塩基配列を深く決定した。最初の方法は、市販のNew England Biolabs社製RNA-seq法を用いたもので、RNAの配列決定に有利であったが、予期せぬDNAベクターに対して500倍以上のカバレッジを示した(図1および2)。2番目の方法は、RNase A処理でRNAを除去し、Watchmaker Genomicsフラグメントライブラリーキットを用いてDNAのみを配列決定するものである。DNAに焦点を当てたアセンブリーでは、よりインタクトなポリAトラクトを持つベクターアセンブリーが得られた(図3)。
これらのアセンブリを利用して、ワクチンmRNAとDNAベクターの両方に存在するスパイク配列を標的とし、DNAベクターのみに存在する複製起点配列も標的とする多重qPCRおよびRT-qPCRアッセイを設計した(図3)。ファイザーバイアル1のアセンブリには、ファイザーバイアル2のアセンブリには存在しない72bpの挿入が含まれている。このインデルは、SV40プロモーターと核局在化シグナルを増強することで知られている(Dean et al. 1999)(Moreau et al. 1981)。
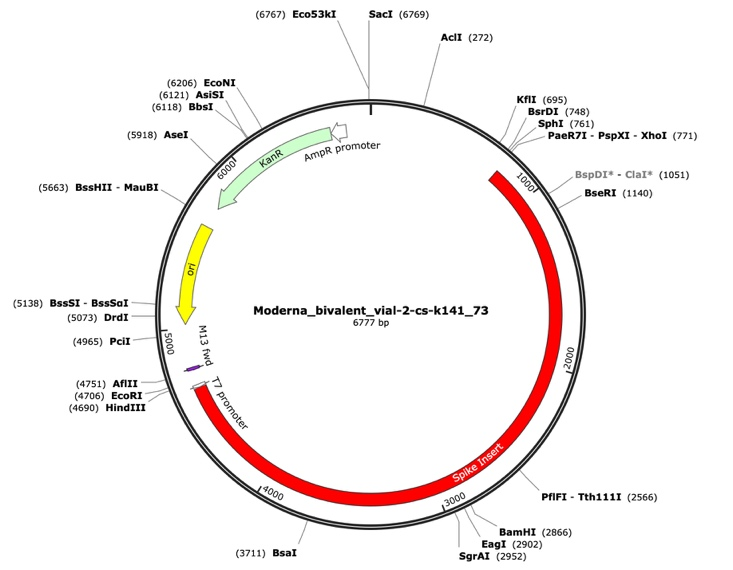
図1 スパイクインサート(赤)、AmpRプロモーターで駆動するカナマイシン耐性遺伝子(緑)、高コピー細菌複製起源(黄)を有するRNA-seqライブラリーのモデルナベクターアセンブリ
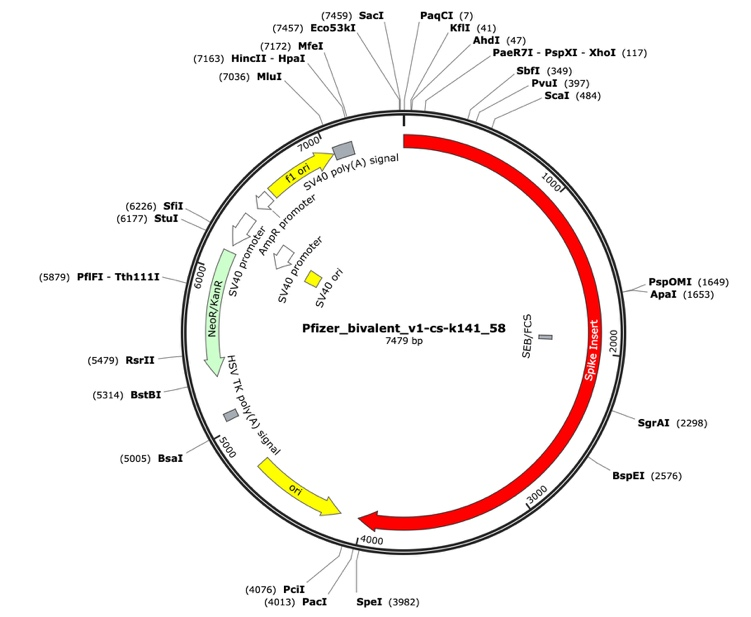
図2 RNA-seqライブラリのPfizer二価ワクチンアセンブリ
SEB/FCS、スパイクインサート(赤)、細菌複製起源(黄)、Neo/Kan耐性遺伝子(緑)、F1起源(黄)、SV40プロモーター(黄と白)で注釈付けされている。
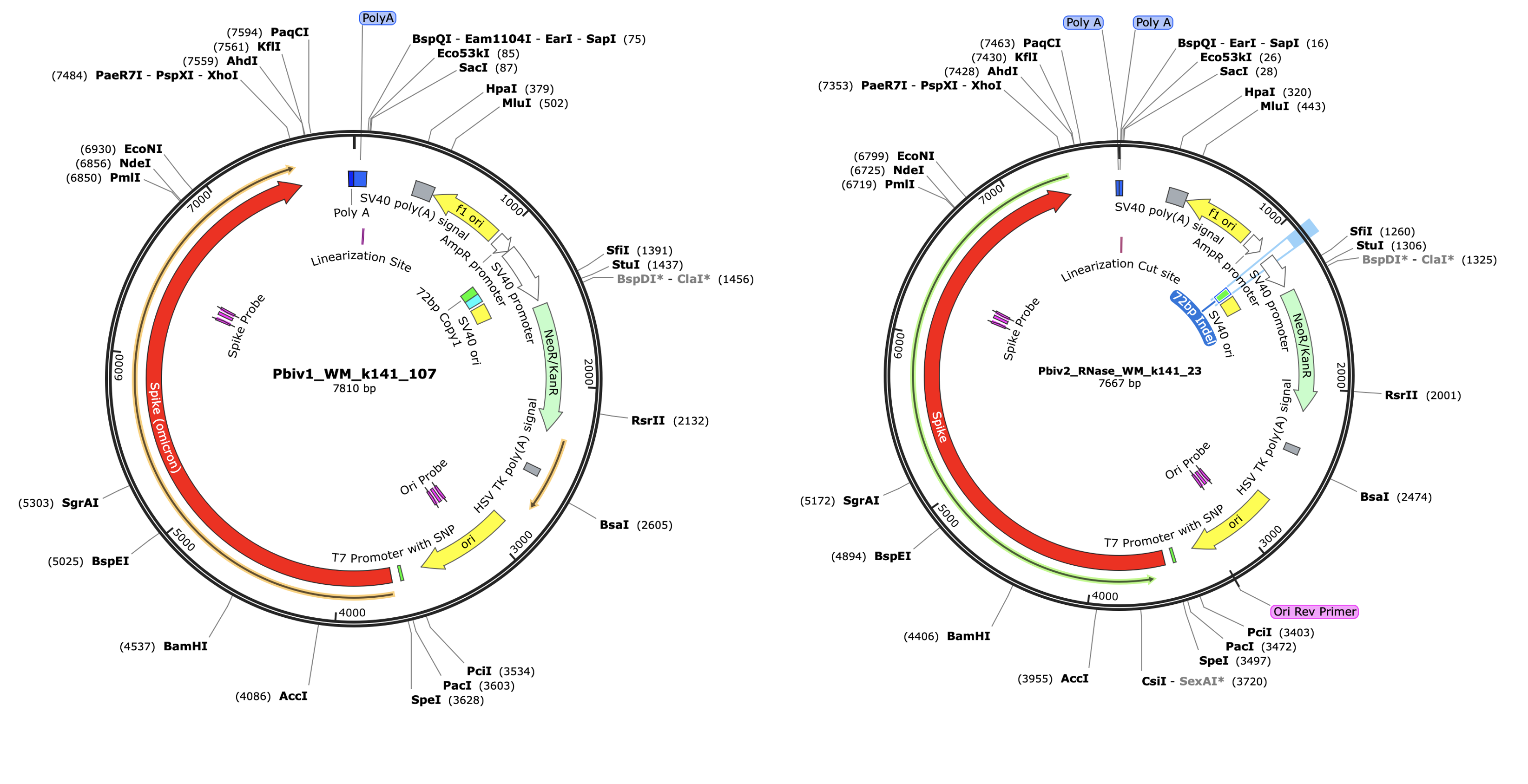
図3 RNase処理したワクチンをIlluminaでショットガンシーケンスした(RNA-seqではなくRNase-Seq)
バイアル1(左)とバイアル2(右)のPfizerベクターは、SV40プロモーターに72bpの違いがある(緑と水色の注釈)。qPCRアッセイは、ピンクでスパイクプローブとOriプローブとして描かれている。RNaseシーケンスにより、Eam1104iの直線化部位とPolyアデニル化配列の解像度が向上した。ベクターは、ポリAテールの長さ(おそらくシーケンシングアーチファクト)と72bpのインデルが異なる。
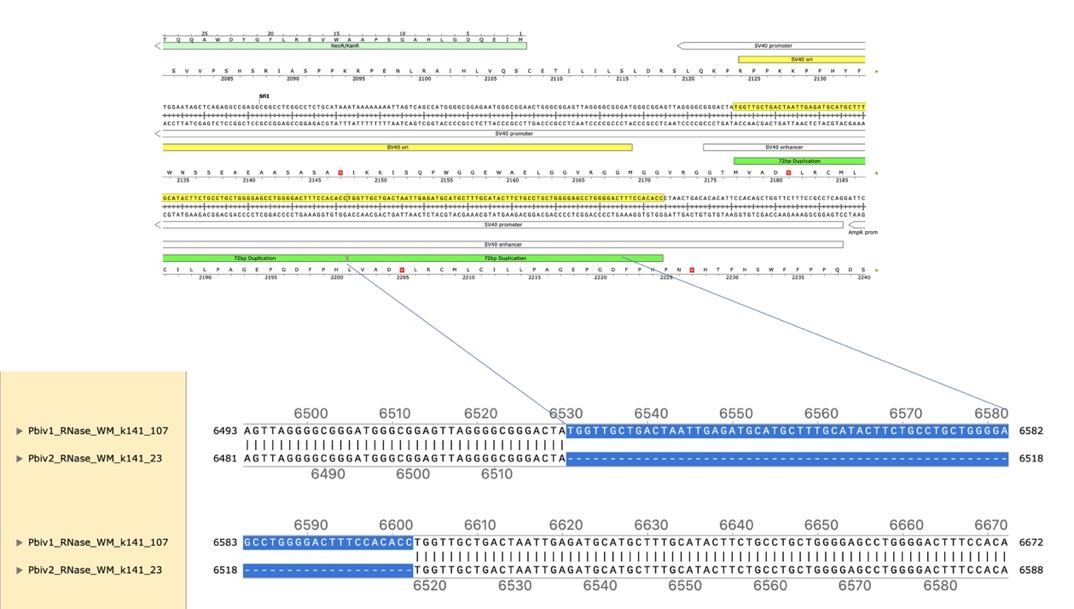
図4 Pfizer vial 1とPfizer vial 2ベクターのローカルアライメントでは、72bpのタンデム重複が青色で強調されている
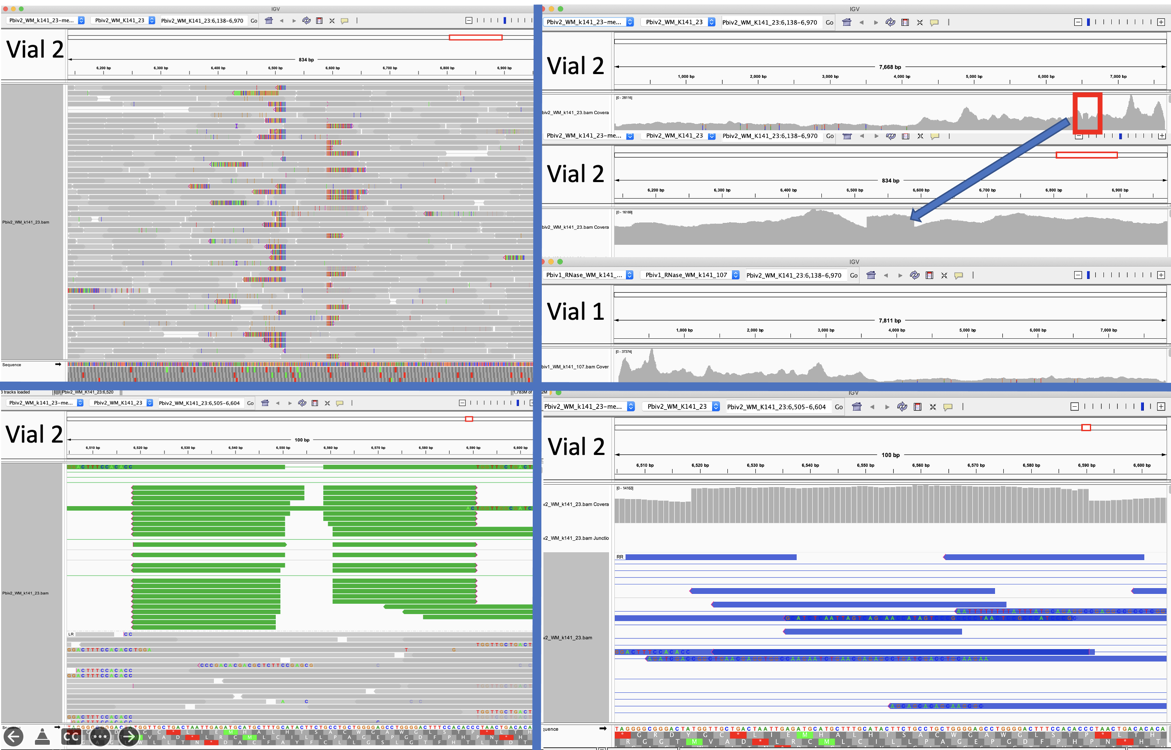
図5A. Integrative Genome Viewer(IGV)の精査により、Pfizerバイアル2において異形成である72bp挿入の出現が実証された
左上のIGVビューは、色のついたマークがインデルを描いている拡大ビューである。左下のIGVビューは、72bp挿入がタンデムリピートであり、72bpより短いペアリードは2種類の方法でマッピングできるため、反転ペアリードを示す。右上のIGVビューは、リードカバレッジの積み重ねまたは「Plateau」を示している。これは、リファレンスが72bpリピートを1コピー、サンプルが2コピー持っている場合に発生する。注-右上のIGV表示では、Vial 1の配列はVial 2とIGVで逆方向になっている。右下のIGV図は、右上のIGV画面を拡大したものである。
2つのファイザー社製バイアルは同じロット番号を共有しているため、2つのバイアル間でヘテロ接合性のコピー数変化を見つけることは予想外だった。ヘテロ接合性のコピー数変化の出現は、配列決定ライブラリーの挿入サイズが短すぎる(105bp)ため、メガヒットアセンブラが72bp配列の実際の2コピーを1コピーに折り畳んだ結果であるとの仮説が立てられた。ライブラリー中の長いペアエンドリードが72bpのタンデムリピートを解決していることは注目に値する。
リファレンスが72bpリピートの単一コピーを持ち、サンプルがリピートの2コピーを持つ場合、図5Aに見られるように、単一コピー72bp遺伝子座に対してリードが2倍のカバー率で積み上がるはずだ。この仮説を検証するために、短いプラスミドアセンブリに2番目の72bp配列を追加し、リードがアーチファクトなしにマッピングされ、ヘテロプラスミーの証拠もないことを観察した(図5B)。
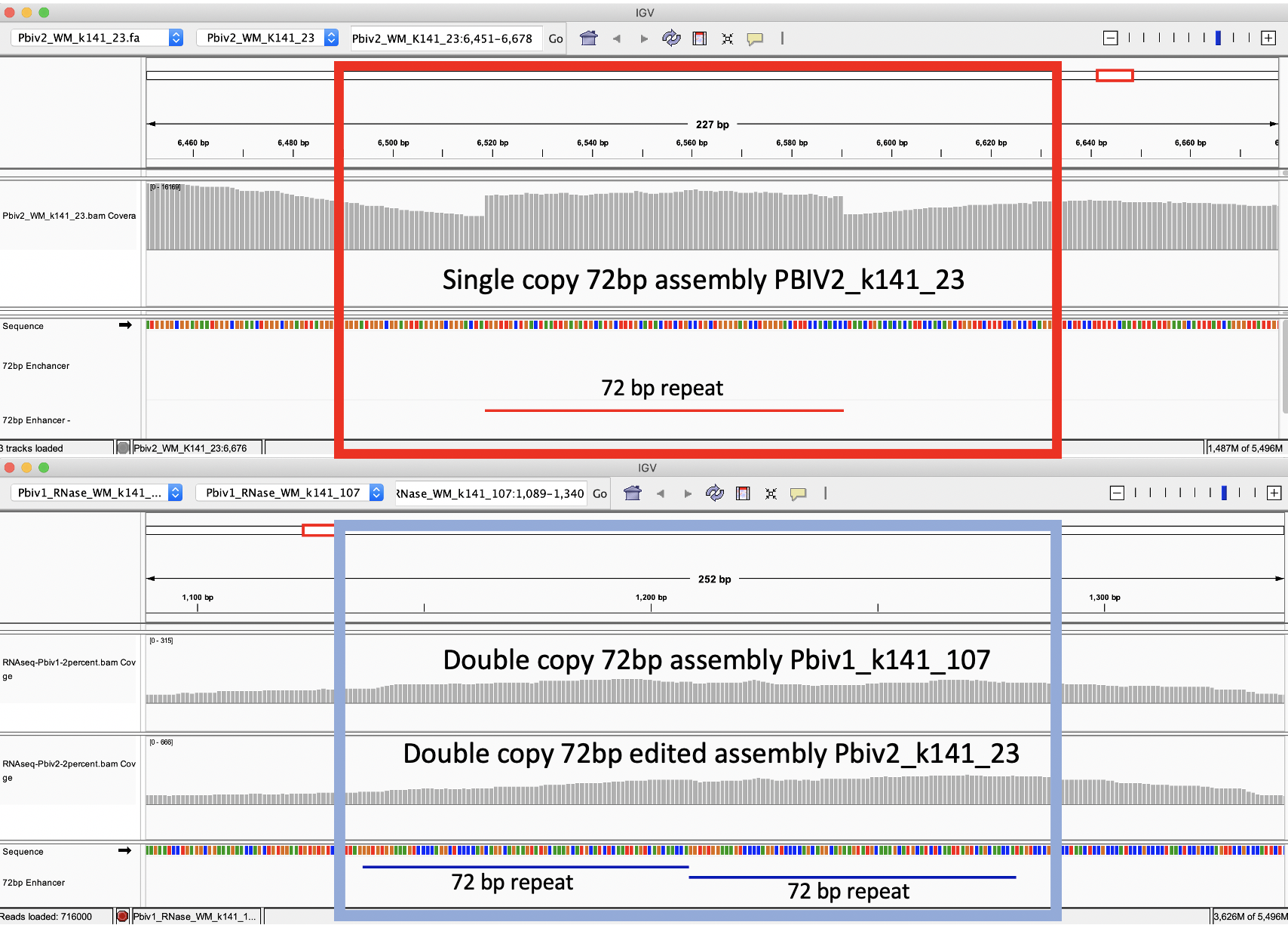
図5B. Pbiv2_k141_23上のリードカバレッジのIGVビューでは、カバレッジに離散的な72bpのプラトー(赤い四角形)がある
Pbiv2_k141_23リファレンスを編集して72bp配列の2つのコピーを含み、配列データをこの修正された配列に再マッピングすると、両方のベクター上のカバレッジはより正常で、ファイザーバイアル2にはカバレッジプラトーがないことがわかる。
これらのデータから、すべてのファイザー社製ベクターは、より強固な発現と核局在に関連する2コピーの72bp SV40 Enhancerをホモプラティックに含んでいると結論づけられた。最初のヘテロプラスチックインデルは、Megahitアセンブラとショートインサートライブラリのアーチファクトであった。
DNAのサイズを推定するため、精製ワクチンをAgilent Tape Station™ で、DNA(ゲノム DNA スクリーンテープ)および RNA ベース(高感度 RNA テープ)電気泳動テープを使用して評価した。
Agilent Tape Station™ 電気泳動では、各 300µl サンプルから検出された。23.7 -55.9ng/µlのmRNAと比較して、7.5 – 11.3 ng/µlのdsDNAが検出された。Qubit™ 3蛍光測定では、1~2.8ng/μlのDNAと21.8ng~52.8ng/μlのRNAが検出された。DNAの電気泳動では、より高いフラグメンテーションが確認された。総RNA量は、予想される30ug(100ng/μl)および100ug(200ng/μl)量より少なく、DNAおよびRNA分離における収量の損失、製造上のばらつき、または期限切れロットによるRNAの減衰を示唆している。
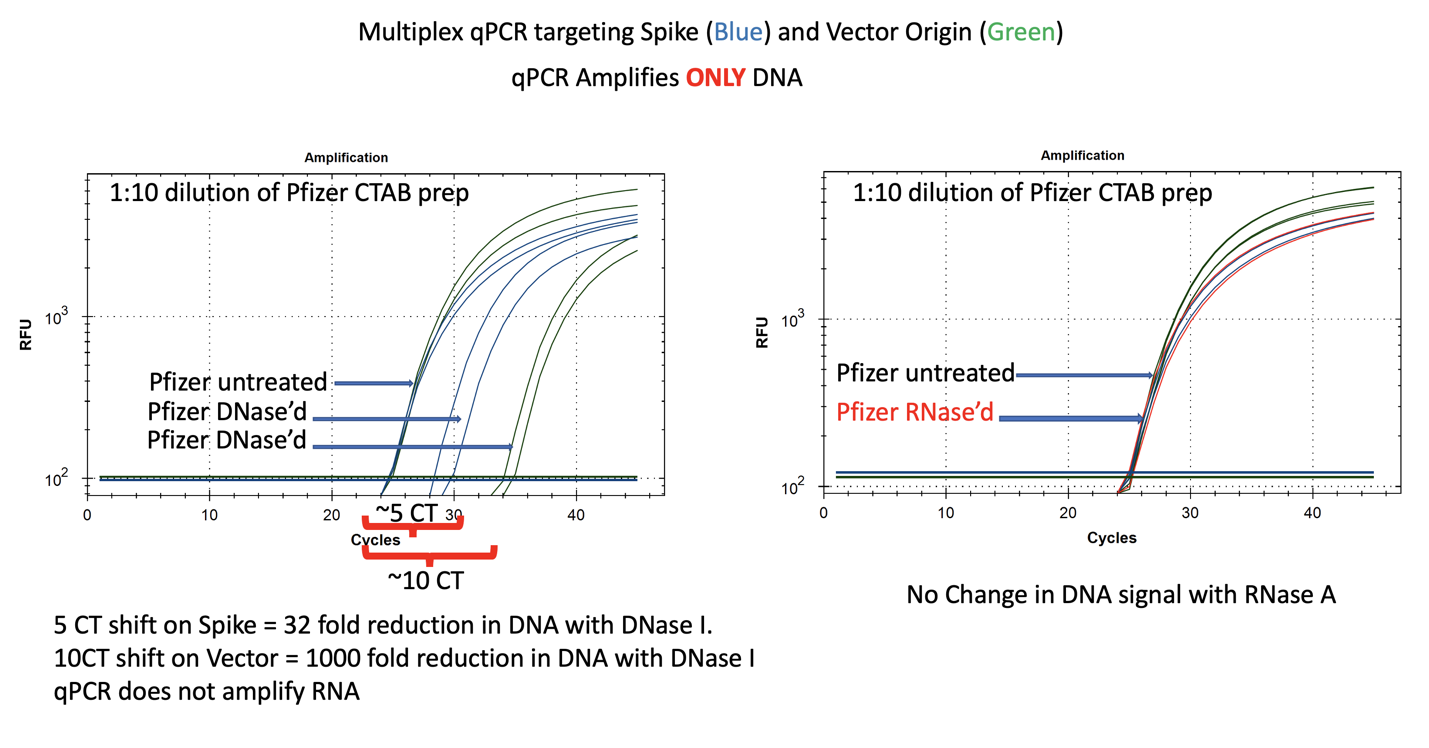
図6 Agilent Tape Station™ 電気泳動により、23.7ng/µl ~ 55.9ng/µlのRNAが確認された(左)
DNA ベースのTape Station™ では 7.5ng-11.3ng/µlが観察された。DNAエレクトロフェログラムでは全長プラスミドを示唆するピークを示すが、このサンプルはN1-methylpseudouridine RNAが多量に存在することが知られている。N1-methylpseudouridine mRNAを含むDNAハイブリッドは、ピークを生成するのに十分なインターカレート色素のクロストークを提供するかもしれない。左のRNAテープのピークの大きさは、予想より短いである。これは、N1メチルシュードウリジンがDNAの二次構造または質量電荷比を変化させた結果であると考えられる。
定量的PCRアッセイは、IDT社のPrimer Questソフトウェアを使って、モデルナとファイザーのスパイク配列とベクターの複製起点の共有配列の間で同一であるスパイクプロテインの領域をターゲットに設計された。qPCRはDNAのみを増幅するのに対し、RT-qPCRはDNAとRNAの両方を増幅するため、ワクチンのqPCRおよびRT-qPCR評価が可能になった。RT-qPCRとPCRの両方で同じサイクリング条件下で両方のターゲットが機能する条件を探るために、グラジエントqPCRが利用された(グラジエントPCRのデータは示されていない)。

図7 DNase I(左)およびRNase A(右)を使用した場合と使用しない場合のファイザー社製二価ワクチンのqPCR
未処理のmRNAは、予想通りスパイクおよびVectorアッセイで同等のCTを示す。ベクターはスパイクよりもDNase Iに敏感であることから、modRNAが相補的なDNAターゲットに対するDNase Iのヌクレアーゼ活性を阻害している可能性が示唆される。RNase A処理では、qPCRシグナルは変化しなかった。
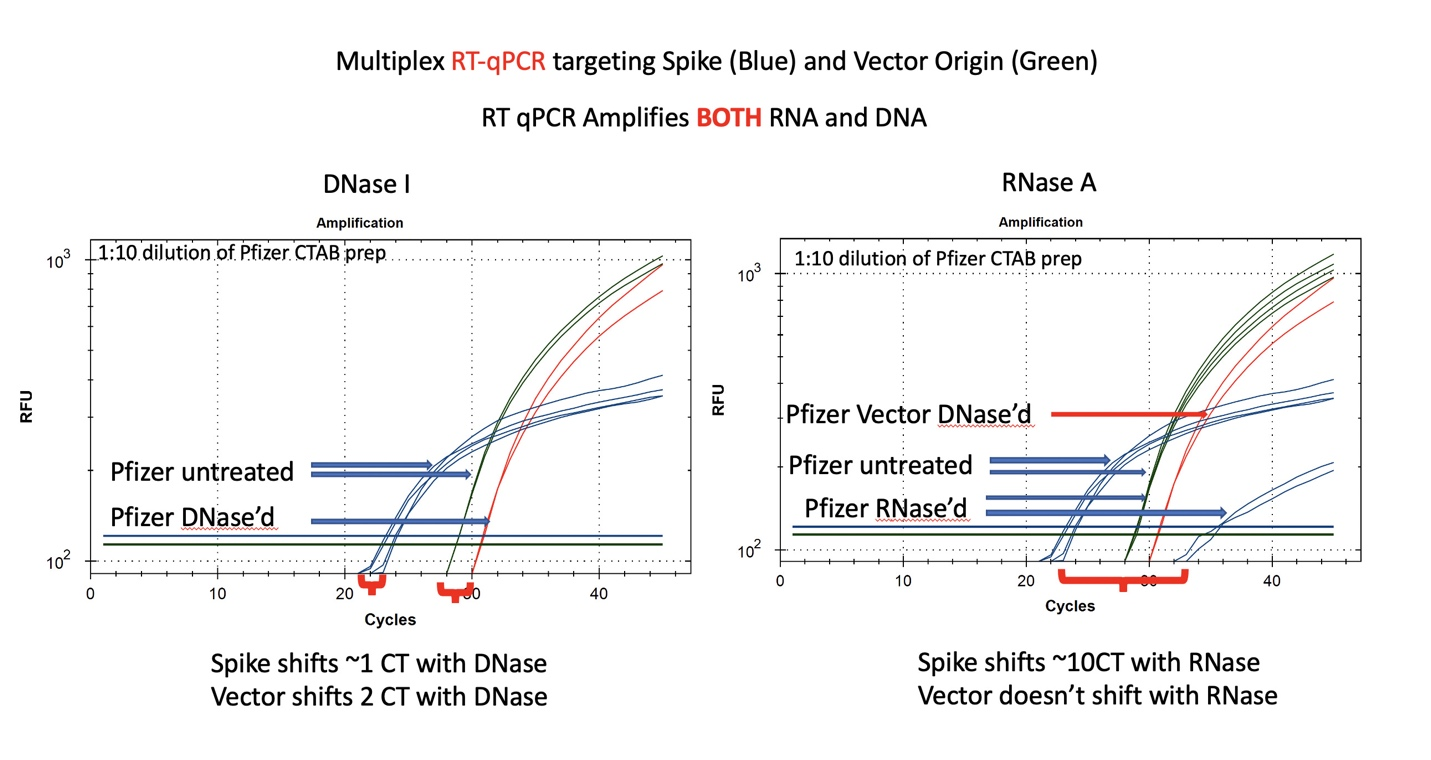
図8 RT-qPCRは、DNAとRNAの両方を増幅する
未処理のサンプルは、Pfizer スパイクおよびVectorアッセイで大きなCTオフセットを示している(左青対緑)。これは、T7重合によってベクターよりもスパイクの方がより多くのmRNAが生成されるはずなので、予想されることである。DNase I処理では、1-2個の小さなCTシフトが見られる。これは、RT-PCRにおいて、DNAが等濃度の核酸よりも少ない場合に予想されることである。RNase処理(右)は10CTのオフセットを示すが、DNAベクターのCTは変化しない。
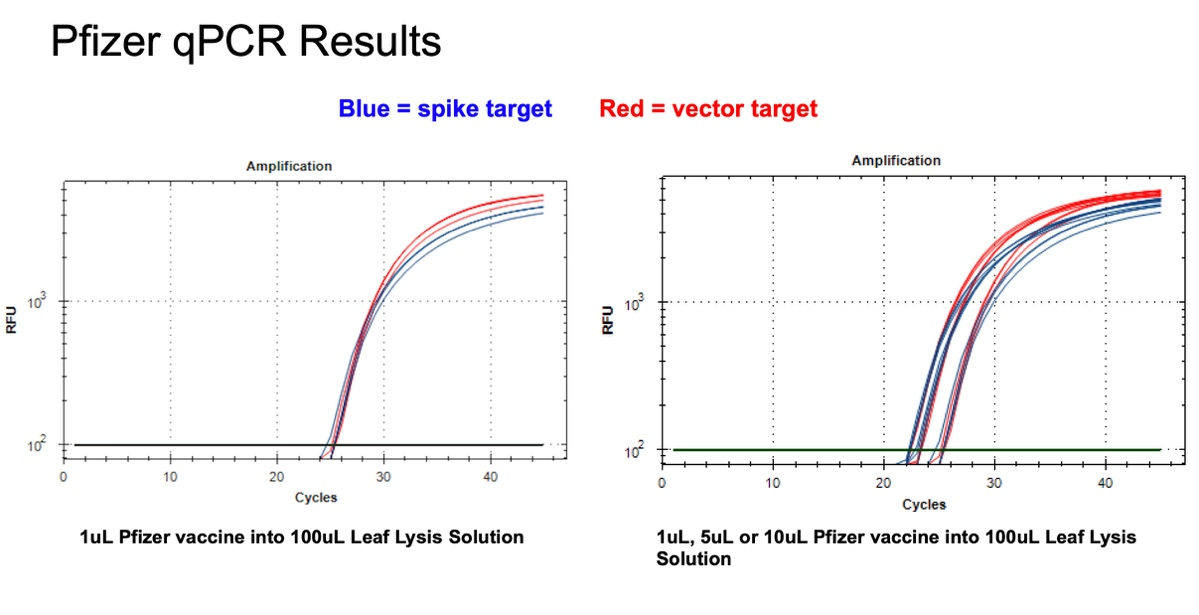
図9 1µlのPfizer二価ワクチンを100µlのLeaf Lysisバッファーに入れ、8分間の煮沸工程を行うと、qPCRのベクターおよびスパイクターゲットともに24のCTが得られる(左)
アッセイは1,5,10µlの入力に反応する(右)。
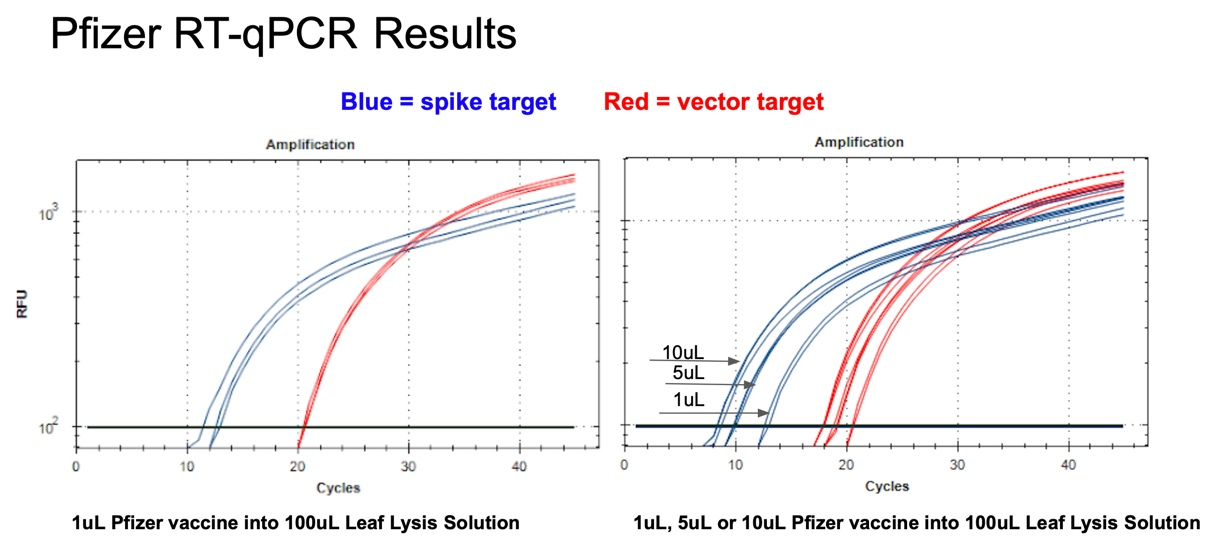
図10.
1µlのPfizer二価ワクチンを100µlのLeaf Lysisバッファーに入れ、8分間のボイルステップを行うと、RT-qPCRのVectorターゲットとSpikeターゲットの両方で20と12のCTが得られる(左)。アッセイは1,5,10µlの入力に反応する(右)。
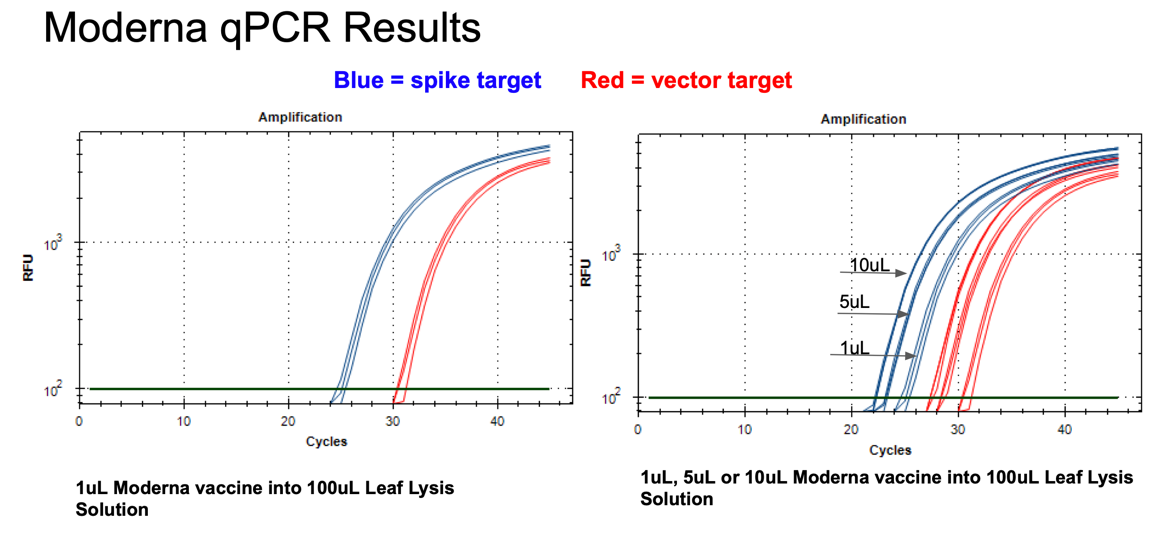
図11 モデルナ2価ワクチン1µlは、qPCRによりスパイクターゲットとベクターターゲットで異なるCTs値を示した(左)
Pfizers社のワクチンでは、アッセイで同等のCTスコアが得られ、アンプリコンの配列は2つのベクター起源の間で同一であるため、この点をさらに調査する必要がある。モデルナとファイザーのスパイクアンプリコンには2つのミスマッチがあるが、いずれのミスマッチもプライマーやプローブの下にはない。このアッセイは、1,5,10µlの直接沸騰mRNAに反応する(右)。
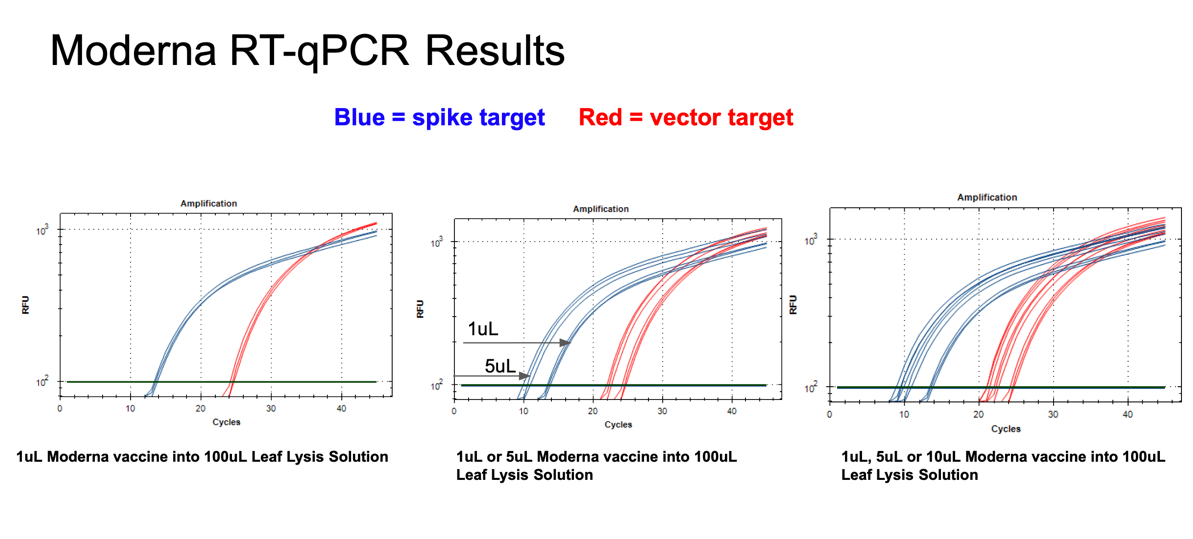
図12 モデルナ二価ワクチンの1µlは、RT-qPCRでスパイクとベクターターゲット(左)に対して異なるCT値を示した
スパイクとベクターの間の10CTの大きなシフトは、qPCRコントロールが5CTのオフセットを示していることを考慮する必要がある。ボイルプリップは、1~10µlのワクチンに耐えることができる(中・右)。
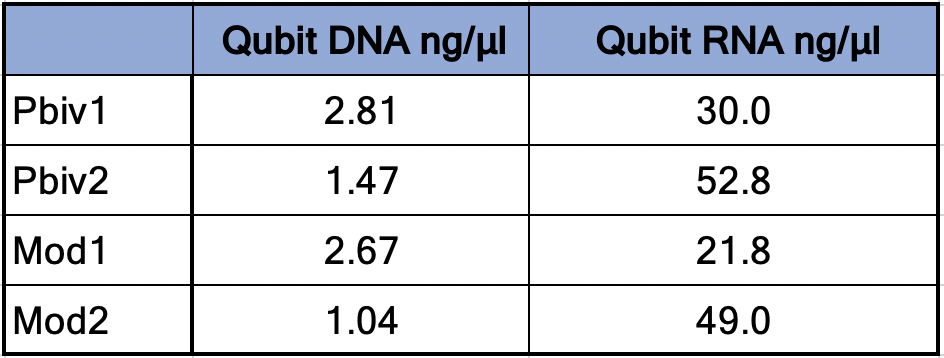
表1 Qubit™ 3 Fluorometryは、ワクチン中のdsDNAを1.04~2.8 ng/µl、RNAを21.8ng~52.8ng/µlと推定
反応中のDNAの質量に対してCTをベンチマークするためのRT-qPCR標準曲線を構築するために、IDTで合成テンプレートを合成した。この方法は理想的なテンプレートを使用し、アンプリコンサイズより小さいDNA分子を定量化することができない。予想通り、この方法では、Qubit™ 3 フルオロメトリーやAgilent Tape Station™ よりも低い。DNA 濃度の推定値が得られる。また、この方法は理想的な環境を表しており、DNA ターゲットと同一の配列を持つ大量のmRNAが、qPCR アッセイに共存している場合に発生し得る阻害やプライマーの枯渇を捉えることはできない。
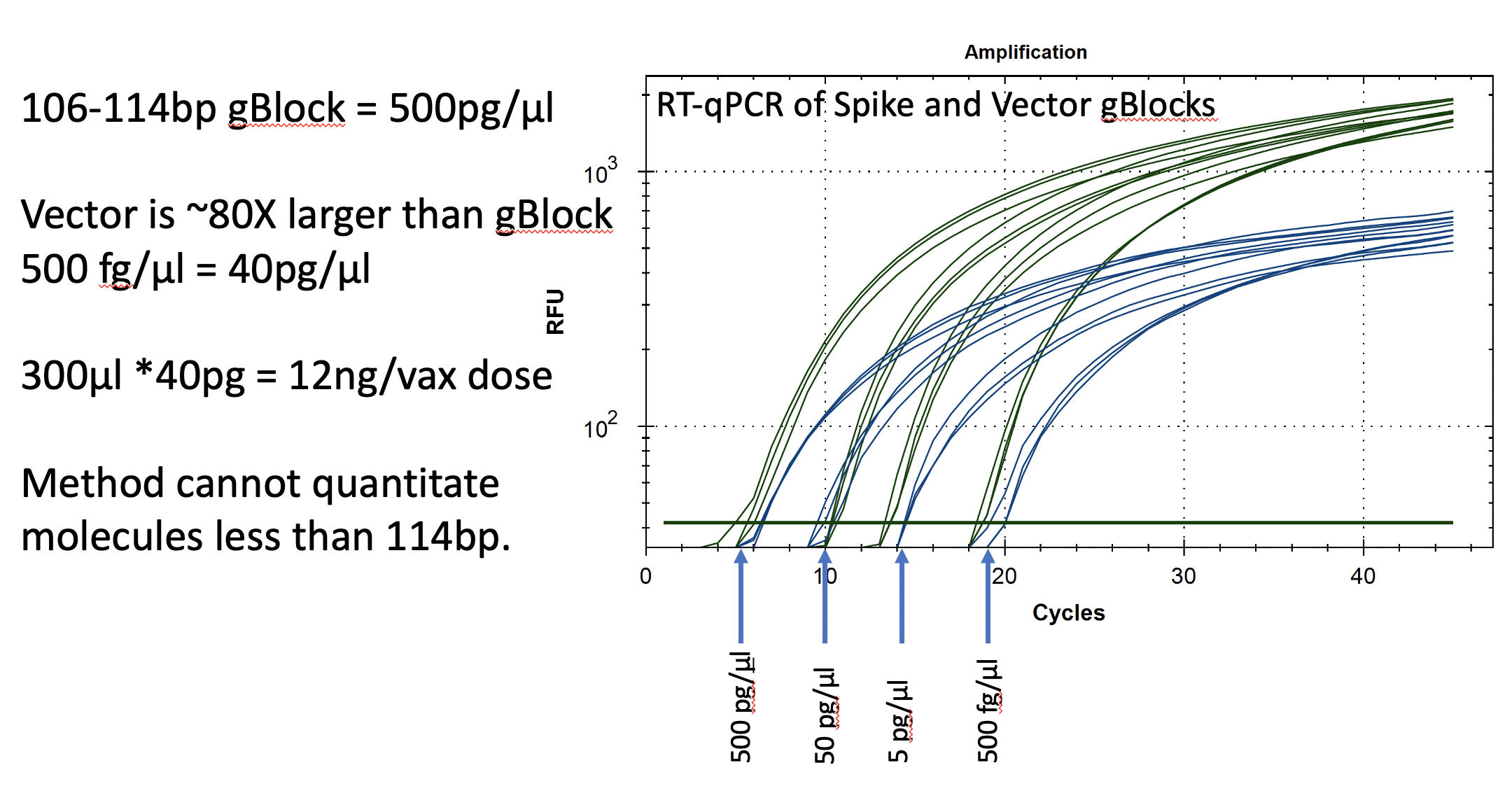
図13 RT-qPCRアッセイで使用するスパイクおよびOriポジティブコントロールテンプレートとして、IDTで2つのgBlockを合成した
10倍連続希釈を3回行い、CTスコアとDNAのピコグラムを相関させた。バックグラウンドの見直しのため、閾値は102から下げている。CTが~20=500fg/RT-qPCR反応。100bpのターゲットは、潜在的な汚染物質として存在するベクターDNAの1/80に過ぎないので、500fg/μlは40pg/μlのベクターDNAに相当する。DNase I処理され、アンプリコンサイズより小さいDNAは、本方法では増幅も定量もできない。このメソッドは、Qubit™ 3やAgilent Tape Station™と比較して、DNase I 処理されたサンプルの定量性が低くなる。
この研究は、qPCRとRT-qPCRの両方で未開封のファイザー一価ワクチン 8 種をテストすることでさらに検証された。
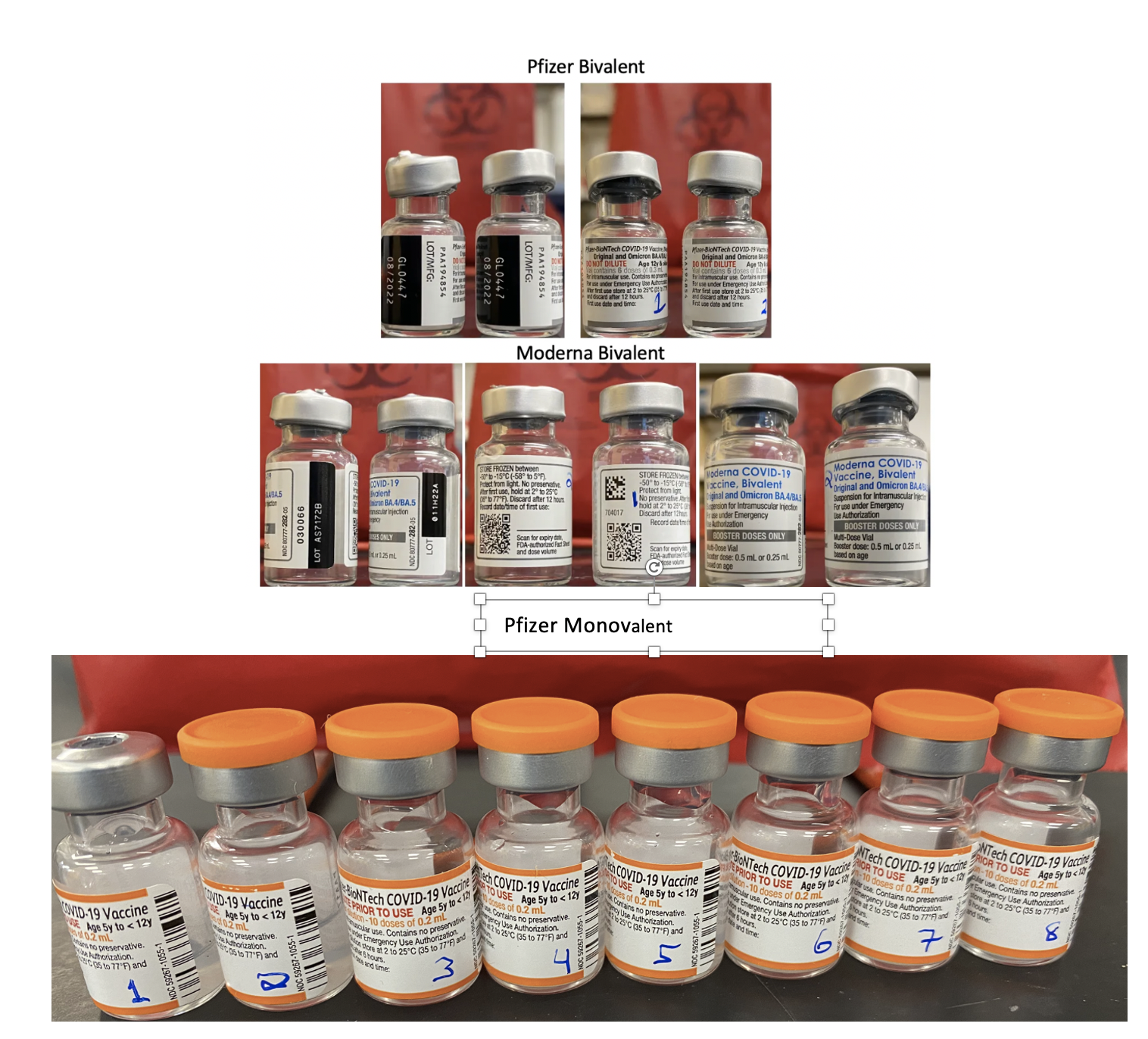
図 14. モデルナとファイザーの二価ワクチン(上)。ファイザー社製一価mRNAワクチン8種。未開封だが有効期限を過ぎたもの(下)。
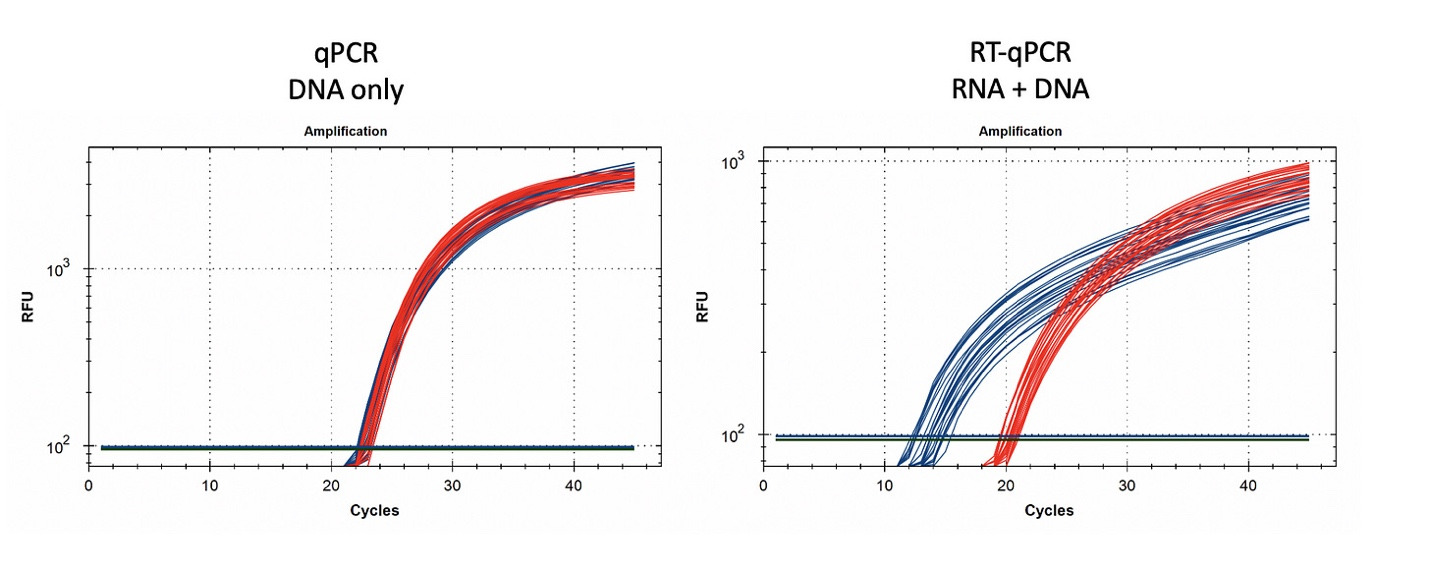
図15 1µlのワクチンを100µlのLeaf Lysis bufferで煮沸し、ベクター(赤)およびスパイク(青)についてqPCR(左)およびRT-qPCR(右)を実施した
8サンプルを三重に試験した。
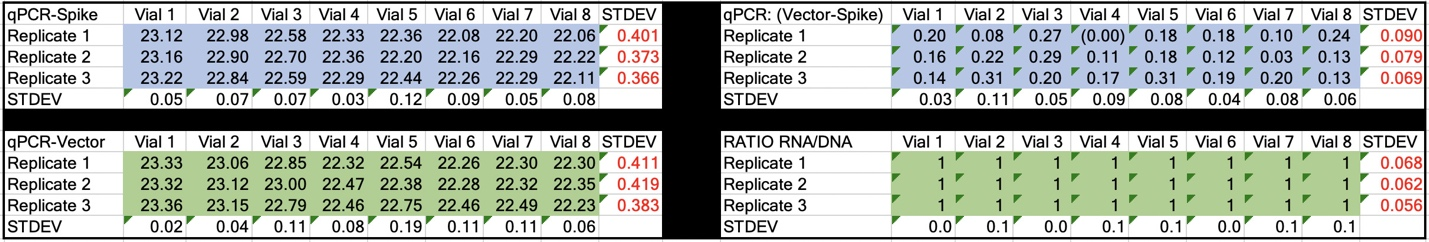
表2 スパイクとVectorのqPCR時のCT値(DNAのみ)
三重測定の標準偏差は、黒字で横書きしている。バイアル間の標準偏差は、赤色で縦に表示。デルタ CTまたは(Vector CT minus Spike CT)は、スパイクとVectorのDNAの比率を表し、1でなければならない。
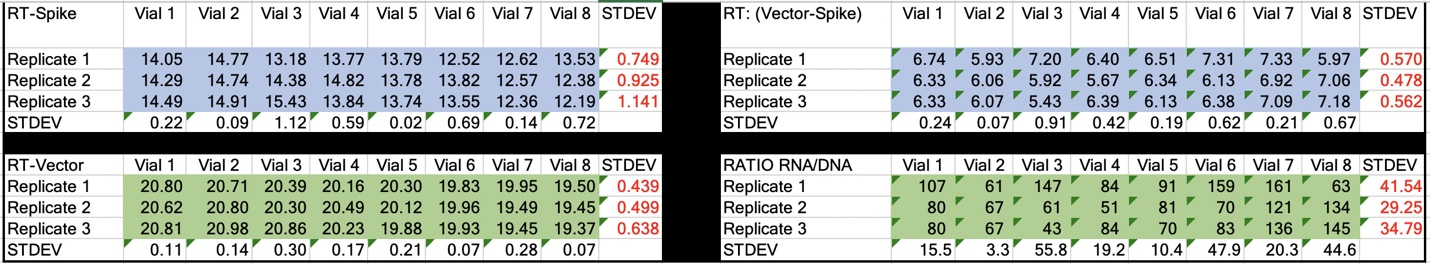
表3 RT-qPCR(RNA+DNA)時のスパイクとVectorのCT値
RNA:DNAの比率は43:1~161:1である。EMAの許容値は3030:1である。これは、EMAの許容値を18~70倍も超えている。
考察
一価ワクチンと二価ワクチンの両方において、複数の方法で高レベルのDNA混入が確認された。Qubit™ 3とAgilent Tape Station™の絶対定量は異なるが、どちらの方法も、EMAの制限値である。330ng DNA/1mg RNA よりも桁違いに高いことを示している。qPCRとRT-qPCR は、RNAとDNAの相対比率を確認している。スパイクとベクターのRT-qPCRシグナルの間には、1:3030の汚染限界(2^11.6 = 3100)を表す11-12 CTオフセットが見られるはずだ。しかし、これらのワクチンでqPCRおよびRT-qPCRデータを見ると、CTオフセットがはるかに小さい(5~7CT)ことがわかる。Qubit™ 3とAgilentの方法は溶液中のすべてのDNAを染色するが、qPCRはプライマー間にDNase Iカットサイトを持たない増幅可能な分子のみを測定することに注意する必要がある。qPCRのプライマーを離すほど、Qubit™ 3とAgilentの検出可能な分子の増幅は少なくなる。この研究で使用したプライマーは 106bpと114bpの間隔であるため、この長さ以下で、DNase I カットされた分子は、Qubit™ 3やAgilent Tape Station™による一般的なdsDNA 測定と比較して qPCR 法では過小評価されることになる。
このことは、100%インタクトな合成 DNA 標準を使用した。qPCR 標準曲線がより効率的に増幅し、その結果、消化された。DNAの総汚染量が過小評価されることも示唆している。例えば、106-114bpの合成テンプレートを用いた標準曲線では、CTが20以下のピコグラム領域(低いナノグラム領域ではない)となり、ライブラリの大部分が増幅可能な最小サイズより小さいことを示唆している。また、純粋な標準品には、qPCR法の競合プライマーシンクや阻害剤となりうる同一配列の修飾mRNAが高濃度で含まれていない。
あるいは、Qubit™ 3とAgilent Tape Station™ は、N1-メチルシュードウリジン RNAとのインターカレート色素のクロストークにより、DNA 定量値が高くなる可能性がある。このため、これらの分子をqPCRやRT-qPCRで各テンプレートタイプに特異的なポリメラーゼを用いてより注意深く調べたときに観察された比率が、より適切な指標であると考えられる。EMAの指標もこのような比率で記載されている。
また、このEMAの基準値が、DNA汚染物質の性質を考慮したものだろうかどうかも焦点となる。レプリケーションコンピテントDNAは、間違いなく、より厳しい制限を受けるはずだ。哺乳類プロモーターや抗生物質耐性遺伝子を持つDNAは、プラスミド調製から得られるランダムなバックグラウンド大腸菌ゲノムDNAよりも懸念されるかもしれない(Sheng-Fowler et al. 2009)。バックグラウンドの大腸菌DNAはqPCRで測定され、35以上のCTを有していた。
DNAの発がん性と感染性についての研究により、感度の高い生体内試験や試験管内試験のシステムで両活性が検出されるレベルは、これまで実証されてきたよりも低いことが明らかになった。従って、残存する細胞基質DNAに関連する潜在的なリスクに関して、両者を考慮する必要がある。がん原性活性と感染性活性は感度の高いアッセイから得られたものであり、クロマチンの存在など他の要因を考慮に入れていない。(Sheng-Fowler et al. 2009)
SAR-CoV-2がヒトゲノムに統合する能力について健全な議論が行われてきた(Zhang et al. 2021)。この研究は、mRNAワクチンがゲノム統合する能力についても疑問を抱かせた。このような場合、Aldenら(Alden et al. 2022)によって説明されたように、LINE-1によるmRNAのDNAへの逆転写が必要になる。スパイクプロテインをコードする配列のdsDNA汚染は、逆転写にLINE-1を必要とせず、ファイザーのワクチンベクターにSV40核局在シグナルがあることは、さらに統合の確率を高めると考えられる。この研究はゲノム統合の証拠を示すものではないが、これらのワクチンに含まれるdsDNAレベルを考慮すると、LINE-1活性は必要ないことを明確に示している。これらのベクターの核内局在性についても検証する必要がある。
Jeongらによる一価ワクチンの先行シーケンスでは、コンセンサス配列のみが公開された(Dae-Eun Jeong 2021)。このプロジェクトの生リードが利用できないため、ベクター配列の存在を精査する必要がある。
これらのワクチンがQubit™ 3とアジレントのデータでEMAの限界値(330ng/mg DNA/RNA)を超え、これらのデータもより保守的なqPCR標準曲線でFDAの限界値(10ng/投与)を超えることから、リポポリサッカライド(LPS)レベルを再検討する必要がある。大腸菌の前処理によるプラスミド汚染は、しばしばLPSと共汚染される。内毒素の汚染は、注射時にアナフィラキシーを引き起こす可能性がある(Zheng et al. 2021)。
この研究の限界は、研究対象のワクチンバイアルの出所が不明であることである。これらのバイアルは、保冷剤なしで匿名で郵便で送られてきたものである。RNAはDNAよりも早く劣化することが知られており、保存状態が悪いとDNAよりもRNAの方が早く劣化する可能性がある。分子としてのRNAは非常に安定しているが、金属や熱、あるいは背景にユビキタスなRNaseが存在すると、非常に速く分解される。この研究で使用されたワクチンはすべてバイアルに記載された有効期限を過ぎており、新鮮なロットでのDNAとRNAの比率を理解するためにさらなる研究が必要であることが示唆される。このqPCRプライマーが発表されたことで、より管理されたサプライチェーンにおける追加ロットの調査に役立てることができるかもしれない。母乳や血漿中のワクチンの寿命を評価する研究では、ベクターDNAの調査は有益であると考えられる。
配列決定によりプラスミドバックボーンは完全に網羅されたが、DNase Iで断片化したライブラリーからプラスミドを組み立てるのが通例だ。これらの方法では、バイアル内の直鎖状DNAと環状DNAの比率を識別することができなかった。プラスミドDNAはより有能で安定である一方、線状DNAはゲノム統合リスクが高い可能性がある。
Qubit™ 3およびAgilentシステムで使用されているインターカレート色素は、DNAおよびRNAとの蛍光クロストークが少ないことが知られているが、N1-methylpseudouridineがこれらのインターカレート色素の特異性をどの程度変化させるかは分かっていない。その結果、EMAの比メトリック制御の最良の相対評価として、ベクターとスパイク配列を用いたRT-qPCRとqPCRの間のCTオフセットに依存していた。これらのqPCRおよびRT-qPCR試薬は、将来、ワクチン、血液バンクまたは患者組織におけるこれらの汚染物質の追跡に役立つと考えられる。
方法
LNPからのmRNAの精製
LiDs/SPRI精製
各バイアルから100μlをサンプリング(1/3~1/5量)した
- 100µlのワクチンに5µlの2%LiDsを加え、LNPsを溶解させた
- 100%イソプロパノール100µl
- 233µlのAmpure(Beckman Genomics社製)
- 25mM MgCl2(New England Biolabs)25μlを入れる
サンプルを10倍にチップミキシングし、磁気ビーズ結合のために5分間インキュベートした。磁気ビーズを96ウェルマグネットプレート上で10分間分離し、200µlの80% EtOHで2回洗浄した。ビーズを3分間風乾させ、100µlのddH20で溶出した。溶出したサンプル2µlをAgilent Tape Station™で実行した。
ワクチンのCTAB/クロロホルム/SPRI精製
LiDs/SPRIによるワクチンの精製方法によって、qPCR性能に若干のばらつきが認められた。このため、一部のサンプルは不透明で、精製中にLNPが残留している可能性がある。この問題に対処するため、CTAB/クロロホルム/SPRIによる分離が最適化され、さらなる。qPCRとAgilent 電気泳動に使用された。簡単に説明すると、300μlのワクチンを500μlのCTAB(SenSATIVAx MIP精製キットのMGC溶液A、#4 20004)に加えた。その後、サンプルをボルテックスし、37℃で5分間加熱した。800µlのクロロホルムを加え、ボルテックスし、19,000rpmで3分間回転させた。上部の250µlの水相を集め、250µlの溶液Bと1mlの磁気結合バッファーに加えた。サンプルをボルテックスし、5分間インキュベートし、磁気的に分離した。上清を除去し、ビーズを70%エタノールで2回洗浄した。サンプルは最後に300μlのMGC溶出バッファーで溶出した。
ワクチンqPCRを評価するための簡単なボイル準備。
この煮沸調製は、ワクチンを1~10µl採取し、PCR対応のリーフライシスバッファーに希釈して加熱するだけです(Medicinal Genomics品番420208)。
- 65℃、6分間
- 95℃、2分間
シーケンシングのためのライブラリー構築
100μlの各サンプルから50μlを、NEB NEBNext UltraII Directional RNA library Kit for Illumina(NEB#E7760S)を用いてイルミナシーケンス用のRNA-Seqライブラリーに変換した。
長いインサートライブラリーを充実させるため、断片化時間を15分から10分に短縮し、プロトコルの長いインサートの推奨に従って、一本鎖合成時間を42℃で50分に延長した。
Riboの枯渇やPolyAの濃縮は行わず、ライブラリ内の全フラグメントを最も公平に評価できるようにした。製造元のプロトコルに従って、16サイクルの増幅を行った。mRNAの一本鎖の性質を評価するために、指向性ライブラリー構築法を使用した。dsRNA(0.5%以上)は自然免疫反応を引き起こす可能性があるため、これはEMAおよびTGAの開示文書において重要な品質指標である。dsRNA含有量は、しばしばELISAを用いて推定される。方向性 DNA シークエンシングは、より包括的な推定方法であり、以前 Jeong らにより測定され、99.99%であった。
ワクチンのRNase A処理
RNase Aはウラシルとシトシンの両方を切断する。N1-メチルシュードウリジンはRNAse-L耐性であることが知られているが、RNase AはmRNA中にまだ存在するシトシンを切断することができる。これにより、塩基配列を決定するためのDNAが優勢になる。以前に配列決定し、ここで議論したワクチンmRNAを、NEB社の20Units/μl Monarch RNase A 10μlで37℃、30分間処理した。RNase反応は、1.5倍のSenSATIVAx(Medicinal Genomics #4 20001)を用いて精製した。DNA精製後、サンプルを20µl ddH20で溶出した。15µlをDNAシークエンスに使用した。
ワクチンのDNase処理
50µlのCTAB精製ワクチンを2µlのDNase Iと6µlのDNase I buffer(Grim reefer MGC#4 20143)で37℃、30分間処理した。2.5µlのLiDs Lysis bufferを加え、DNase反応を停止させた。60μlの100%イソプロパノール、140μlのAmpure、15μlのMgCl2を用いて反応液を精製した。磁気ビーズを10回チップミキシングし、5分間放置してインキュベートした後、磁気的に分離し、80% EtOHで2回洗浄した。
RNaseを用いたワクチンの全ゲノムショットガン。
15μlのDNAをWatchmakers Genomics WGS library construction kitを使用してシーケンスレディライブラリーに変換した。このキットはDNAをさらに小さく断片化するため、ワクチン中の断片長を予測することが困難になる。
Qubit™ 3 Fluorometry
Biotum AccuBlue RNA Broad Range kit (#3 1073)とBiotum AccuGreen High Sensitivity dsDNA Quantitation Kit (#3 1066)を用いて、Qubit™ 3蛍光測定をメーカーの指示に従い実施した。
大腸菌qPCR
Medicinal Genomics PathoSEEK™ E.coli Detection assay (#4 20102)は、メーカーの説明書に従って使用した。
qPCRおよびRT-qPCRスパイクアッセイ
- MedGen-モデルナ_Pfizer_Janssen_Vax-Spike_Forwardの略
- アガットグックタックグットカ
- MedGen-モデルナ_Pfizer_Janssen_Vax-Spike_Reverseの略
- TCAGGCTCTGATCTT(トカグクトクトカクトガットタクト)
- MedGen-モデルナ_Pfizer_Janssen_Vax-Spike_Probeを使用
- >/56-FAM/CGAGAACCA/ZEN/GAAGCTGATCGCCAA/3IABkFQ/ である
qPCRおよびRT-qPCRベクターオリジンアッセイ
- メドジェン_ワクチン接種ベクター_オリ_フォワード
- ctacatacctcctgctgctaatcとする
- メドジェン_バックスベクター_オリ_リバース
- gcgccttatccggtaactatc
- MedGen_Vax-vector_Ori_Probe(メドジェン・バックスベクター・オリ・プローブ
- 5HEX/AAGACACGA/ZEN/CTTATCGCCACTGGC/3IABkFQ/である
IDTの説明書に従ってプライマーを100uMに溶出する。
50Xプライマー・プローブミックスを作る。
- 1. 25μl 100uM Forwardプライマー
- 2. 25μl 100uM リバースプライマー
- 3. 12.5µl 100uM プローブ
- 4. 37.5µl ヌクレアーゼフリーddH20
この混合液の15µlを、以下に示すqPCRマスターミックスのセットアップに使用する。(1回の反応につき0.5µlのプライマー/プローブを使用)
この混合液10µlを以下に示すRT-qPCRマスターミックスのセットアップに使用する。
使用したMedicinal Genomicsマスターミックスキット
- 1. store.medicinalgenomics.com/qPCR-Master-Kit-v3-200-rxns
- 2. store.medicinalgenomics.com/pathoseek-rt-qpcr-master-kit
30反応分のqPCRの反応設定
- 114µl エンザイムミックス(グリーンチューブ)
- 24µl Reaction Buffer(青色チューブ)
- 246µl ヌクレアーゼフリーddH20
- プライマー・プローブセット・スパイク15µl
- プライマー・プローブセットOri 15µl
上記マスターミックス13.8µlと精製サンプル5µl(ワクチンDNA/RNA 1µl + CT<15の場合はddH20 4µl)を使用する。
RT-qPCRを34反応させるための反応設定
- 200µl エンザイムミックス
- 96µl ヌクレアーゼフリーddH20
- 20µl RNase Inhibitor(紫色のチューブ)
- 4µl DTT(緑色のチューブ)
- 10µl プライマー・プローブセットスパイク
- 10µl プライマー・プローブセット Ori
マスターミックス10µlとワクチンDNA/RNA 1µl
メディシナルゲノミクス社製MIP DNA精製キットを使用
CTAB/クロロホルム/SPRIを用いたDNA/RNAの分離方法は前述の通りである。
サイクリング条件
これらの条件は、qPCRとRT-qPCRの両方に有効である。注:qPCRでは50℃のRTステップを省略することができる。使用したMGC qPCR MasterMixキットは、この50℃のステップに影響されないホットスタート酵素を有している。RNAとDNAの比較をコントロールするために、qPCRとRT-qPCRアッセイを同じプレートに置き、すべてのサンプルでRTステップを含む以下のプログラムを実行した。
qPCRおよびRT-qPCRに使用したサイクル条件
gBlock Positive Controlsのアンプリコンのシークエンス。Ori = 106bp, Spike = 114bp.
Oriターゲット
スパイクターゲット
塩基配列データ
イルミナリードRNA-seq生データ
- ファイザー Bivalent Vial 1 フォワードリード
- ファイザー Bivalent Vial 1 逆方向の読み方
- ファイザー Bivalent Vial 2 フォワードリード
- ファイザー Bivalent Vial 2 逆方向の読み方
- モデルナ Vial 1 フォワードリード
- モデルナ Vial 1 逆読み
- モデルナバイアル2 フォワードリード
- モデルナバイアル2逆読み
読み込まれたファイルはsha256(Hash and stash)にかけられ、DASHブロックチェーンに刻まれる。読み込まれたファイルのsha256ハッシュは、不変の台帳のOP_RETURNに費やされる。ファイルのハッシュがこれらのトランザクションのハッシュと一致しない場合、ファイルは改ざんされたことになる。
- ファイザーバイアル1 フォワードハッシュ
- ファイザーバイアル1逆ハッシュ
- ファイザーバイアル2フォワードハッシュ
- ファイザーバイアルの2逆ハッシュ
- モデルナ Vial 1 フォワードハッシュ
- モデルナ Vial 1 リバースハッシュ
- モデルナ Vial 2 フォワードハッシュ
- モデルナバイアル2 リバースハッシュ
メガヒットアッセンブリー
- ファイザーバイアル1
- ファイザーバイアル2
- モデルナバイアル1
- モデルナバイアル2
Megahitアセンブリにマッピングされたイルミナリード
- Pfizer Vial 1 BAM ファイル。インデックスファイル
- Pfizer Vial 2 BAMファイル。インデックスファイル
- モデルナ Vial 1 BAMファイル。インデックスファイル
- モデルナ Vial 2 BAMファイル。インデックスファイル
Q30 Filtered Illumina Reads(転写エラー率推定に使用する)
FastQ-Filterのダウンロード: usage> fastq-filter -e 0.001 -o output.fastq input.fastq
- Pfizer bivalent Vial 1 フォワードリード
- ファイザー2価バイアル1逆リード
- ファイザー 2価バイアル2 前方読み出し
- ファイザー2価バイアル2逆リード
- モデルナ bivalent Vial 1 Forward Readsの略
- モデルナ bivalent Vial 1 Reverse Reads(モデルナバイバレントバイアル1)
- モデルナ bivalent Vial 2 フォワードリード
- モデルナ bivalent Vial 2 リバースリード
Q30 BAMファイル Q30リードをMegahitアセンブリにマッピングしたもの
- ファイザーバイアル1 q30-BAMのファイルである。インデックスファイル
- ファイザーバイアル2 q30-BAMのファイルである。インデックスファイル
- モデルナ Vial 1 q30-BAMのファイルである。インデックスファイル
- モデルナ Vial 2 q30-BAMのファイルである。インデックスファイル
IGVtoolsのq30リードの塩基別エラーについて
フィールド=コンティグ内の位置、正立(+)A、+C、+G、+T、+N、+削除、+挿入、負立(-)A、-C、-G、-T、-N、-削除、-挿入
- モデルナバイアル1
- モデルナバイアル2
- ファイザーバイアル1
- ファイザーバイアル2
解析パイプライン
でリードをデマルチプレックスし、処理した。
- Trimgalore – イルミナシーケンスアダプターを除去する
- Megahit- リードをコンティグにアセンブルする
- SARs-CoV-2のためのMegahit
- Samtools-IGVで表示するためのBAMファイルを生成する
- Samtoolsのstatsは、outie readsの計算に使用される
- BWA-mem- ショートリードマッパーは、リードをアセンブルしたリファレンスに戻すために使用される
- SnapGene software- (www.snapgene.com) – 発現ベクターの可視化およびアノテーションに使用される
- IGV- イルミナシーケンスリードの可視化に使用するIntegrated Genome Viewer
RNase処理済みライブラリ-BAMファイル
samtoolsを使用してコンティグ固有のBAMファイルを作成した。
samtools view -h input.bam contig_name -O BAM > contig.bam; samtools index contig.bam;
各アセンブリの各コンティグに対してSamtools statsを実行する。
for out_prefix in `ls *.sort.bam | perl -pe 「s/.sort.bam//」`; do mkdir -p ${out_prefix}-samtools-stats; for contig in `samtools view -H ${out_prefix}.sort. bam | grep 「^@SQ」 | cut -f 2 | perl -pe 「s/SN://」`; do echo 「Now calculating stats for ${contig}/$out_prefix…」; samtools stats ${out_prefix}.sort.bam $contig > ${out_prefix}-samtools-stats/${contig}-samtools-stats.txt; done; done
- Pbiv1_RNase_WM_k141_107.fa(ピーブイワン_アールナーゼ_WM_k141_107)
- Pbiv1_RNase_WM_k141_107.bamの場合
- Pbiv1_RNase_WM_k141_107.bam.bai
- Pbiv2_RNase_WM_k141_23.fa
- Pbiv2_RNase_WM_k141_23.bamの場合
- Pbiv2_RNase_WM_k141_23.bam.bai
著者による寄稿
KJM-シーケンスライブラリーの構築、qPCRアッセイの設計、Qubit™ 3sとAgilent Tape Station™の実行と解析、原稿作成。
YH-DNAの分離、Tape Station™、qPCRの結果を最適化した。
SM、LTK-リードのデマルチプレックスとトリミング、アセンブリのトラブルシューティングをサポートした。
利益相反
本論文の著者は、本研究で使用したqPCRおよびDNA分離キットの一部を製造するMedicinal Genomics社の従業員である。
謝辞
Lei Zhangには、シーケンスのアウトソーシングをオーガナイズしてくれて、感謝いたします。Jessica Rose、Sabine Hazan、Jikkyleaks、Pathogenetics、Steve Massey、Valentine Bruttel、Lynn Fynn、Sasha Letypova、David Wiseman、Pharmacoconutsにはこのテーマについて参考になるコメントをいただいた。
参考文献
Alden M, Olofsson Falla F, Yang D, Barghouth M, Luan C, Rasmussen M, De Marinis Y. 2022年。Pfizer BioNTech COVID-19 mRNA Vaccine BNT162b2 In Vitro in Human Liver Cell Lineの細胞内逆転写。Curr Issues Mol Biol 44: 1115-1126.
Bansal S, Perincheri S, Fleming T, Poulson C, Tiffany B, Bremner RM, Mohanakumar T. 2021年. 最先端:COVIDスパイクプロテインを含む循環エクソソームは、抗体開発前にBNT162b2(Pfizer-BioNTech)ワクチン接種によって誘導される: mRNAワクチンによる免疫活性化の新たなメカニズム。J Immunol 207: 2405-2410.
Castruita JAS、Schneider UV、Mollerup S、Leineweber TD、Weis N、Bukh J、Pedersen MS、Westh H. 2023年. SARS-CoV-2スパイクmRNAワクチン配列は、COVID-19ワクチン接種後28日まで血中に循環する。APMIS 131: 128-132.
Dae-Eun Jeong MM, Karen Artiles, Orkan Ilbay, Andrew Fire*, Kari Nadeau, Helen Park, Brooke Betts, Scott Boyd, Ramona Hoh, and Massa Shoura*. 2021. ワクチンBNT-162b2およびmRNA-1273の推定SARS-CoV2スパイクをコードするmRNA配列のアセンブル。GitHub。
ディーンDA、ディーンBS、ミュラーS、スミスLC。1999. プラスミド核内輸入のための配列要件。Exp Cell Res 253: 713-722.
Hanna N, Heffes-Doon A, Lin X, Manzano De Mejia C, Botros B, Gurzenda E, Nayak A. 2022年。ヒト母乳中のメッセンジャーRNA COVID-19ワクチンの検出。JAMA Pediatr 176: 1268-1270.
ジョセフソンF. 2020-11-19. 報告者のローリングレビュー評価報告書。ヒト用医薬品委員会 EMEA/H/C/005735/RR.
Moreau P, Hen R, Wasylyk B, Everett R, Gaub MP, Chambon P. 1981. SV40の72塩基修復反復は、SV40および他のキメラ組換え体の両方で、遺伝子発現に顕著な影響を及ぼす。Nucleic Acids Research 9: 6047-6068.
ナンス KD、マイヤー JL. 2021. 緊急時の修飾: COVID-19 ワクチンにおける。N1-Methylpseudouridineの役割。ACS Cent Sci 7: 748-756.
Sheng-Fowler L, Lewis AM, Jr., Peden K. 2009. ウイルスワクチンにおける細胞基質DNAの残存に関連する問題。Biologicals 37: 190-195.
Ulrich-Lewis JT, Draves KE, Roe K, O’Connor MA, Clark EA, Fuller DH. 2022. STINGは、DNAワクチンによるI型ヘルパーT細胞細胞依存の抗体反応の誘導に、従来の樹状細胞で必要である。Front Immunol 13: 861710.
Zhang L, Richards A, Barrasa MI, Hughes SH, Young RA, Jaenisch R. 2021. 逆転写されたSARS-CoV-2 RNAは培養ヒト細胞のゲノムに統合でき、患者由来の組織で発現することができる。米国国立科学アカデミー紀要 118.
Zheng Q, Wang T, Zhu X, Tian X, Zhong C, Chang G, Ran G, Xie Y, Zhao B, Zhu L et al. 2021. 低エンドトキシン大腸菌株由来プラスミドは、試験管内試験および生体内試験の両方でrAAVベクターを介した免疫応答を低下させる。Mol Ther Methods Clin Dev 22: 293-303.
