Contents
Drug Repurposing: A Network based Approach to Amyotrophic Lateral Sclerosis
受理日:2021年4月2日
概要
筋萎縮性側索硬化症(ALS)は、進行性で慢性的な衰弱を伴う神経疾患であり、治療法が確立されていない希少疾患である。従来の「1ターゲット、1ドラッグ」パラダイムに固執するあまり、異質で多因子にわたる疾患に対する有効な治療法はこれまで存在しなかった。本研究は、ネットワーク医療と薬物再利用戦略から得られた新しい知見を活用し、ALS発症機序における革新的な解決策と治療パラダイムを見出すことを目的としている。このアルゴリズムは、ヒト・インタラクトーム中の疾患関連遺伝子と創薬標的の近接度を定量化するものである。SAveRUNNERによって予測された403の医薬品を、ALSとのネットワークの類似度の高い順に優先順位付けした。カテコールアミン、ドーパミン、セロトニン、ヒスタミン、GABA受容体調節薬、アンジオテンシン変換酵素、シクロオキシゲナーゼアイソザイム、セロトニントランスポーター阻害薬の中から、アモキサピン、クロミプラミン、ミアンセリン、modafinilなど、従来はなかった面白いALS治療薬を発見することができた。さらに、遺伝子セットの濃縮解析によりSAveRUNNERによる予測を強化し、スコア0以上の121の薬剤の中で最もスコアの高い薬剤としてmodafinilを確認した。 この結果は、ALSの病態における治療の革新的な解決策をさらに証明することに貢献するものである。
キー ワード Drug repositioning – Network Medicine – Amyotrophic lateral sclerosis – Modafinil – Histaminergic drugs
ハイライト
- ALSは、治療法が確立されていない希少な上部および下部運動ニューロン疾患である。
- ALSに関連する再利用可能な新薬として、modafinil、amoxapine、clomipramine、mianserinを特定し、SaveRunnerネットワークベースのアルゴリズムで優先順位をつけた。
- SAveRUNNERによって予測された再利用可能な薬剤は、遺伝子セット濃縮分析(GSEA)によってさらに調査される。
- SAveRUNNER/GSEAアルゴリズムは、ALS治療薬の同定と将来の臨床評価のための統合的なフレームワークを導入している。
はじめに
筋萎縮性側索硬化症(ALS)は、随意筋の制御と運動を障害する稀な進行性かつ慢性の衰弱性運動ニューロン疾患であり、過去200年以上にわたって科学者と臨床医の注目を集めてきた[1, 2]。運動皮質、脳幹、脊髄にまたがる上部および下部運動ニューロンの損傷 [3-5]、神経筋接合部の欠陥による骨格筋の分解と脱神経 [6 ]、筋力の低下と収縮装置の変化をもたらす緩やかな筋萎縮 [7] などが主な病理学的特徴である。これらの過程はすべて、活性化T細胞から分泌される毒性因子による炎症、活性化マクロファージ/ミクログリア/アストロサイトによるグリオシス、損傷したシュワン細胞や乏突起デンドロサイトによって引き起こされる脱髄を伴っている [8]。
これらの特徴から、ALSがホットな話題であり、研究上の課題であることは疑う余地がない。しかし、ALSは複数の遺伝的・環境的要因が複雑に絡み合って発症するという異質かつ多因子性であり、また世界的に見ても発症率が低いことから、特にヒトの病気を利用しようとする製薬会社にとっては、今後の研究の進展が阻まれる可能性がある。さらに、従来の「1ターゲット1ドラッグ」のパラダイムは、ALSに有効な治療法を提供できておらず、ALSは新しいマーカーと治療法を求める不敗の病である [9, 10]。
より効果的な治療法を模索する中で、米国食品医薬品局(FDA)で承認された薬剤の新規用途(本来の適応症以外)を特定する薬剤再利用が有望な戦略となっている[11, 12]。
この分野では、ネットワーク理論の概念とツールを応用して、ヒトのインタラクトーム(細胞内のあらゆる物理的相互作用の統合ネットワーク)の構造特性、その機能組織、およびその摂動がもたらす結果との関係を解明している[13-18]。ネットワーク医学では、疾患は単一の遺伝子変異によって引き起こされることは稀であり、より一般的には、互いに接続された遺伝子のネットワークの制御が解除されることによって引き起こされると考えられている。この革新的なヒト疾患のビジョンでは、インタラクトームは地図であり、疾患は局所的な摂動であると解釈することができる。この地図では,同じ病気に関連する遺伝子は,特定のネットワーク近傍,すなわち「病気モジュール」内に集まる傾向がある[14, 19].病気の影響と同様に、薬の作用もインタラクトームの局所的な摂動として解釈することができ、その結果、特定の疾患モジュールに近い創薬標的は、その疾患に対してより有効である傾向がある[13, 20-22]。
本研究では、SAveRUNNER (Searching off-lAbel dRUg aNd NEtwoRk) [23 , 24] と呼ばれるネットワーク医学ベースの新しい薬剤再利用アルゴリズムをALSにおける薬剤の適応外使用の可能性を事前に予測するために採用している。ALS関連遺伝子と創薬標的の相互作用をヒトインタラクトームで定量化することにより、新たな創薬関連性を見出す。
研究方法
データ検索
ヒトタンパク質-タンパク質相互作用は、Cheng and coauthors [21]からダウンロードした。このデータベースは、著者らが、いくつかのタイプの実験的証拠(例えば、…)を持つ15の一般的に使用されるデータベースとインハウスで体系化したヒトタンパク質-タンパク質相互作用の集合体である。例えば、3次元タンパク質構造からのバイナリPPI、アフィニティ精製後質量分析、Y2H、および/またはBioGRID [25], HPRD [26], MINT [27], IntAct [28], InnateDB [29] などの文献由来のロースループット実験により同定された文献-キュレーションPPI、文献由来のロースループット実験からのシグナリングネットワーク、文献由来のロースループットとハイスループットから得られたキナーゼ-基質の相互作用)である。このバージョンのインタラクトームは、15,970個のユニークなタンパク質に接続する217,160個のタンパク質-タンパク質相互作用で構成されている。
疾患関連遺伝子は、Phen-opedia[30]からダウンロードした。
DrugBank [31]には、2627の承認済み低分子医薬品、1373の承認済み生物製剤、131の栄養補助食品、6370以上の実験的医薬品を含む13,563の医薬品エントリーがある(2020年4月22日にリリースされた)。DrugBankから提供された標的Uniprot IDは、BioMart – Ensemblツール( www.ensembl.org/ )を用いてEntrez遺伝子IDにマッピングされた。DrugBankでターゲットが見つからない一部の薬物については,Therapeutic Target Database [32]から得られる薬物-ターゲット間相互作用を統合した。
研究デザイン
解析のパイプラインは以下のようにまとめられる(図1 )。ヒトインタラクトームネットワーク(ヒト細胞の分子構成要素をノードとし、それらの間に物理的相互作用が存在する場合にリンクが生じる)、および疾患関連遺伝子リストを入力として、Random Walk with Restart(RWR)アルゴリズムを適用し、ヒト疾患ネットワークを構築した。ここで、ノードは疾患であり、ALSの疾患モジュールから出発して、アクトーム間においてランダムウォーカーが他の疾患モジュールに到達する可能性が高い場合、ALSと他の疾患の間にリンクが生じる(「Random Walk with Restart Algorithm」参照)。次に、RWRアルゴリズムと同じ入力データに、薬物-ターゲット相互作用を追加したものを与え、SAve-RUNNERアルゴリズムを適用した。SAveRUNNERは、ヒトインタラクトームネットワークにおける薬物モジュールとALS疾患モジュールの近傍性を利用して、ALS疾患に対する再利用可能な候補となる薬物を探索した(「SAveRUNNERアルゴリズム」参照)。SAveRUNNERは、FDA承認の医薬品と疾患をノードとする医薬品-疾患ネットワークを出力として与え、それらの間のリンクは、医薬品がその疾患に対して再位置付けされると予測された場合にのみ発生する。SAveRUNNERがALSに再利用可能であると予測した薬剤のうち、ヒト疾患ネットワークにおいてALSに関連する疾患を本来の適応症とする薬剤に注目した。
SAveRUNNERアルゴリズム
最近、我々は、現在販売されている薬剤の適応症の可能性を効率的にスクリーニングし、仮説検証実験の有効性を最適化することを目的として、SAveRUNNERと呼ばれるネットワーク医学ベースの薬剤再利用のための新しいアルゴリズム[23]を開発した。SAveRUNNERアルゴリズムの詳細な説明は、参考文献[23, 24]に記載されている。
遺伝子セットエンリッチメント解析
SAveRUNNER によって予測された抗 ALS 薬候補が ALS 病態によって引き起こされる遺伝子発現の擾乱に対抗できるかどうか(すなわち、ALS によって制御される遺伝子を増加させることができるか、またはその逆か)を検証するために、遺伝子セット濃縮分析(GSEA)を行った。まず、ALS患者およびコントロールサンプルの3つの遺伝子発現データセットをGEO公開リポジトリから収集した。特に
(i) ALS患者4名と対照者4名の人工多能性幹細胞(iPSC)由来運動ニューロンのハイスループットシーケンスによる発現プロファイル(RPKM正規化発現値に対応)(GSE52202 [33] )。
(ii) ALS患者7名と対照者4名の死後脊髄灰白質のアレイによる発現プロファイリング(Affymetrix Gene-Chip 3.1 ソフトウェアから得られたノルマル化発現値に対応)(GSE833 [34] ); (iii) ALS患者10名と対照者10名の死後中枢神経組織の広範囲なコホートのアレイによる発現プロファイリング(ロゼッタ誤差モデル[35]を用いて得られたノルマル化発現値に対応)(GSE26927 [35] )。GSE833データセットでは、GEO reposi-toryから入手可能なプラットフォームGPL80 (Affymetrix Human Full Length HuGeneFL Array) を用いて、プローブセットを公式遺伝子シンボルにマッピングした。ある遺伝子の複数のプローブ測定値は、平均値を考慮することにより、1つの遺伝子測定値に折り畳まれた。各データセットについて、発現値を対数(log2)変換し、各遺伝子の四分位範囲(IQR)の計算による前処理解析を行うことにより、データを処理した。IQRは中央値付近のデータのばらつきの指標であり、データ分布の75パーセンタイルと25パーセンタイルの差に相当する。IQR分布の10パーセンタイルより小さいIQR値を持つ遺伝子(中央値付近のばらつきが少ない遺伝子に相当)は、フィルターで除外された。GSE52202 と GSE26927 ではノンパラメトリックの Wilcoxon 符号順位検定を、GSE833 ではノンパラメトリックの Mann-Whitney 検定を行った。最後に、得られたp値をBenjamini-Hochberg法により多重仮説検定用に調整した。統計的に有意な発現遺伝子を選択するために、サンプル数が最も多いデータセット(GSE26927)の調整済みp値の閾値を0.05に設定した。他の2つのデータセット(GSE52202とGSE833)では、修正p値は常に標準的な有意水準よりも大きくなっている。そこで、これらのデータセットについては、p値の調整をやめ、元のp値に対してより厳しい0.01の閾値を用いることによって、この欠点を補うことにした。このように定義された3つの発現差異遺伝子リストを、3つのALSシグネチャーとして使用した。
次に、L1000 assayで得られたハイスループットな縮小表現遺伝子発現データを集めたConnectivity Map (CMap) データベースにクエリーをかけた[36, 37]。L1000プロファイリングは、培養・処理プロトコルが確立されている様々な薬剤処理ヒト細胞株で実施される。このように、CMapデータベースは、化学的・遺伝的刺激に対するヒト細胞の転写反応をカタログ化したものである。9つの細胞株のコアセットで、合計27,927の妨害因子がプロファイリングされ、476,251の発現シグネチャーが作成されている。CMapデータベースから薬剤処理したヒト細胞株の差分発現遺伝子を薬剤シグネチャーとして使用した。
CMap データベースと SAveRUNNER で ALS に有効であると予測された薬剤について、CMap クエリツールを用いて ALS の表現型の特徴である差分発現遺伝子に対する治療効果を評価し、分離入力リストとして与えられた各 ALS シグネチャーを使用した[36]。疾患シグネチャーと薬剤シグネチャーをfold-changeの増加順に並べ、CMapは薬剤の効果が疾患の効果を打ち消すか(ES < 0)、そうでないか(ES > 0)を測定する濃縮スコア(ES)を計算した [37, 38]。これは、疾患シグネチャーと薬剤シグネチャーを比較し、疾患シグネチャーで最も発現量の多い(発現量の少ない)遺伝子が薬剤シグネチャーの下(上)付近にあるかどうかを判断するというものである。これは薬剤と疾病の発現プロファイルが一致する(ES < 0)ことを意味し、その薬剤は対象疾病の治療オプションとなる可能性がある。このスコアの計算の詳細は、[37-39]に記載されている。特に、選択されたリパーポージング候補薬剤は、薬剤シグネチャーがALSシグネチャーと負の相関がある場合、ALSに対する治療効果の可能性があると考えられた。薬物と疾患は、対応するESが負の場合、負の相関があるとし、その疾患のシグネチャーに対してその薬物に1というスコアを割り当てた。20]で採用された方法にヒントを得て、この基準を満たすALSシグネチャーのデータセット数を各薬剤の最終的なGSEAスコアとして使用した。本研究では、N = 3 疾患のシグネチャを考慮することで、各薬剤の最大 GSEA スコアは 3 となる。
モジュールの意義
ネットワーク医学の原則[13, 14, 19]で確立されているように、疾患関連遺伝子は、他の遺伝子と区別するユニークで定量化可能な特性を持っている。このことは、疾患関連遺伝子はアクトーム間においてランダムにマップされるのではなく、むしろ局所的に密で位相的によく定義されたネットワーク領域(疾患モジュールと呼ばれる)に集積し、そのノード同士が偶然に予想されるよりも頻繁に相互作用する傾向があることを検証することができる。
我々は、ALSに関連する遺伝子(他の疾患にも関連する遺伝子)が、ヒト・インタラクトームの局所的な領域に集積する傾向があり、統計的に有意な疾患モジュールを構成しているかどうかを調査した。そのために、疾患関連遺伝子をヒト・インタラクトーム上にマッピングし、対応する疾患サブネットワークを抽出し、以下の3つの指標を計算した[17]。(相互作用(エッジ)の総数、最大連結成分(LCC)のサイズ、LCC内のエッジの数。この指標は、与えられた疾患遺伝子のリストが、偶然に予想されるよりも頻繁に、あるネットワーク近隣に局在する確率を測定するものである[17]。具体的には、各疾患について、ヒト・インタラクトームの元の疾患遺伝子リストと同じサイズと次数分布のタンパク質群をランダムに選択した。そして、対応するランダムサブネットワークを抽出し、上記の3つの指標を計算した。この手順を1000回繰り返した。この1000回の並べ換えは、最先端の手法[40-48]で報告されているように、無作為化検定の検出力を推定するためによく使われ、有意水準5%の検定では妥当であると考えられている。最後に、ランダムな遺伝子セットによって誘導されるサブネットワークに対応する3つのメトリクスの分布を導出した。zスコアを用いて、疾患遺伝子のオリジナルリストに対して計算された3つのメトリクスを、対応する参照ランダム分布に関して正規化し、与えられたz統計量に対するp値を割り当てた。3つの指標すべてが統計的に有意であれば(p値≦0.05)、疾患遺伝子はヒト・インタラクトームにおいて統計的に有意なモジュールを形成していると結論づけることができる。
Random Walk with Restart アルゴリズム
ALSの疾患モジュールとヒトアクトーム間ネットワークにおける他の疾患モジュールの近接度を測定するために、Random Walk with Restart (RWR) アルゴリズムを実装した。RWRは、ランダムウォークを中心とした直感的な概念に基づくアルゴリズムである。あるノードxから出発したランダムウォーカーが与えられたとき、各反復において、隣接するノードの一つに移動するか、ある確率でxに戻るかの二つの異なる選択肢が存在する。したがって、課題はウォーカーが行きそうな最も可能性の高い場所を計算することだ。形式的には、RWRアルゴリズムは以下の式で記述される。
Rt = WRt-1 + (1 – )E
ここで、W はネットワーク隣接行列で、ノード間の遷移行列を表し、その要素 W[i, j] はノード j からノード i への遷移確率を表す。E は始点ベクトルで、その要素 E[i] は i が始点の場合は 1、それ以外は 0 に等しい。Rt は確率ベクトルで、その要素 Rt [i] は繰り返し t 時にノード i にいる確率を表す; (0,1) の範囲の数で、 (1 – ) は繰り返し毎に始点ノードから「再出発」する確率を表す。確率ベクトル Rt は収束点(Rt = Rt-1、または滞在確率と移動確率の差がある閾値より小さくなる)に達するまで繰り返し計算される。最後に、RWRはネットワーク内の各ノードの定常状態の確率のベクトルRを出力として返す。
ヒトインタラクテオームから構築した隣接行列Wmxmを遷移行列とし、ALS疾患遺伝子のベクトルEを始点ベクトルとして、RWRを実行した。到達頻度が30を超える(すなわち、到達した疾患遺伝子の数が30を超える)疾患のみを選択した。各疾患について、対応する疾患遺伝子のRWR定常確率を平均化し、各疾患の平均確率、つまりALSからスタートした場合の到達確率を求めた。この疾患確率を、次のように定義される修正z-scoreを用いて正規化した。
ここで、x は疾患確率、x はすべての疾患確率の分布の中央値、MAD は中央値絶対偏差で、標本中央値からの観測値の差の絶対値の中央値(すなわち、中央値(x – x ))、c は 0.6745 に等しいスケールファクターで、zmod が正規分布に対する標準 z-score と等しくなるように定義される[49]。このzで正規化した疾患確率をALS近接度と名付け、zスコアに対応するp値を割り当てた。ALS 近縁度 2.5 の値は正の外れ値、-2.5 未満の値は負の外れ値としてラベル付けすることが可能である。正の外れ値に対応する疾患は,ALSから始まるran-dom walkerによって到達する可能性が高く,ALSに近い疾患を表している.
モジュール分離
ALS 疾患モジュールの他の疾患モジュールに対する非ユークリッド分離距離を以下のように計算した[50]。
分離距離の値が正であれば、2つの疾患モジュールはヒトインタラクトームにおいて位相的によく分離していることを示し、分離距離の値が負であれば、2つの疾患モジュールは同じネットワーク近隣に位置しており、重なりあっていることを示す。そこで、ヒト・インタラクトームから無作為に選んだ、疾患遺伝子と同じ大きさと次数分布を持つ2つのタンパク質群間の距離を基準距離として、モジュール分離の重要性を評価した。参照距離分布を構築するために、無作為抽出を1000回繰り返した。2つの疾患遺伝子リスト間のモジュール分離指標は、参照分布の平均と標準偏差を使用してz-scoreで正規化した。その後、与えられた z 統計量に対する p 値を計算した。p値<0.05は、疾患遺伝子の2つのリストのヒトインターゲートにおけるモジュール分離が、偶然に予想されるよりも多い(または少ない、下記参照)ことを示す。
パスウェイエンリッチメント解析
最も有望なヒスタミン関連化合物(すなわち、アモキサピン、クロミプラミン、ミアンセリン、モダフィニル)の標的遺伝子が関与する経路を調べるために、Enrichr web ツール [53] を用いて、京都遺伝資源データベース [51] と Reactome パスウェイ知識ベース [52] に問い合わせを行った。p 値は Ben-jamini-Hochberg 法で調整し、0.05 の閾値を設定して、入力リストとして与えられた標的遺伝子に有意に濃縮された分子パスウェイを同定した。
結果
ALS治療薬ネットワーク
本研究では、最近開発されたSAveRUNNERアルゴリズム[23, 24]を適用し、ALSの再利用可能な薬剤候補を同定した。
表 1 SAveRUNNER により ALS 向けに再利用可能な薬剤として同定されたヒスタミン受容体モジュレーター(対応する Drug Bank 番号付き)の一覧表
原文参照
各リガンドのH1Rに対する作用について、SAveRUNNERは入力として創薬標的のリストを必要とし、与えられた薬剤が最終的にどの程度疾患治療に再利用できるかを評価するために疾患遺伝子のリストを必要とする。
ここで、疾患関連遺伝子は Phenopedia [30] からダウンロードし、ALS 関連遺伝子 267 個を提供し、一方、薬剤-標的の関連は DrugBank [31] から取得した。特に、FDA承認薬1860種のターゲット情報を収集した。薬物標的の入力リストには、単体の薬物に加え、ALS研究で最近注目されている80の薬物の組み合わせも考慮した。1940種類の薬剤の全リストと、それに対応する標的タンパク質の数を補足表1に示す(合計2138種類の標的タンパク質が存在する)。
SAveRUNNER の背景には、ある薬剤が特定の疾患に対して有効であるためには、その薬剤に関連する標的(薬剤モジュール)と疾患特異的な関連遺伝子(疾患モジュール)がヒトインタラクトーム上で近接していなければならないという仮説がある[21]。SAveRUNNERは、薬剤モジュールと疾患モジュールの近傍性を定量化するために、新しいネットワーク類似性指標を実装し、次数保存ランダム化手順を適用してその統計的有意性を評価する[23]。
SAveRUNNERの特徴は、予測された適応外薬物の適応を優先順位付けする手順を実装していることだ。この優先順位付けは、ある薬とある病気が一緒になっている場合、その薬はその病気に対して効果的に再利用できる可能性が高いという仮定に基づいて、同じクラスターに属する病気と薬の間の関連性を評価するクラスタリング分析を利用したものである。この意味で、同じグループに属する医薬品と疾患は、他のグループに属する医薬品と比較して、より類似していると言える。
SAveRUNNERは、対応する創薬ターゲットと疾患遺伝子がインタラクトーム上で偶然に予想されるよりも近い場合に、創薬と疾患のリンクが発生する重み付き二部構成の創薬-疾患ネットワークを出力として放出する。その相互作用の重みがネットワークベースの類似性指標に対応する。
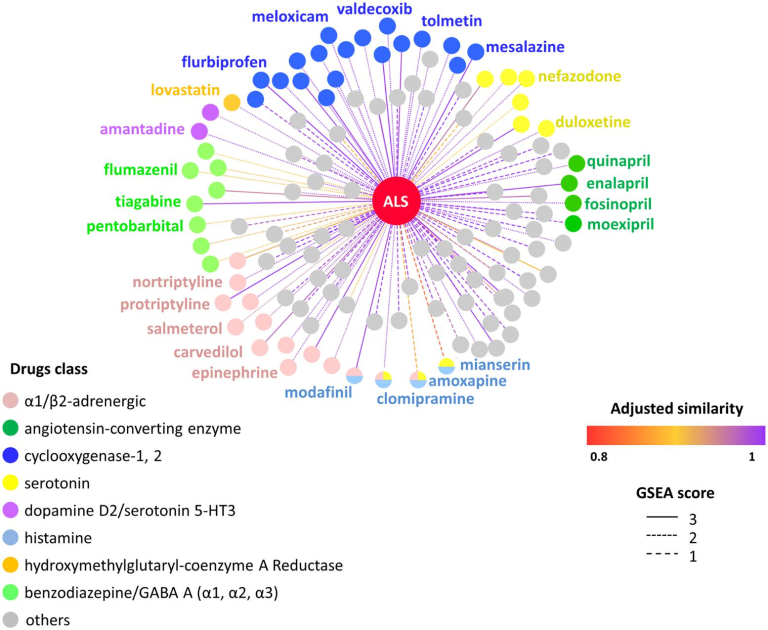
図2 予測されたALSの薬物-疾患ネットワークの模式図
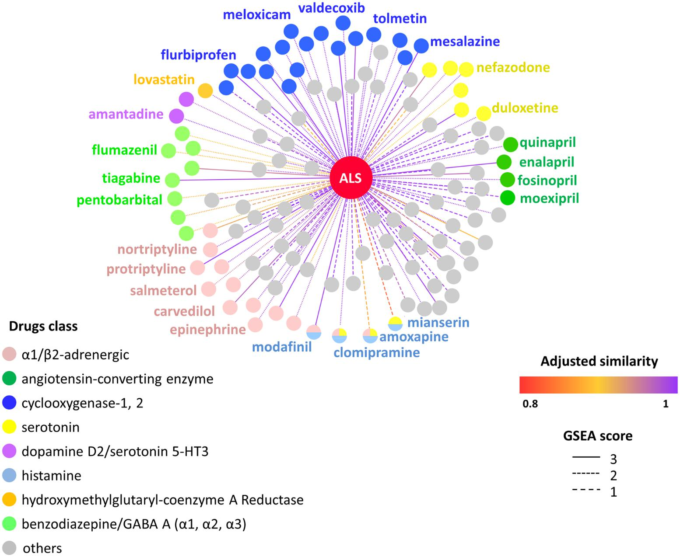
この図は、ALSをGSEAスコア>0を示す121のFDA承認された非ALS薬剤と接続する高信頼性の予測された薬物-疾患関連(p値<0.07)を示している。薬剤は、伝説で報告された受容体/酵素クラスの標的に従って色付けされている。エッジの色は調整された類似性を示している。
本研究では、SAveRUNNERにより、H2R、H3R、H4R受容体がALSに有意に関連する再利用可能な医薬品403種(1940種中)が、その追加受容体リガンド結合特性( https:// www.drugbank.ca/drugs/ より)(p値 < 0.07)と共に示された(補表2)。その中で、大脳新皮質と視床下部でのヒスタミン放出とレベルを上昇させ[54-56]、その効果は神経細胞ヒスタミンの枯渇によって消失し[57]、ナルコレプシーの過眠の治療や運動量の増加に用いられる薬剤、modafinil(調整類似度値=0.99、p値=0.04)を発見した。モダフィニルはドーパミントランスポーターにも大きく作用し、ドーパミンの再取り込み阻害剤として働き、さらにグルタミン酸回路を活性化し、GABA作動性回路を阻害する [58] 。ALSとモダフィニルのヒスタミン作動性関与を裏付けるように、SAveRUNNERは、アモキサピン[59、60]など、主にヒスタミン作動性システムに干渉する(ただし、それだけではない)追加の薬物を同定した(修正類似度=0. 89, p値=0.006)、clomipramine [61] (調整済み類似度=0.99, p値=0.003)、mian-serin [62] (調整済み類似度= 0.92, p値=0.06)である(図2、表1、補足表2参照)。さらに、アリモクロモール、エリスロポエチン、マシチニブ、ミノサイクリン、オザネズマブ、ペランパネルなど、ALSの臨床試験で既に採用されている化合物の組み合わせ検索や解析を行った結果、SAveRUNNERは、modafinilが確かにp値や修正類似度ランキングを向上させることを明らかにした(修正類似度 = 0.003)。 99、p値=0.05)、マシチニブ(調整済み類似度=0.99、p値=0.04)、ペランパネル(調整済み類似度=0.99、p値=0.05)である(補足 表2)。
抗ALS再利用可能な薬剤のGSEA解析
SAveRUNNER が予測した抗 ALS 薬をさらに検討するため、ALS 患者の神経系組織のトランスクリプトームデータを疾患シグネチャーとして、Con-nectivity Map (CMap) データベースから薬剤処理したヒト細胞株の遺伝子発現データを薬剤シグナルとして GSEA を実施した。SAveRUNNERによって予測され、CMapデータベースに含まれる各薬剤について、ALSの病態に起因する遺伝子発現擾乱に対抗しうる指標としてGSEAスコアを算出した。特に、各 ALS データセットについて、CMap クエリツール[37-39]に従って ALS シグネチャーと負の相関があるシグネチャーを持つ薬剤を、ALS 表現型の特徴である遺伝子に対して治療効果が期待できるものとして選択した(「方法」参照)。GSEAスコアは0から3まであり、特定の薬剤についてこの基準を満たすALSデータセットの数に対応する。
GSEA解析の結果、403の候補薬剤のうち、GSEAスコア3が21、2が52、1が48で、合計121がALSに対するリポジショニングの対象となった(図2、補足表2)。
GSEAスコア0以上の薬剤は、ヒスタミン受容体調節薬、アンジオテンシン変換酵素(ACE)阻害薬、プロスタグランジンG/H合成酵素1および2(シクロオキシゲナーゼ1/2アイソザイム)阻害薬、α1/β2アドレナリン受容体、ドーパミンD2受容体、ベンゾジアゼピン/GABA受容体調節薬などALSのメカニズムや経路と直接的に関わっているいくつかのクラスと相互に関連している(Fig. 2、補足表2)。ヒスタミン調節剤の中で、モダフィニルが3という最も高いGSEAスコアを示したことは重要な点である。また、他のヒスタミン受容体モジュレーター(アモキサピン、クロミプラミン、ミアンセリン)もGSEA解析でGSEAスコア>0であることが確認された(表1)。
ALSの併存疾患の予測
ALSと他の疾患との潜在的な併存パターンを予測するために、併存率を測定するネットワークベースのアプローチを2つ実施した。(1) RWR (Random Walk with Restart) アルゴリズム、(2) モジュールセパレーション測定。
ALS関連遺伝子がヒトアクトームネットワークにおいて統計的に有意な疾患モジュールを形成していることが確認された後(補足表3)、RWRアルゴリズムを適用した。このアルゴリズムは、ALS疾患モジュール(出発点)とヒトインターゲームの他の疾患の間の近さを計算し、それぞれの疾患に修正zスコア正規化値(ALS close-ness scoreとして定義)を割り当てる(「方法」参照)。このスコアが2.5より大きい値は正の外れ値であり、ALSに近い疾患とみなされた。その結果、前頭側頭型認知症(修正zスコア=6.65)、大脳皮質認知症(修正zスコア=5.21)、筋力低下(修正zスコア=5.18)、神経筋疾患(修正zスコア=3.27)、多発神経炎(修正zスコア=2.25)が検出され興味深い結果であった。 89)、糖尿病性神経障害(修正z-score = 2.58)など、重症度、併存疾患、薬物療法は異なるものの、いずれもALSといくつかの特徴を有する病態であり、SAveRUNNER/GSEAが事前に予測した抗ALS薬(例, また、SAveRUNNER/GSEAで事前に予測された抗ALS薬(clomipramine、mian-serin)は、もともと承認されていたものである(図3a、補足表3)。
この結果は、ALSモジュールと他の解析対象疾患とのモジュール分離を計算することによっても確認されたようである(図3 b、c)。まず、すべての解析対象疾患(神経筋疾患、筋力低下、前頭側頭型認知症、多発性神経炎、血管性認知症、糖尿病性神経炎)が、ヒトインターフェイスネットワークにおいて統計的に有意な疾患モジュールを形成していることを確認した(Supplement Table3)。その結果、ALSの疾患モジュールは上記の疾患モジュールと統計的に有意に重複しており(図3b)、負の分離値およびp値≦0.05を示した(図3c、補足表3)。このことから、ALS治療の可能性は、他の特定の疾患に対して承認されている治療法のアーセナルから得られることが確証された。
ヒスタミン関連化合物のパスウェイ解析
解析で指摘された最も有望なヒスタミン作動性化合物(すなわち、アモキサピン、クロミプラミン、ミアンセリン、モダフィニル)がALS疾患経路に作用するかどうかを調べるため、次に、WebツールEnrichr [53] による機能濃縮解析を行い、それらの標的遺伝子が関与している経路を調べた。その結果、「アミンリガンド結合受容体、クラスA/1(ロドプシン様)受容体、GPCRリガンド結合、GPCR下流シグナル伝達、Gα(i)/Gα(q)/Gα(s)シグナル伝達イベント」などのパスウェイが有意に濃縮されていることがわかった。ヒスタミン受容体活性化のシグナル伝達機構とALS発症時に制御が低下することが知られている経路を含む「PKCとMAPKを介したCREBシグナル伝達経路、アミン化合物SLCトランスポーター」(Fig. 4および補足表4)。
考察
ALSを含む中枢神経系疾患は、新しい技術により、かつてないスピードで多くの成果を生み出している。オミックスデータ、デジタル化された医療記録、全ゲノム配列、表現型シグネチャーなど様々な種類のデータを取り込むことにより、人間の健康や病気に関連する問題に取り組むために、スケーラブルなアルゴリズム手法などの革新的な計算アプローチが現在確立され実施されている[63]。ここでは、最近開発されたSAveRUNNERと呼ばれるネットワーク医療ベースのアルゴリズムをALSに再利用可能な薬剤の同定に適用することに成功した。この研究は、実験的な知識を計算機的な解析に結びつけ、その情報を臨床に応用することを最終的な目標としている。ALSの治療薬として、カテコールアミン、ドーパミン、セロトニン、ヒスタミン、GABA受容体モジュレーターに加え、ネットワークの類似度が高い順に優先順位を付け、ALSの治療薬となる可能性のある薬剤を予測した。また、アンジオテンシン変換酵素、シクロオキシゲナーゼアイソザイム、セロトニントランスポーター阻害剤と同様に、modafinil (2-[(diphenylme-thyl) sulfinyl] acetamide) などの慣習的でない興味深いALS治療薬を発見した (p value≤ 0.) 。 05、GSEAスコア=3)。この化合物は、強力で長時間の覚醒促進物質としてずいぶん前に開発され、1998年に睡眠障害の治療薬としてFDA(プロビジル®)から承認された[64]。モダフィニルは睡眠不足の状態で認知機能を正常化し、非中毒性であることから、ニコチンやコカイン中毒、さらには注意欠陥、感情障害、うつ病、統合失調症に対する臨床研究が行われており、前臨床試験ではアルツハイマー病やパーキンソン病における効果も示唆されている[65-68]。さらなる評価では、モダフィニルが慢性神経疾患 [69]、筋緊張性ジストロフィー2型 [70]、そして驚くべきことにALS [71]における疲労症候群をわずかに改善することが示され、Save-RUNNER/ GSEAによる薬剤-疾患ネットワーク予測結果から得られたモダフィニルの優先順位が確認・検証された。
モダフィニルが正確に作用する脳領域を特定することは困難であることが証明されているが [72]、主な標的は以下の通りである。皮質下視床、視床下部および扁桃体:覚醒および認知能力の活性化および維持の強化 [73]; 視床皮質回路:皮質介在ニューロン間の電気的結合の増加 [74]; 視覚、前頭皮質および小脳:全機能結合の増加 [75]; 最後に脊髄の腹内側領域:反射亢進状態の正常化 [76]. 運動ニューロンを包含する腹側脊髄と前頭葉および運動皮質は、ALSにおいて明確な神経変性パターンを示し、さらに視床および小脳のびまん性異常もALS患者にあらかじめ認められるので [77, 78] 、これらの同じ脳領域を標的とするmodafinilは、ALSに対するSAveRUNNER/GSEA予測結果を支持し、その利用および再利用を促進することになるであろう。
モダフィニルの覚醒促進作用に関する研究の多くは、脳内のヒスタミン、ノルエピネフリン、セロトニン、ドーパミン、オレキシン系を刺激することによるモノアミン作動性作用に焦点をあてている。モダフィニルは、ドーパミン再取り込み阻害剤 [58] として作用するほか、間接的にヒスタミン作動性システム [72] を活性化し、おそらくは視床下部後部の結節性乳頭核にあるヒスタミン作動性ニューロンへの抑制性GABA作動性入力の減衰とオレキシン作動性ニューロンによるヒスタミン作動性緊張の強めにより、活性化する [55, 72, 79, 80]。モダフィニルが回路レベルでどのように機能するかはまだ不明であるが、ヒスタミン作動性回路の調節を通じてALSに作用する可能性は、アモキサピン、クロミプラミン、ミアンセリンなどのいくつかの追加のヒスタミン作動性調節物質が、0以上のGSEAスコアを持つことで実際にALSに著しく関連しているという我々のSaveRUNNER/GSEA分析によって立証されている。注目すべきは、SAveRUNNER/GSEAのヒスタミン作動性予測は、ALSの最も特徴的な動物モデルであるSOD1-G93Aマウスで得られた前臨床結果による確かな実験的支持を得ていることである[81]。ヒスタミン前駆体ヒシジンは、ALSの病理的特徴を改善する、病気の進行を遅らせる、運動能力を改善する、寿命を延ばす、脊髄の運動ニューロン損失と神経炎症を減らす、最後に神経-筋結合部の断片化と筋肉萎縮を減らすことが証明されている[82]。実際、ヒスタミン化合物は、様々な疾患動物モデルにおける薬物ターゲットとして新しいものではなく、さらに、ALSを含む幅広い臨床CNS状態 [83] において明確な価値を示すことが示唆されている [84, 85]。
ヒトの疾病分類に対する現代のアプローチは、病理学的解析と臨床状態に関する既存の情報との観察的相関関係に基づいている部分があり、旧来の臨床診断の指針となった還元主義のパラダイムが原因で、多くの疾病の相互関連性が無視されがちであった。しかし、現在では、Network Medicineの情報とデータの相関を利用した疾患分類に注目が集まっている。本研究では、ネットワークに基づくモジュール分離指標とRWRアルゴリズムを採用することで、ALSにおけるヒスタミン作動性化合物の前向きな使用について、さらなる確証を得ることができた。特に、ミアンセリンやクロミプラミンなどのヒスタミン受容体モジュレータによって臨床的に既に緩和されている認知症や多発性神経炎などの病態とALSとの併存パターンや疾患モジュールの重複を明らかにした。この文脈では、ALSは、主要な疾患原因遺伝子と、すべての疾患に共通する疾患修飾遺伝子、およびそれらのネットワークに基づく環境および行動決定因子(転写またはエピジェネティックレベルで遺伝子発現を制御するもの、転写後のプロテオーム修飾やメタボロームの独自性を駆動するもの)を統合する一連のリンクしたネットワークの崩壊と見なすことができる。これらのサブネットワークは、今回の解析で明らかになったように、認知症とALSのような複雑な疾患の併存をもたらす一方で、それぞれの臨床表現型を非常に個別化した形で区別することに寄与しているのである。今後、薬物・疾患ネットワークとALSの相互作用をさらに解明することで、ALSをより包括的かつ正確に地図化し、より好ましい治療法の出現を促すことができると確信している。
要約すると、本研究では3つの重要な知見が得られた。まず、SAveRUNNER/GSEA による予測は、いくつかのヒスタミン作動性化合物が ALS の治療薬開発においてトランスレーショナルインパクトを持つかもしれないという、前臨床試験で確認された我々の仮説を支持するものであった。第二に、様々なヒスタミン作動性薬物の中で、我々の分析は、ALSでよく知られている運動回路を制御する部位と一部重なる脳の特定部位を標的とする薬剤であるmodafinilに優先順位をつけていることだ。この考え方は、Modafinilの作用機序や標的をさらに解明し、ALSの特徴を細分化し、病気を追跡するための基礎となるものである。最後に、ネットワーク予測型とこれまでの前臨床試験をマッチングさせることにより、我々のSAveRUNNER/GSEAアルゴリズムは、ALSに機能する可能性のある薬剤の特定と将来の臨床評価のためのより統合的なフレームワークを導入している。
ALSを克服するための「1分子1標的」戦略はこれまで失敗しており、次に取り組むべきは「マルチターゲット薬」「ポリファーマコロジー」アプローチによる治療法であり、ALSの様々な異質な側面を理解し、病気の進行を止めることだと強く信じている。この点でも、モダフィニルは非常に有望な候補であると思われる。モダフィニルは、ヒスタミン作動性、ドーパミン作動性、グルタミン酸作動性、GABA作動性の神経伝達を阻害するだけでなく、マシチニブ(86)、エリスロポイエチン(87)、ペランパネル(88)などのALSの臨床試験で実際に使われている化合物と組み合わせると、p値や調整済み類似性ランキングが有意に改善する薬剤としてSaveRUN-NERで特定されている。ヒスタミン作動性薬剤は、ALSに再利用される可能性のあるいくつかの要件を確かに満たしているが、ALSの発症、進行および薬理学的治療に関する理解を深めるためには、薬剤可能な標的の検証を継続しなければならず、ヒスタミン信号伝達、その投与および薬剤可能性に関するさらなる研究が確実に必要である。今回のSAveRUNNER/GSEAやモジュール分離解析で示されたように、事前予測値が成熟しスケールアップするにつれ、継続的な統合も必要であり、これが我々の継続的かつ将来の目標である。
実際、SAveRUNNER/GSEA解析によって実装された薬剤リポジショニングに対するネットワークメディスンのアプローチは、ALSに対するさらなる調査と実験的検証に値する、有望な再利用可能薬剤候補の発見における革新を大きく促進することができると考えている。しかし、ネットワークとヒト疾患の相互作用を探る最初のステップは、現在の分子およびフェノタイプネットワークマップがヒトに対してどの程度包括的で正確であるかを評価することだ。ここ数年、ヒトのインタラクトームマップのカバー範囲を広げ、インタラクトームのサイズを推定し、既知のバイアスを補正するための系統的な取り組みが行われてきた。しかし、ヒトのインタラクトームマップは依然として不完全でノイズが多いため、疾患の研究においてこの事実を考慮する必要がある。しかし、ALSに関連する遺伝子のリストはまだ完全ではなく、ALSの表現型に影響を与える変異遺伝子の発見により、常に更新されている。したがって、文献に基づく入力パラメータの不完全性に悩まされ、SAveRUNNERもまた不完全な予測を導く可能性がある。この点で、最初の例は、SOD1-G93AマウスモデルでALSの病勢を改善することが示されたヒスタミンH1受容体直接拮抗薬のクレマスチン[89、90]かもしれないが、SAveRUNNERアルゴリズムが予測した候補薬剤には含まれない。一方、ヒスタミン作動性仮説を満たすものとして、SAveRUNNERは、新皮質と視床下部でのヒスタミン放出とレベルを上昇させる、従来にないヒスタミン受容体調節薬、modafinil(プロビジル®)を指摘した[54、55、91]。SAveRUN-NERは、時間とリソースの大幅な節約を可能にし、従来の創薬前臨床研究ではALSのために考慮されなかった、有望で研究価値のある候補を特定することができたと思われる。
これらの考察から、ALSに内在する複雑な病態をより深く理解し、さらに重要なことに、この病気の治療法を開発するために、ネットワークベースの解析と前臨床薬物試験の相乗効果をさらに見出すことが切実に求められていることが確認された。治療法を探す試みはこれまですべて失敗しており、現在の治療法では罹患率を下げることしかできないが [10] 、ALSに対する正しい答えは、モダフィニルやヒスタミン作動性化合物が実際に行っているように、複数の病態経路に干渉できる多剤併用戦略または幅広いスペクトルの分子によって得られるかもしれないと我々は考えている。何がALSの治療法を改善するのか、それは今後の研究によって明らかになるだろう。