Molecular characterization and the mutation pattern of SARS-CoV-2 during first and second wave outbreaks in Hiroshima, Japan
pubmed.ncbi.nlm.nih.gov/33544733/
公開 2021年2月5日
要旨
背景
本研究では、広島で発生したSARS-CoV-2株の分子解析を行い、第1波と第2波の間の変異パターンを明らかにした。
研究方法
広島での第一波の鼻腔スワブサンプル55検体と第二波の鼻腔スワブサンプル13検体をRT-qPCRで定量的に、また特定のプライマーを用いたネステッドPCRで定性的に調べた。各ウェーブの4検体について次世代シークエンシングと系統樹解析を行い、対照を含む系統樹解析を行った。その後、突然変異を調べた。
結果
ウイルス負荷は7.85×101~1.42×108 copies/mlであった。68検体中,アジア型O型が1検体,欧州型GRが65検体,検出不能が2検体であった。系統樹解析の結果,日本では1月から4月にかけてアジア型(L,S,V,O)に感染していた。3月第2週までにヨーロッパ株(G, GH, GR)が出現し,3月中旬以降はGR株が優勢となった。広島での第1例はアジア株O、残りはGR株であった。その後、11~15の塩基変異を持つGR株の第2波が独立して出現した。第1波と第2波のGR株を比較すると,1年あたりの変異率は1.17~1.36×10-3塩基置換で,ORF1aではS1361PとP3371S,ORF1bではA314V,NではP151Lでアミノ酸の変化が見られた。GRの7株はすべてD614G変異株であり、NではR202KとG203Rの変異が見られた。ORF8タンパク質に欠損をもたらすORF8の1ヌクレオチド挿入が第2波からの1株(S66)に見られた。
結論
本研究では、日本におけるSARS-CoV-2の進化階層を明らかにした。また、広島ではD614G変異体が優勢であることや、新たなORF8欠失が見られたことから、SARS-CoV-2の局所発生におけるウイルス因子の役割を明らかにする手がかりとなった。
競合する利益
著者らは、競合する利益は存在しないと宣言している。
背景
新型コロナウイルスは、以前は2019-nCoVと命名され、後に国際ウイルス分類学委員会(ICTV)によって正式にSARS-CoV-2と呼ばれるようになったが、SARS-CoV-1やMERS-CoVを含むBetacoronavirus属に属している[1]。この新規コロナウイルスは、約30kbのポジティブセンス一本鎖RNAゲノムであり、RNAウイルスの中では最大のゲノムである[2]。
SARS-CoV-2は下気道感染症を引き起こし、重症急性呼吸器症候群や多臓器不全に進行する [3,4]。このウイルスに感染した結果、100万人以上の方が命を落としている。2019年12月下旬に中国・武漢でSARS-CoV-2が検出された後 2020年9月27日時点で世界で約3,300万人の確定症例と99万6,309人の死亡が報告されている[5]。世界保健機関(WHO)は2020年3月11日にこの感染症をパンデミックと宣言した[6]。
2020年8月3日現在、日本では合計40,244人の確定症例と1,018人の死亡が報告されている[7]。発生率は都道府県によって異なり、東京都では人口10万人あたり181.51人、青森県では人口10万人あたり2.81人となっている[7]。また、罹患率も都道府県によって異なり、岩手県と秋田県では0%、東京都では1.48%となっている。
日本におけるコロナパンデミックの第1波は 2020年3月の最終週から5月の第1週にかけて発生し、1日あたりの新たに確認された症例数は701例とピークを迎えた。第2波の発生は2020年6月最終週から継続しており、1日あたりの新規確認症例数は1,762例でピークを迎えている[7]。
日本本島の南西部に位置する広島県の2019年の推計人口は110万人であった[8]。3月の最終週から5月の第1週にかけて発生した広島でのSARS-CoV-2アウトブレイクの第1波では、162人が影響を受けた。6月最終週から 2020年8月第1週までの第2波では、204人が罹患した[9]。全体では 2020年8月3日現在、広島では合計366人の確定症例があり、3人が死亡している。広島では毎日のように確定症例が報告されているが、県内に流通するSARS-CoV-2の分子疫学に関する詳細な研究は行われていない。
本研究では,広島で検出されたSARS-CoV-2株の分子解析を行い,第1波発生時と第2波発生時の変異を比較した。
材料と方法
被験者とその臨床データ
SARS-CoV-2の第1波発生時に確認された55例の鼻腔スワブ検体と第2波発生時に確認された13例の鼻腔スワブ検体を広島県研究所から提供していただいた。患者の平均年齢は第1波が62.2±23.9歳,第2波が33.1±14.9歳であった。男性患者の割合は32.7%と69.2%であった。匿名化された臨床データはそれぞれの病院から収集された。その情報には、年齢、性別、発症日、入院日、退院日、症状、転帰、接触歴が含まれている。
SARS-CoV-2の核酸抽出と定量測定
鼻腔スワブサンプル(50μl)を、SMITEST EX-R&D(MBL、米国)を用いて核酸抽出に供した。最終ペレットを、25μlのRNaseインヒビターベースの水(サーモフィッシャーサイエンティフィック、米国)で希釈した。各サンプルについて、ヌクレオカプシド(N)遺伝子特異的プライマーNIID_2019-n-CoV-N-F2(nt29を標的とする。 125-nt29,144)およびNIID_2019-n-CoV-N-R2(標的nt29,299-nt29,280)とプローブNIID_2019-n-CoV-N-P2(nt29,222-nt29,241)(Eurofins Genomics、日本)。
GISAIDクラッドの分類
GISAIDクラッドを分類するために、SARS-CoV-2の部分ゲノムを、表1Aに示すように、7セットのプライマーを用いたポリメラーゼ連鎖反応(PCR)により、全サンプルから増幅した。第1ラウンドのPCRは、Prime Script One-Step RT- PCR kit Ver.2(タカラバイオ株式会社、滋賀県、日本)を用いて、50℃で30分間、94℃で1分間行った。その後、94℃で30秒、55℃で30秒、72℃で1分、最終伸長を72℃で7分間、それぞれ40サイクル行った。次いで、TaKaRa Ex Taq® Hot Start version(タカラバイオ株式会社、滋賀県、日本)を用いて、98℃で10秒、55℃で30秒、72℃で1分の各30サイクルにさらし、次いで72℃で7分間の最終伸長を行い、2ラウンド目のPCRを行った。最終的なPCR産物を、表1Bに示すように、対応するプライマーセットを用いて、3730xl DNAシーケンサーおよびBigDye Terminator v3.1 Cycle Sequencing Kit(Applied Biosystems, Foster City, CA, USA)を用いて、部分ゲノムシークエンシングを行った。
表1に示すように、SARS-CoV-2特異的なプライマーを使用している。特定領域の部分配列に使用したSARS-CoV-2特異的プライマー
GISAID AsiaとEuropean cladeを区別するために、European cladeではアデニン(A)、Asia cladeではグアニン(G)となるnt23403を調べた。アジアンクレードについては、アジアンクレードSのチミン(T)をnt8782,アジアンクレードVまたはOのチミン(T)をnt11083,アジアンクレードVのチミン(T)をnt26144,アジアンクレードOのチミン(T)をnt8782,nt11083,nt26144で調べ、いずれもアジアンクレードOとして調べた。続いて、欧州クラッドについては、nt25563(欧州クラッドGHのT)nt28881(欧州クラッドGRのA)それ以外を欧州クラッドGとして検討した(図1)。
図1 GISAID クレードごとの特異的な位置におけるヌクレオチドの特徴的な変化
この図は、GISAIDクラッドを分類するために使用できる、異なるヌクレオチドの特定位置でのスペクトルを示している。各色は前述の塩基(A:アデニン、G:グアニン、T:チミン)を表している。
完全長ゲノムシークエンシング
全長ゲノム配列は、次世代シークエンシング(NGS)により取得した。まず、テンプレートRNAをSuperScript IV First-Strand Synthesis System(Thermo Fisher Scientific)を用いて相補的DNA(cDNA)に翻訳した。次いで、このcDNAを、Q5 HoT START DNAポリメラーゼキットおよびプライマープールストック1および2を用いて、マルチプレックスポリメラーゼ連鎖反応(PCR)によって処理した。同一サンプルに対するプール1および2の反応におけるPCR産物をAmpureXP(1×容量)で混合および精製し、QIAseq FX DNAライブラリーキットを用いて、Illumina MiSeqシステム上でフルゲノムシークエンシングを行った。シーケンシングライブラリーの品質は、Quant-iT PicoGreen dsDNA Assay Kit(Thermo Fisher Scientific)を用いて評価した。予想されたアンプリコンサイズは250bpであった。結果として得られた断片をMiSeq Reagent Kit v3およびPhiX Control Kit v3を用いてクラスタリングし、MiSeq Control Software (MCS) v2.6.2.1,Real-Time Analysis (RTA) v1.18.54,およびBC12fastaq v2.17を用いて配列を解析した。
系統樹解析
GenBank(http://www.ncbi.nlm.gov/genbank/)およびGlobal Initiative on Sharing All Influenza Data(GISAID: …www.gisaid.org)から、合計651のSARS-CoV-2全ゲノム配列が収集された。ソース間で重複している配列をチェックし、手動で除� Evolutionary Genetics Analysis(MEGA)バージョン7.0 [10]を用いて、隣接結合法による進化解析を行った。
広島県産SARS-CoV-2株の分子特性と変異の検討
GenBankの参照株(MN908947)を用いて、本研究で得られた8つのSARS-CoV-2全ゲノム配列をMEGA v7.0のMUSCLE多重配列アライメントアルゴリズムを用いてアラインメントした後、アミノ酸配列に変換した。全ゲノム配列において、ORF1a、ORF1b、S、ORF3a、E、M、ORF6,ORF7a、ORF8,N、ORF10の各断片において、参照株とSARS-CoV-2との間で変異を比較した。変異率は、サンプル採取日に応じて算出し、1年あたり、1kbあたりの変異の単位に換算した。
GISAIDから検索した日本全国の株と本研究のORF8欠失株のスクリーニング、同定および関連性
前述の類似部分シーケンシング法を用いて、全検体のORF8領域を標的とするプライマーhCoV-ORF8セット(表1)を用いて、広島の残り60検体のSARS-CoV-2検体を同定し、スクリーニングした。その後、GISAIDを用いて日本全国の類似ORF8欠失変異体を検索し、本研究と報告株との間の分子パターンを検討した。
倫理的配慮
本研究は広島大学倫理委員会(E2124)の承認を得た。すべての参加者は研究開始時に書面によるインフォームドコンセントを提供した。すべての手順は、ガイドラインとヘルシンキ宣言を厳守した。
研究結果
SARS-CoV-2の定量的測定
RT-qPCRによるアジア型のウイルス負荷量は5.45×105 copies/mlであった。第1波のヨーロッパ型は2.18×102~1.42×108 copies/ml、第2波のヨーロッパ型は7.85×101~9.16×106 copies/mlであった(図2)。
サムネイル
PPTPowerPointスライド
PNGより大きな画像
TIFFオリジナル画像
図2 広島における SARA-CoV-2 のウイルス力価の変動。
GISAIDクラッドに分類された68検体におけるSARS-CoV-2の定量測定。2 つは GISAID クレードサブタイプ G、GH、または GR に分類できなかったもの、1 つはアジア株 O(緑の点)65 つはヨーロッパ株 GR(青の点)であった。タイターは、各サンプルのサイクル閾値に対してプロットされた。
doi.org/10.1371/journal.pone.0246383.g002
広島におけるSARS-CoV-2のGISAIDクラッド(遺伝子型
GISAIDクラッドを分類するための部分シーケンシングには68検体中66検体(66検体)が利用可能であった(97.1%)が、2検体は検出不能であった(2.9%)。これらの未検出サンプルはいずれもウイルス負荷が低く、それぞれ2.42×102,2.18×102 copies/mlであった(図2)。3月に1週間間隔で採取した2検体のうち,最初の分離株はアジア型のO型,後者はヨーロッパ型のGR型であった。その後 2020年4月から 2020年8月までの間に第1波と第2波のサンプルを採取したが、いずれもヨーロッパ型GRであった。
系統樹解析
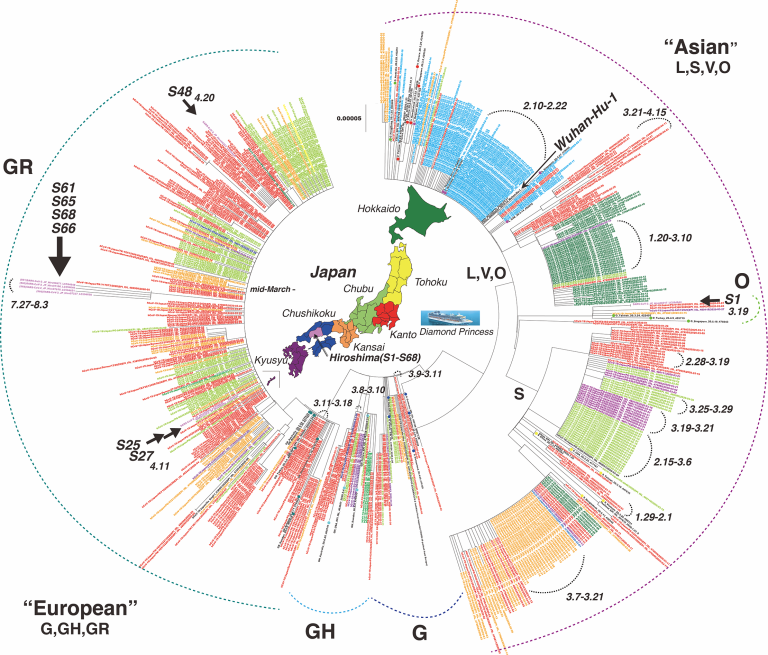
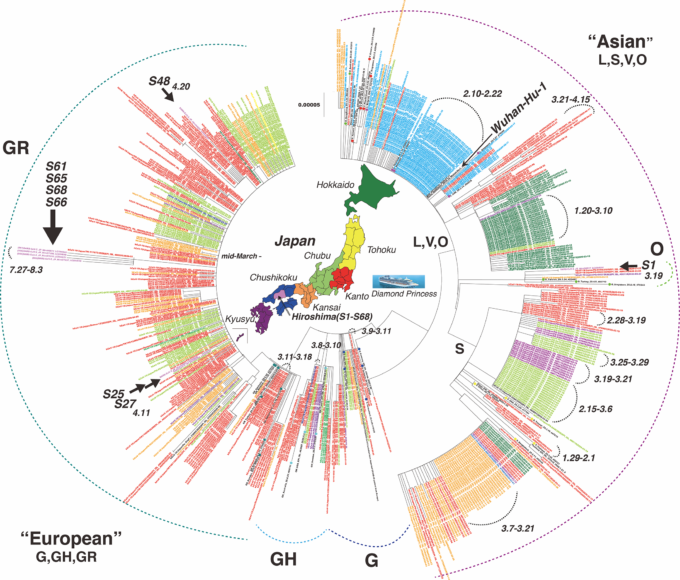
日本国内に分離された全分離株と、GISAIDおよびGenBankから検索した対照株を用いて、日本の地域別に系統樹を構築し、北から北海道、東北、関東、中部、関西、中四国(当初は中国と四国を別々に分類していたが、本研究では統合した)九州に分類した(図3)。日本では、SARS-CoV-2のアジア型が2020年1月から4月にかけてパンデミックし、中部・関西・北海道やダイヤモンド・プリンセス・クルーズの船内で小規模なクラスターが一過性に多く発生した。これらの地域では、様々なタイプのアジア系クラッドが循環していた。その後、3月第1週目からヨーロッパ型のG型、GH型のクラスターが出現した。3月中旬以降、ヨーロッパ型GRのクラスターが全国で同時に多数観測され、日本ではこの株が優勢となった。広島では、8つのフルゲノムを解析したところ、1つ(S1. 広島市では,SARS-CoV-2_JP_Hiro66017_LC584644株のうち1株(S1:SARS-CoV-2_JP_Hiro66017_LC584644)がアジア型O型であった。残りはヨーロッパ型GRであり,S25(SARS-CoV-2_JP_Hiro99262_LC594645),S27(SARS-CoV-2_JP_Hiro99268_LC594646),S48(SARS-CoV-2_JP_Hiro94253_LC594647)は第1波で,S61(SARS-CoV-2_JP_Hiro96977_LC594648)は第2波で循環していた。第二波では、S65(SARS-CoV-2_JP_Hiro97166_LC594649)S66(SARS-CoV-2_JP_Hiro97237_LC594650)S68(SARS-CoV-2_JP_Hiro97618_LC594651)が流通していた(図3)。第2波のGR株は11-15塩基変異を持つ株が独立して出現した。
図3 日本におけるSARS-CoV-2の系統樹(広島から分離された8株のSARS-CoV-2を含む)。
この系統樹には、世界各国の対照株52株、GISAIDおよびGenBankから検索した日本の522株、および本研究で分離された8株のSARS-CoV-2が含まれている。対照株を色付きの点で示した。日本から分離された菌株は、日本を7つの地域に分けた日本地図の色に対応する色文字で示されている。空色は、ダイヤモンド・プリンセス号のクルーズからの菌株を表している。また、当グループのSARS-CoV-2の8株は紫色で表示されており、サンプル番号も表示されている。ツリーはMEGA7を用いた隣接結合法により作成した。最終的なデータセットには29038個の位置が含まれている。
doi.org/10.1371/journal.pone.0246383.g003
広島のSARS-CoV-2株の突然変異とその後のアミノ酸変化
SARS-CoV-2の8株のうち,参考株(MN908947)との比較では,44部位に7種類の変異が認められ,59.1%(26/44)が非同義変異,残り(40.9%)が同義変異であった。これら44部位のうち、ORF1aと1bは27部位(61.4%)ヌクレオカプシド(N)は5部位(11.4%)スパイク(S)は4部位(9.1%)の変化を含んでいた(図4)。
図4 広島から分離されたSARS-CoV-2分離株の全ゲノムにおけるヌクレオチド突然変異とアミノ酸置換。
この図は、広島から分離された8つのSARS-CoV-2分離株で確認されたヌクレオチド突然変異とその結果のアミノ酸置換を示している。色はヌクレオチドまたはアミノ酸を表し、各パネルの下の凡例に従っている。ゲノム領域は上段の全ゲノムモデルで示し、点状変異は色の変化で示している。
doi.org/10.1371/journal.pone.0246383.g004
アジア型O株では7つのヌクレオチド変異が検出され、そのうち5つはORF1aにあり、SまたはNにそれぞれ1つずつあった。第1波のヨーロッパ型GR株はすべて、8つの異なる部位で同様の突然変異が認められたが、そのうちの1つはノンコーディング領域(C241T)2つはORF1a(C313T、C3037T)1つはORF1b(C14408T)1つはS(A23403G)3つはN(G28881A、G28882A、G28883C)であった。さらに、図3に示すように、特定の株の様々な部位で、いくつかの離散的な突然変異が見られた。全体では、第一波のヨーロッパ型株では15部位に32の変異が見られた:ORF1abに50%(16/32)Nに31.3%(10/32)Sに9.4%(3/32)ORF3a、ORF10,Mにそれぞれ3.1%(1/32)であった(図4)。
前述の8つの類似した変異は、第2波の全ヨーロッパ株でも発生していた。さらに、別の類似の変異パターンは、これらの株間で6つの異なる部位で共有されていた:ORF1a、ORF1b、N、および非コード領域におけるそれぞれ3つの部位(T4346C、C9286T、C10376T)1つの部位(C14708T)1つの部位(C28725T)および1つの部位(G29692T)であり、そのうちの4つは非同義であった。さらに、特定の株では、16個の離散的なヌクレオチド変化も様々な部位で見られた(図3)。このように、第2波のヨーロッパ株では、30部位で72の突然変異があった:ORF1abに59・7%(43/72)Nに22・2%(16/72)Sに8・3%(6/72)非特異的領域に5・5%(4/72)ORF8に2・8%(2/72)Mに1・4%(1/72)であった(図3)。特筆すべきは、ヨーロッパの7株すべてがN遺伝子のG28881A、G28882A、G28883Cのトリプル変異を共有していたことである。推定変異率は38.4ヌクレオチド/年、1部位あたり1.16〜1.87×10-3塩基置換/年であった。第1波内の突然変異の数は1変化から7変化までの範囲であったが、第2波では8変化であった。
アジア株Oでは4つのアミノ酸置換が認められた:3つ(218. アルギニンからグリシンへ 2016.スレオニンからリジンへ、3606. ORF1aには3つ(218:アルギニン→グリシン 2016:スレオニン→リジン、3606:ロイシン→フェニルアラニン)Nには1つ(13:プロリン→ロイシン)のアミノ酸置換が見られ、第1波と第2波のいずれのヨーロッパ株でも4つの同様のパターンのアミノ酸置換が見られた。ORF1bは1つ(214:プロリン→ロイシン)スパイクは1つ(614:アスパラギン酸→グリシン)ヌクレオカプシドは2つ(2020:アルギニン→リジン、203:グリシン→アルギニン)であった。特筆すべきことに、第2波からの4つのヨーロッパ株はすべて、4つの類似したアミノ酸置換パターンを共有していた(1361:セリンからプロリンへ、3371:グリシンからアルギニンへ。ORF1aではプロリンからセリン、ORF1aでは314. ORF1bではアラニンからバリンへ、151. ヌクレオカプシド中のプロリン→リジン)。) さらに、残りのアミノ酸置換は、特定のヨーロッパ株では個々に様々な部位に分布していた。アミノ酸置換の大部分は非構造タンパク質(ORF1a、1b)に見られ、全置換の57.7%を占めた。構造タンパク質の中では、N(15.4%)とS(11.5%)が全欧州株で最も多くのアミノ酸置換部位を示しており、その他にもORF3aとMにも置換が見られた(図4)。
欧州株GRのD614G変異体
欧州株GRのスパイク領域にはD614Gのアミノ酸変化が見られ、このアミノ酸変化は、非コード領域のC241Tの無声変異、ORF1aのC313TおよびC3037T、ORF1bのC14408T(P214L)の非同義変異、およびNのアミノ酸変化(R202KおよびG203R)をもたらすG28881A、G2882AおよびG28883Cの三重変異を伴うものであった。特筆すべきことに、スパイク領域は、図3に示すように、3つのアミノ酸変化をもたらす4つのヌクレオチド変異を含んでいた。さらに、S65のD614G変異体は同じ領域にT22020C変異を有し、S66はスパイク領域にC21789T変異を有しており、これらの変異はいずれもアミノ酸の変化(153:メチオニンからスレオニンへ、76:スレオニンからイソロイシンへ)をもたらした(図4)。
SARS-CoV-2_JP_Hiro97327_LC594650における一塩基挿入
広島での第2波発生時に採取された試料S66から 1つのヌクレオチドの挿入が見つかった。このヌクレオチド挿入は、363ヌクレオチド(121アミノ酸)を含むORF8のnt27906とnt27907の間に発生したものと推定された。この単一ヌクレオチドの挿入は、図4に示すように、フレームシフトを引き起こし、その結果、ORF8は消失した。この突然変異体のORF8は、野生型では通常の121アミノ酸の長さではなく、20アミノ酸に短縮されていた。その結果、野生型と同じ長さのアミノ酸は初期の6アミノ酸のみで、残りのアミノ酸はフレームシフトにより異なっていた(図5)。図5)2021年1月12日時点でGISAIDに提出された日本全国のSARS-CoV-2株9,844株の中で、図5に示すように、類似のヌクレオチド挿入(1つは一塩基挿入、2つは二塩基挿入、1つは三塩基挿入)を有し、その後のフレームシフトと早期に停止コドンが出現した変異株は4株のみであった。この患者は60歳男性で、ORF8欠失変異株に感染しており、症状は軽度で、13日間の入院で治癒した。
図5 フレームシフトとORF8欠失をもたらすオープンリーディングフレーム(ORF8)への一塩基挿入。
上部の枠線は、次世代配列(NGS)で得られた全ゲノム配列を用いて、野生型S68と変異体S66のORF8を比較したもの。サンプルではORF8の消失が観察された。さらに、GISAIDから検索した類似の変異パターンを持つ日本のSARS-CoV-2株4株もこの図に含まれている。この小さなピークは、nt27906とnt27907の間にシングルとダブルのチミン(T)ヌクレオチドが挿入されたSARS-CoV-2変異体の混合クローニングによるもので、大きなドミナント波はシングルのチミン挿入の優性変異体を、小さなピークはダブルのチミン挿入の劣性変異体を表している。
doi.org/10.1371/journal.pone.0246383.g005
議論
本研究では、最初に確認された症例が発生した日から 2020年8月3日までの間に広島県で流通しているSARS-CoV-2株の分子的特徴と変異パターンを明らかにした。当時、広島県では全国で40,244人の新たな確定症例が報告されていたのに対し、広島県では366人となっている。当時の広島県の患者数は0.9%であったことになる。このうち,広島県の研究所から提供された68検体を政府の研究所で使用し,第一段階としてリアルタイムPCRによるウイルスの定量とGISAID分類のための領域特異的パーシャルシークエンシングを行った。その後、8サンプルを無作為に選択し、NGSフルゲノムシークエンシングを実施した。2021年1月14日現在,日本国内の確定症例298,172例のうち9,885株(3.3%)がGISAIDに登録可能である。今回の調査では366例中8例(2.2%)が登録されており,日本全体とほぼ同等である.興味深いことに,第1波で採取された68検体のうち,第1波で採取された1検体のみがアジア型であり,第1波と第2波の両方で採取された残りの検体はGISAID clade GRであった。系統樹解析により、日本におけるSARS-CoV-2の進化を説明することができたが、感染の起源やカスケードの特定は困難であった。2020年1月から4月にかけて、ダイヤモンド・プリンセス・クルーズでは、北海道、関東、関西、中部、九州地方で多数の症例群が発生した。3月第2週には、欧州株G、GH、GRが日本に入ってきて、主に関東地方で見られた。3月中旬以降、ヨーロッパ株GRが日本全国で優勢となった。今回の研究で得られた完全長ゲノムの結果から,7株の欧州株はすべて遺伝子型GR(具体的にはD614G変異株)であることが明らかになった。日本では発生時の対応が急務となっており,3月下旬に警戒レベルが引き上げられたが,今回の研究では,アジア株が日本全体に優勢であることが明らかになった。その結果,コロナウイルス対策が強化された後は,アジア株の発生は少なく,日本では報告されなくなった。このことから,アジア株はヨーロッパ株に比べて複製率が低く,感染性が低いことが示唆された。そのため,厳しい公衆衛生対策によって感染の連鎖を断ち切ることができたと考えられる.一方,欧州株はアジア株に比べて感染力,病原性,複製率が高く,特にD614G変異株の方が感染力が高いことが示された[11].また,ヨーロッパ株はアジア株に比べてウイルス負荷が高く,複製効率が高いことが示唆され,GR型のヨーロッパ株7株はすべて高い変異率を示した。厳格な公衆衛生対策のもと,一時的に新規症例は減少したが,この間にSARS-CoV-2の進化が起こった。その後,病原性,感染性,複製性,伝播性に差のある変異体が新たに出現し,新たな感染症が発生した。
広島で発生したSARS-CoV-2株の全蛋白質のうち、アジア株およびヨーロッパ株では、特異的な一塩基変異によるアミノ酸置換が認められた。SARS-CoV-2はRNAウイルスであるため、突然変異率が高く、時間の経過とともに進化を続け、複製、感染性、伝達性、病原性、免疫原性に影響を及ぼす可能性がある[12]。我々の研究でも、第1波と第2波の間の突然変異率は34.9~40.6ヌクレオチド/年であり、これは1部位あたり1.17~1.36×10-3塩基置換/年に相当することが示された。この突然変異率はC型肝炎ウイルスの突然変異率よりも低い[13]。最近の研究では、可変部位におけるSARS-CoV-2の変異率は、1年あたり3.5×10-3変化/部位であり、我々が測定した値よりも高かったことが報告されている[14]。興味深いことに、すべてのヨーロッパ株は特定の部位で同様の変異パターンを共有しており、それに伴うアミノ酸置換は構造タンパク質ではなくポリタンパク質のOFR1abに多く見られた。Md Rafiqul Islamらもまた、ほとんどのヌクレオチド変異がポリタンパク質で発生しており、ORF1aが最も頻繁に変異した領域であると報告している[15]。我々の知見は、これまでの報告と一致している。ORF1aと1bの変異は、ポリプロテインの処理とウイルス複製の制御に関与するメインプロテアーゼ(Mpro)とレプリカーゼに必須であるため、ウイルスの複製効率に影響を与える可能性がある[16, 17]。
特筆すべきは、広島産のGR株7株すべてにおいて、ヌクレオカプシドタンパク質にアミノ酸の変化をもたらすトリプル変異(G28881A、G28882A、G2888C)を発見したことである。さらに、第2波のGRの4株はC28725Tで同じ変異を持っていた。ヌクレオカプシドタンパク質は、RNA複製、転写、ゲノムパッケージングに重要な役割を果たしている[18, 19]。ゲノム、特に構造タンパク質の変異の頻度が高いため、形態学的構造、病原性、感染力、複製効率に違いのあるウイルスの新株の出現が促進されている。この現象は、ワクチン候補や薬剤の開発を複雑にしている。さらに、我々は、ORF1a の無声変異 C241T、C313T、C3037T、S の P214L アミノ酸置換、N の R202K と G203R の二重変異を伴う D614G 変異体の優勢型を同定した。最近の研究では、D614G 変異体が世界的に優勢型として流通していることが示唆された。D614G変異体の感染性と伝播性の変化を考慮すると,我々の知見は,SARS-CoV-2の臨床管理に重要な示唆を与えるだけでなく,将来的に予防戦略と制御戦略をどのように変更するかについても重要な示唆を与えるものである.スパイク領域には、D614G変異に加えて、S65のM153TとS66のT76Iという2種類の変異が含まれていた。スパイクタンパク質は、ウイルスの侵入に不可欠な役割を果たし、宿主細胞の受容体と相互作用する重要な表面糖タンパク質である[21, 22]。したがって、スパイク領域の高変異は、ワクチンや抗体ベースの治療薬の開発を妨げる可能性がある。
もう一つの注目すべき発見は、nt27906と27907の間の一塩基挿入の検出であり、結果としてORF8タンパク質の欠失をもたらす。382ヌクレオチド欠失を有するSARS-CoV-2変異体が、シンガポール、バングラデシュ、オーストラリア、スペインで報告されている[23-25]。このタイプの欠失はORF8調節配列を除去し、ORF8転写を除去する;この変異体は感染性の軽度化および急性期のサイトカイン放出の低レベルと関連している[25]。SARS-CoV-2のORF8の役割は依然として不明であるが、ある研究では、ORF8は抗原提示を阻害し、細胞傷害性Tリンパ球(CTL)によるウイルス感染細胞の認識と排除を減少させることが示唆されている[26]。我々の研究では、ORF8に一塩基挿入すると、その後のアミノ酸のフレームシフトと停止コドンの早期出現が起こり、それに伴ってORF8が消失することが示唆された。その結果、8つの全長ゲノムと60の部分ゲノムのうち、1例のみが発見された。また,GISAIDに投稿された全日本9844株のうち,類似変異株は4株のみであった。このことから,ORF8欠失変異株の感染は,第2波の間に一時的に発生したものと考えられ,寿命が短く,ウイルス複製が少なく,感染が少ないことが示唆された。この知見は、最近発表された報告[24]と一致している。
我々の研究にはいくつかの限界があった。サンプル量が不足しているため、すべてのサンプルでフルゲノムシークエンシングを行うことは不可能である。しかし、全ゲノム配列解析のために8つのサンプルをランダムに盲検選択することで選択バイアスを低減したが、ランダムエラーを排除することはできない。サンプル数は少ないが、広島のSARS-CoV-2株のフルゲノム解析は日本でも初めての報告であり、今後の分子生物学的サーベイランスのベースラインとして、またワクチンや免疫療法、診断ツールの開発の糸口となる貴重なデータと知見である。また、将来的には、突然変異の結果と患者の臨床転帰、感染性、病原性、ウイルス因子との関連性を明らかにするための大規模な研究が必要である。本研究では、系統、分子特性、およびそれらの変異パターンに焦点を当てた研究を行ったため、同定した変異に起因する詳細な形態変化や生物学的機能の変化については検討していない。
以上の結果から,広島で最近出現したSARS-CoV-2株はヨーロッパ型GR系統であることが明らかになった。このことから、この地域におけるSARS-CoV-2株の感染性と伝播性の変化には、伝播カスケードと高い変異率が関係している可能性が示唆された。また、D614Gの優勢な変異とORF8欠失の新形態は、広島でのSARS-CoV-2の局所発生におけるウイルス因子の役割の手がかりを提供するものであった。経時的な分子特性や変異パターンを理解することは、臨床現場や公衆衛生対策の設計、ワクチンや治療薬の開発に役立つと考えられる。今後は,循環株のさらなる研究と分子レベルでのサーベイランスが必要である.また、新規コロナウイルス感染症の臨床管理を最適化する必要がある。