Contents
Mendelian randomization analysis identified genes pleiotropically associated with the risk and prognosis of COVID-19
www.ncbi.nlm.nih.gov/pmc/articles/PMC7698677/
2020 Nov 28
要旨
目的
COVID-19は世界的に大規模なパンデミックを引き起こした。COVID-19患者では、疾患行動にかなりのばらつきが見られる。これまでのゲノムワイド関連研究により、COVID-19のリスクや予後に関与する遺伝子変異が同定されているが、その根底にある生物学的解釈はほとんど明らかにされていない。
研究方法
本研究では、サマリーデータに基づいたメンデルランダム化(SMR)法を用いて、重症呼吸器確認済みCOVID-19および入院COVID-19を含むCOVID-19のリスクおよび様々な転帰と多元的に関連する遺伝子を同定し、その遺伝子を用いてCOVID-19のリスクおよび転帰を解析した。
結果
血液中では、IFNAR2をタグ付けしたILMN_1765146とILMN_1791057の2つのプローブが、入院したCOVID-19と多値関連を示した(β[SE]=0.42 [0.09], P=4.75×10-06,β[SE]=0.48 [0.11], P=6.76×10-06,それぞれ)。血液と肺の両方で多重検査を補正しても他のプローブは有意ではなかったが、上位5つのプローブでタグ付けされた複数の遺伝子は炎症や抗ウイルス免疫に関与しており、PON2やHPS5のような他のいくつかのタグ付けされた遺伝子は血液凝固に関与していた。
結論
本研究では、COVID-19の感受性や予後に関与している可能性のあるIFNAR2やその他の遺伝子を同定した。これらの知見は、COVID-19におけるサイトカインストームや静脈血栓塞栓症のメカニズムをよりよく理解するための重要な手がかりとなり、COVID-19の効果的な治療のための潜在的な治療標的を提供する。
キーワード:コロナウイルス疾患2019,IFNAR2,遺伝子発現量的形質遺伝子座、要約メンデル無作為化
序論
重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)を原因とするコロナウイルス病2019(COVID-19)は、世界的に大規模なパンデミックを引き起こし、公衆衛生に深刻な脅威をもたらしている(1,2)。2020年8月7日現在、全世界で1,930万人以上の確認症例があり、総死亡者数は71万9,830人を超えている(3)。SARS-CoV-2は、主に呼吸器の飛沫や密接な接触を介して拡散する高病原性・感染性のコロナウイルスである(4)。COVID-19がもたらす世界的な課題に対処するためには、COVID-19の蔓延を抑制するための解決策を模索し、効果的な治療法を模索することが最も重要である。したがって、COVID-19の基礎となる病理学的メカニズムをさらに明らかにすることが急務である。
COVID-19患者は、疾患行動にかなりのばらつきがある。最近、COVID-19の診断や予後に関連する遺伝子変異を同定するためにゲノムワイド関連研究(GWAS)が行われているが(5,6)それらの知見の生物学的解釈はまだほとんど明らかになっていない。これまでの研究では、GWASで検出された形質関連遺伝子変異の約88%がゲノムの非コード領域に存在し、遺伝子発現に制御機能を持つ可能性があることが明らかにされている(7)。したがって、COVID-19研究の文脈では、COVID-19の感受性や発症に多元的/因果関係のある遺伝子の発現を探索することが重要である。
従来の無作為化比較試験(RCT)とは異なり、遺伝子変異を無作為化の代理として用いるメンデル無作為化(MR)は、曝露(例えば遺伝子発現)がアウトカム(例えばCOVID-19の感受性)に与える多面的/潜在的な因果関係を探索するための有望なツールである(8)。MRは、従来の関連研究でよく見られる交絡や逆因果関係を最小化し(8,9)、心血管疾患、BMI、関節リウマチなどの様々な表現型と多元的/潜在的に因果関係にある遺伝子発現部位やDNAメチル化座を同定することに成功している(10,11,12,13)。
本論文では、COVID-19のGWASデータとcis-eQTL(発現量的形質遺伝子座)データを統合したサマリーデータベースMR(SMR)法を適用し、COVID-19のリスクや予後に好転的/潜在的に因果関係にある遺伝子の優先順位付けを行った。
研究方法
データソース
eQTLデータ
SMR解析では、遺伝子発現のための器具変数(IV)としてcis-eQTL遺伝子変異体を用いた。血液と肺の遺伝子発現については、別々に SMR 解析を行った。血液については、2765人の参加者を含むCAGE eQTL要約データ(14)を用いた。肺については、V7リリースのGTEx eQTL要約データ(15)を使用し、278名の参加者が含まれていた。血液および肺のeQTLデータは、https://cnsgenomics.com/data/SMR/#eQTLsummarydata からできる。
COVID-19のGWASデータ
GWAS要約データは、COVID-19宿主遺伝学イニシアティブ(6)から提供され、https://www.covid19hg.org/results/ でできる。重症呼吸器確認COVID-19,COVID-19および入院COVID-19を含む3つの表現型を検討した。表現型の定義の詳細は、表S1に記載されている。対照群は、特定の表現型を持たない一般集団からの被験者、予測または自己申告によりCOVID-19陰性であった被験者、または入院せずにCOVID-19を発症した被験者であり、合計5つの比較を行った:重度呼吸器確認済みCOVID-19(n=536)対集団(n=329,391,以下重度COVID-19)COVID-19(n=6,696)対集団(n=1073072,以下重度COVID-19)COVID-19(n=6,696)対集団(n=1073072,以下重度COVID-19)。人口(n=1073072;以下COVID-19)、COVID-19(n=3,523)対COVID-19陰性(n=36,634;以下COVID-19陰性)、入院COVID-19(n=3,199)対COVID-19陰性(以下COVID-19陰性)。母集団(n=897,488;以下、入院COVID-19)入院COVID-19(n=928)vs.入院COVID-19(n=2,028;以下、入院COVID-19)と入院なしCOVID-19(n=2,028;以下、入院COVID-19)とを比較した。また、重症呼吸器確認済みCOVID-19については、Severe COVID-19 GWASグループ(5)のGWAS要約データを用いてSMR解析を繰り返した(以下、重症COVID-19 NEJM)。本研究では、重度の呼吸器性COVID-19が確認された患者1,160人と、COVID-19を対照としない一般集団からの参加者2,205人が含まれていた(表1)。本研究における重症呼吸器性COVID-19の定義は、COVID-19宿主遺伝学イニシアティブで定義されたものとは異なっていた(表S1)。GWAS要約データは、https://ikmb.shinyapps.io/COVID-19_GWAS_Browser/ でできる。
表1 SMR解析におけるGWASデータおよびeQTLデータの基本情報※
| 表現型 | ケース | コントロール | 血液のSMR分析におけるプローブの数 | 肺のSMR分析におけるプローブの数 |
|---|---|---|---|---|
| 重度のCOVID-19 | 536 | 329,391 | 8,457 | 5,531 |
| 重度のCOVID-19NEJM # | 1,610 | 2,205 | 8,431 | 5,467 |
| COVID-19(新型コロナウイルス感染症)(#文字数制限がない場合、初出時にかっこ書きを追加 | 6,696 | 1,073,072 | 8,499 | 5,653 |
| COVID-19ネガティブ¶ | 3,523 | 36,634 | 8,499 | 5,628 |
| 入院COVID-19 | 3,199 | 897,488 | 8,495 | 5,628 |
| COVID-19入院なし§ | 928 | 2,028 | 8,492 | 5,625 |
⁎GWASの要約データは、特に断りのない限りCOVID-19宿主遺伝学イニシアティブ(6)から提供されたものである。血液(14)と肺(15)のeQTLデータは、https://cnsgenomics.com/data/SMR/#eQTLsummarydata でできる。
#¶GWAS要約データは、重症COVID-19 GWASグループ(5)から提供された。
¶コントロールは、非予測および非自己報告のCOVID-19である。
§対照は入院なしのCOVID-19であり、検査室で確認されたCOVID-19または自己報告されたCOVID-19を含む。
COVID-19,コロナウイルス病2019;SMR、要約データに基づくメンデル無作為化
SMR分析
MR は、cis-eQTL を IV、遺伝子発現を曝露、3 つの表現型のそれぞれをアウトカム(異なる対照群との比較)とした。MR 解析は SMR ソフトウェアに実装されている方法で行った。SMR 法の詳細については、以前に発表された論文(10)に報告されている。簡単に言えば、SMRはMRの原理を適用して、遺伝子発現とある遺伝子座で共有された、因果関係のある可能性のある変異に起因する形質との間の多面的な関連性を検定するために、GWASとeQTLの要約統計量を共同で解析する。観察された関連性における連鎖の存在を評価するために、従属尺度の異質性(HEIDI)検定を実施した。帰無仮説の棄却(すなわち、P HEIDI<0.05)は、観察された関連性が、互いに高い連結性不平衡にある2つの異なる遺伝的変異に起因する可能性があることを示している。我々は、SMRのデフォルト設定(例えば、P eQTL <5×10-8,マイナー対立遺伝子頻度[MAF]> 0.01,最上位の関連eQTLと非常に強い連結不平衡[LD、r2 > 0.9]のSNPを除去し、最上位の関連eQTLと低いLDまたはLDにないSNPを除去する[r2 <0.05])を採用し、多重検定を調整するために偽発見率(FDR)を使用した。
転写物のアノテーションは、AffymetrixエクソンアレイS1.0プラットフォームに基づいた。仮定転写産物を機能的にアノテーションするために、機能アノテーションツール「Metascape」(16)を用いて、血液と肺のトップタグ付き遺伝子を別々に機能的エンリッチメント解析を行った。仮定遺伝子に対応する遺伝子記号(P<0.05)は、遺伝子オントロジー(GO)と京都遺伝子・ゲノム百科事典(KEGG)のエンリッチメント解析の入力として使用した。
データのクリーニングと統計・バイオインフォマティクス解析は、R version 4.0.0 (www.r-project.org/)、PLINK 1.9 (www.cog-genomics.org/plink/1.9/)、SMR (cnsgenomics.com/software/smr/)を用いて行った。
結果
まとめデータの基本情報
症例数と対照群の数は、異なる分析の間で劇的に変化した。プローブの数は、血液と肺の分析では、それぞれ約8,500個と5,500個であった。詳細は表1に示した。
血液中のSMR解析
各表現型の上位 5 つのプローブの情報を表2
に示した。COVID-19宿主遺伝学イニシアティブのGWAS要約データを用いて、入院したCOVID-19とプリーオトロピックな関連を示す2つのプローブ、ILMN_1765146およびIFNAR2をタグ付けしたILMN_1791057を同定した(β [SE]=0.42 [0.09]、P=4.75×10-06およびβ [SE]=-0.48 [0.11]、P=6。 42 [0.09], P=4.75 × 10-06およびβ [SE]=-0.48 [0.11], P=6.76 × 10-06, それぞれ)他の表現型については多重検定補正後には有意な多値化関連は認められなかった(図1 , 図S1-5)。また、重度のCOVID-19 NEJMとCOVID-19の解析においても、同じ2つのプローブが上位5つのプローブに含まれていたが、多重検査補正後の統計的有意性には至らなかった(表2)。さらに、TRIM5をタグ付けした複数のプローブが、重度のCOVID-19 NEJMおよび血中入院COVID-19の解析において、トップ5のプローブの中に含まれていることがわかった。
表2 血液のSMR解析で同定された上位5つのプローブ
| 表現型* | CHR | プローブID | 遺伝子 | トップeQTL | P eQTL | P GWAS | ベータ | SE | P SMR | P HEIDI | N SNP |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 重度のCOVID-19 | 7 | ILMN_1751051 | C7orf25 | rs630191 | 2.00× 10−112 | 1.36× 10−04 | -0.39 | 0.10 | 1.68× 10−04 | 1.55× 10−01 | 20 |
| 5 | ILMN_1654685 | MCTP1 | rs306578 | 1.11× 10−168 | 1.89× 10−04 | 0.35 | 0.09 | 2.16× 10−04 | 2.72× 10-01 | 20 | |
| 15 | ILMN_1778734 | FAN1 | rs2240101 | 3.65× 10−122 | 1.85× 10−04 | 0.42 | 0.11 | 2.23× 10−04 | 6.54× 10-02 | 20 | |
| 7 | ILMN_1749432 | MRPL32 | rs601057 | 1.82× 10−33 | 1.60× 10−04 | -0.75 | 0.21 | 3.14× 10−04 | 1.24× 10−01 | 20 | |
| 15 | ILMN_2152178 | FAN1 | rs11070620 | 7.48× 10−65 | 3.86× 10−04 | 0.55 | 0.16 | 5.12× 10−04 | 7.02× 10-02 | 20 | |
| 重度のCOVID-19NEJM # | 21 | ILMN_1765146 | IFNAR2 | rs2252639 | 5.57× 10−44 | 4.23× 10−05 | 0.69 | 0.18 | 8.54× 10−05 | 7.52× 10-02 | 15 |
| 21 | ILMN_1791057 | IFNAR2 | rs2252639 | 5.21× 10−34 | 4.23× 10−05 | -0.78 | 0.20 | 1.04× 10−04 | 1.61× 10-01 | 16 | |
| 11 | ILMN_1704972 | TRIM5 | rs35253098 | 5.68× 10−85 | 7.93× 10−05 | -0.45 | 0.12 | 1.09× 10−04 | 7.86× 10−01 | 20 | |
| 16 | ILMN_1716704 | NLRC5 | rs1684577 | 2.30× 10−35 | 3.80× 10−04 | 0.71 | 0.21 | 6.38× 10−04 | 5.00× 10-02 | 20 | |
| 11 | ILMN_2404665 | TRIM5 | rs34080219 | 3.47× 10-17 | 2.32× 10−04 | -1.03 | 0.30 | 7.44× 10−04 | 1.15× 10−01 | 20 | |
| COVID-19(新型コロナウイルス感染症)(#文字数制限がない場合、初出時にかっこ書きを追加 | 20 | ILMN_1681591 | PTPN1 | rs117437913 | 4.76× 10−56 | 1.29× 10−04 | -0.21 | 0.06 | 1.99× 10−04 | 6.23× 10−03 | 20 |
| 21 | ILMN_1765146 | IFNAR2 | rs2252639 | 5.57× 10−44 | 2.68× 10−04 | 0.20 | 0.06 | 4.23× 10−04 | 1.19× 10−01 | 15 | |
| 21 | ILMN_1791057 | IFNAR2 | rs2252639 | 5.21× 10−34 | 2.68× 10−04 | -0.23 | 0.07 | 4.81× 10−04 | 1.67× 10−01 | 17 | |
| 5 | ILMN_1757106 | 3月6日 | rs2589667 | 4.49× 10−155 | 5.08× 10−04 | -0.10 | 0.03 | 5.66× 10−04 | 6.97× 10−01 | 20 | |
| 16 | ILMN_1681945 | ITGAD | rs2454911 | 6.71× 10−13 | 8.85× 10−05 | 0.41 | 0.12 | 5.79× 10−04 | 1.10× 10−01 | 20 | |
| COVID-19ネガティブ¶ | 1 | ILMN_2123743 | FCER1G | rs2070901 | 1.89× 10−230 | 2.16× 10−05 | -0.15 | 0.04 | 2.54× 10−05 | 5.43× 10−01 | 20 |
| 15 | ILMN_1783956 | ATP8B4 | rs7172615 | 9.53× 10−92 | 1.86× 10−04 | -0.20 | 0.05 | 2.37× 10−04 | 3.06× 10−01 | 20 | |
| 3 | ILMN_1654612 | ZNF589 | rs11720622 | 2.51× 10−52 | 3.24× 10−04 | -0.27 | 0.08 | 4.66× 10−04 | 9.38× 10-02 | 20 | |
| 19 | ILMN_1760858 | RAB8A | rs35848422 | 1.99× 10−89 | 4.38× 10−04 | -0.19 | 0.05 | 5.33× 10−04 | 6.88× 10−01 | 15 | |
| 22 | ILMN_1672128 | ATF4 | rs710192 | 5.43× 10−59 | 6.49× 10−04 | -0.24 | 0.07 | 8.46× 10−04 | 3.05× 10-01 | 20 | |
| 入院COVID-19 | 21 | ILMN_1765146 | IFNAR2 | rs2252639 | 5.57× 10−44 | 1.26× 10−06 | 0.42 | 0.09 | 4.75× 10−06 | 1.48× 10−03 | 15 |
| 21 | ILMN_1791057 | IFNAR2 | rs2252639 | 5.21× 10−34 | 1.26× 10−06 | -0.48 | 0.11 | 6.76× 10−06 | 1.68× 10−01 | 17 | |
| 7 | ILMN_1770030 | C7orf43 | rs6975031 | 1.96× 10−34 | 1.58× 10−04 | 0.40 | 0.11 | 3.06× 10−04 | 3.52× 10-02 | 20 | |
| 14 | ILMN_1675391 | SLC24A4 | rs61977314 | 1.84× 10−69 | 4.57× 10−04 | 0.23 | 0.07 | 5.87× 10−04 | 1.78× 10−01 | 20 | |
| 11 | ILMN_1704972 | TRIM5 | rs35253098 | 5.68× 10−85 | 6.14× 10−04 | -0.21 | 0.06 | 7.41× 10−04 | 8.73× 10−01 | 20 | |
| COVID-19入院なし§ | 11 | ILMN_2060652 | BET1L | rs4980320 | 2.50× 10-16 | 9.91× 10−06 | 1.55 | 0.40 | 1.00× 10−04 | 6.34× 10−01 | 20 |
| 18 | ILMN_1671693 | C18orf10 | rs3909692 | 3.43× 10−55 | 2.80× 10−04 | -0.66 | 0.19 | 4.02× 10−04 | 4.58× 10−01 | 20 | |
| 1 | ILMN_1741801 | CDC7 | rs13447526 | 1.13× 10−83 | 6.07× 10−04 | 0.50 | 0.15 | 7.36× 10−04 | 3.71× 10−01 | 20 | |
| 7 | ILMN_1714158 | PON2 | rs2374993 | 1.45× 10−44 | 5.20× 10−04 | -0.71 | 0.21 | 7.56× 10−04 | 2.09× 10−01 | 20 | |
| 14 | ILMN_1675391 | SLC24A4 | rs61977314 | 1.84× 10−69 | 6.03× 10−04 | 0.54 | 0.16 | 7.61× 10−04 | 1.73× 10-01 | 20 |
⁎GWASの要約データはCOVID-19宿主遺伝学イニシアティブ(6)から提供されたものであり、対照は特に断りのない限り集団である。血液のeQTLデータ(14)は、https://cnsgenomics.com/data/SMR/#eQTLsummarydata でできる。
#サマリーデータはSevere COVID-19 GWASグループ(5)から提供された。
¶対照は非予測で非自己申告のCOVID-19である。
§対照は入院なしのCOVID-19であり、検査室で確認されたCOVID-19または自己報告されたCOVID-19を含む。
PeQTLはeQTL解析における上位関連cis-eQTLのP値、PGWASはGWAS解析における上位関連cis-eQTLのP値。ベータはSMR解析における推定効果量、SEは対応する標準誤差、PSMRはSMR解析のP値、PHEIDIはHEIDI検定のP値、NsnpはHEIDI検定に関与するSNPの数である。
CHR、染色体;COVID-19,コロナウイルス病2019;HEIDI、依存性機器の異質性;SNP、一塩基多型;SMR、要約データに基づくメンデル無作為化;QTL、定量的形質座位。
太字は、偽発見率を用いた多重検定補正後の統計的有意性を意味する。
図1 血中入院COVID-19と関連したIFNAR2周辺遺伝子の優先順位付け
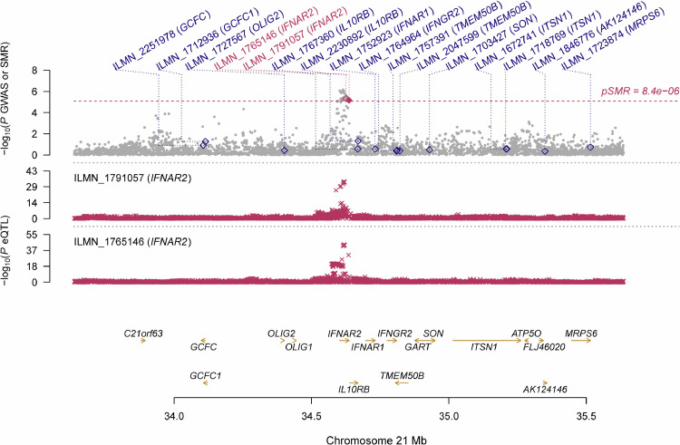
上図、灰色の点は入院COVID-19のGWASからのSNPの-log10(P値)を表し、菱形はSMRテストからのプローブの-log10(P値)を表し、実線の菱形はプローブがHEIDIテストに合格したことを示し、中空の菱形はプローブがHEIDIテストに合格しなかったことを示している。中段のプロット、IFNAR2をタグ付けした2つのプローブ、ILMN_1765146およびILMN_1791057プローブの血液中のeQTLの結果
下段のプロットは、プローブによってタグ付けされた遺伝子の位置。丸で強調表示されているのは、SMR閾値を通過したプローブを示している。COVID-19,コロナウイルス病2019;IFNAR2,インターフェロンアルファおよびベータ受容体サブユニット2;GWAS、ゲノムワイド関連研究;SMR、要約データに基づくメンデル無作為化;HEIDI、依存性機器の不均一性;eQTL、発現量的形質座位
生物学的プロセスと分子機能のGOエンリッチメント解析により、上位5つのプローブでタグ付けされた遺伝子は、I型インターフェロン媒介シグナル伝達経路の制御(GO: 0060338)小胞体輸送の制御(GO: 0060627)小胞体ストレスへの応答(GO: 0034976)酸化ストレスへの応答(GO: 0006979)を含む4つのGO用語に関与していることが示された。IFNAR2,TRIM5,NLRC5,MCTP1,PTPN1,FCER1G、ATF4およびPON2を含む、上位5つのプローブによってタグ付けされた複数の遺伝子が、炎症または抗ウイルス免疫に関与していた(表S2)。
肺におけるSMR解析
多重検定で補正しても、有意な多値関連は認められなかった(図S6-11)。各表現型の上位 5 つのプローブの情報を表 3 に示した。重度のCOVID-19 NEJMの解析では、AP006621をタグ付けした3つのプローブがトッププローブの中に含まれていることがわかった(表3)。さらに、C7orf25,ITGAD、FCER1GおよびZNF589を含む複数の遺伝子が、血液および肺の両方でトップ5のプローブによってタグ付けされていた。
表3 肺のSMR解析で同定された上位5つのプローブ
| 表現型* | CHR | プローブID | 遺伝子 | トップeQTL | P eQTL | P GWAS | ベータ | SE | P SMR | P HEIDI | N SNP |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 重度のCOVID-19 | 7 | ENSG00000136197.8 | C7orf25 | rs583301 | 1.53× 10−33 | 1.59× 10−04 | -0.82 | 0.23 | 3.13× 10−04 | 1.62× 10-01 | 20 |
| 11 | ENSG00000110756.13 | HPS5 | rs4150641 | 3.15× 10-16 | 9.23× 10−05 | -1.02 | 0.29 | 4.21× 10−04 | 3.88× 10−01 | 20 | |
| 15 | ENSG00000244879.3 | GABPB1-AS1 | rs504781 | 1.55× 10−91 | 4.23× 10−04 | -0.27 | 0.08 | 5.14× 10−04 | 5.49× 10-02 | 20 | |
| 15 | ENSG00000259715.1 | CTD-3110H11.1 | rs504781 | 2.39× 10−72 | 4.23× 10−04 | -0.29 | 0.08 | 5.41× 10−04 | 1.29× 10−01 | 20 | |
| 15 | ENSG00000259845.1 | HERC2P10 | rs75874589 | 8.76× 10−39 | 3.43× 10−04 | 0.35 | 0.10 | 5.56× 10−04 | 8.18× 10−01 | 20 | |
| 重度のCOVID-19NEJM # | 11 | ENSG00000177236.3 | AP006621.1 | rs4963156 | 2.04× 10−84 | 1.88× 10−05 | 0.32 | 0.08 | 2.90× 10−05 | 1.33× 10−01 | 20 |
| 11 | ENSG00000255284.1 | AP006621.5 | rs4963156 | 1.31× 10−71 | 1.88× 10−05 | 0.42 | 0.10 | 3.14× 10−05 | 1.93× 10−01 | 20 | |
| 11 | ENSG00000255142.1 | AP006621.6 | rs4963156 | 1.51× 10−33 | 1.88× 10−05 | 0.47 | 0.12 | 5.47× 10−05 | 2.92× 10−01 | 20 | |
| 22 | ENSG00000100299.13 | ARSA | rs6151429 | 6.98× 10−42 | 5.22× 10−04 | 0.32 | 0.10 | 7.74× 10−04 | 7.45× 10−01 | 20 | |
| 20 | ENSG00000231742.1 | RP11-112L6.4 | rs6122883 | 9.14× 10−29 | 4.94× 10−04 | 0.36 | 0.11 | 8.83× 10−04 | 2.47× 10−01 | 20 | |
| COVID-19(新型コロナウイルス感染症)(#文字数制限がない場合、初出時にかっこ書きを追加 | 21 | ENSG00000241837.2 | ATP5O | rs12627646 | 8.98× 10−36 | 3.78× 10−05 | 0.26 | 0.07 | 9.11× 10−05 | 2.68× 10−01 | 20 |
| 10 | ENSG00000214435.3 | AS3MT | rs4919690 | 7.09× 10−41 | 4.30× 10−04 | -0.11 | 0.03 | 6.61× 10−04 | 1.35× 10−01 | 20 | |
| 10 | ENSG00000166275.11 | C10orf32 | rs9527 | 1.76× 10−37 | 6.56× 10−04 | -0.13 | 0.04 | 9.93× 10−04 | 4.25× 10−01 | 20 | |
| 16 | ENSG00000156886.11 | ITGAD | rs2454911 | 1.41× 10−09 | 8.85× 10−05 | 0.19 | 0.06 | 1.00× 10−03 | 6.30× 10−01 | 9 | |
| 16 | ENSG00000223496.1 | EXOSC6 | rs4985407 | 8.47× 10−57 | 8.08× 10−04 | -0.13 | 0.04 | 1.05× 10−03 | 3.14× 10−01 | 20 | |
| COVID-19ネガティブ¶ | 3 | ENSG00000164048.9 | ZNF589 | rs6787500 | 2.81× 10−36 | 2.55× 10−04 | -0.24 | 0.07 | 4.45× 10−04 | 3.37× 10-02 | 20 |
| 1 | ENSG00000158869.6 | FCER1G | rs2070901 | 1.54× 10−09 | 2.16× 10−05 | -1.03 | 0.30 | 5.12× 10−04 | 1.11× 10−01 | 9 | |
| 3 | ENSG00000229759.1 | MRPS18AP1 | rs11130163 | 1.66× 10−47 | 5.76× 10−04 | -0.13 | 0.04 | 8.11× 10−04 | 1.80× 10−01 | 20 | |
| 11 | ENSG00000242689.1 | CNTF | rs73470773 | 1.67× 10-14 | 4.26× 10−04 | 0.30 | 0.09 | 1.36× 10−03 | 1.70× 10-02 | 20 | |
| 17 | ENSG00000261996.1 | CTC-281F24.1 | rs745057 | 1.79× 10-14 | 6.84× 10−04 | 0.27 | 0.09 | 1.91× 10−03 | 4.72× 10−01 | 20 | |
| 入院COVID-19 | 6 | ENSG00000175967.3 | RP11-544L8__B.4 | rs11965077 | 1.78× 10−21 | 4.95× 10−05 | 0.40 | 0.11 | 1.89× 10−04 | 9.36× 10−01 | 5 |
| 16 | ENSG00000103226.13 | NOMO3 | rs55709104 | 7.52× 10−34 | 2.07× 10−04 | -0.21 | 0.06 | 3.88× 10−04 | 7.94× 10−01 | 20 | |
| 11 | ENSG00000117983.13 | MUC5B | rs12802931 | 7.44× 10−10 | 4.19× 10−05 | -0.44 | 0.13 | 6.48× 10−04 | 1.11× 10−03 | 9 | |
| 11 | ENSG00000225101.3 | OR52K3P | rs890417 | 1.50× 10−23 | 4.38× 10−04 | 0.23 | 0.07 | 9.10× 10−04 | 3.83× 10−01 | 20 | |
| 4 | ENSG00000181381.9 | DDX60L | rs6845647 | 8.95× 10-16 | 3.31× 10−04 | 0.38 | 0.11 | 1.05× 10−03 | 4.02× 10−01 | 20 | |
| COVID-19入院なし§ | 11 | ENSG00000185627.13 | PSMD13 | rs1533825 | 9.92× 10−24 | 1.85× 10−05 | -1.43 | 0.36 | 8.18× 10−05 | 4.04× 10−01 | 20 |
| 6 | ENSG00000204536.9 | CCHCR1 | rs1265087 | 5.16× 10−35 | 9.10× 10−05 | 0.72 | 0.19 | 1.91× 10−04 | 1.47× 10−01 | 20 | |
| 11 | ENSG00000142082.10 | SIRT3 | rs6421986 | 6.61× 10−13 | 2.09× 10−05 | 1.99 | 0.54 | 2.51× 10−04 | 2.59× 10−01 | 20 | |
| 18 | ENSG00000134779.10 | TPGS2 | rs80033842 | 1.24× 10−29 | 4.25× 10−04 | -0.69 | 0.20 | 7.67× 10−04 | 2.13× 10−01 | 20 | |
| 22 | ENSG00000100106.15 | TRIOBP | rs5750494 | 7.46× 10−11 | 1.45× 10−04 | 1.24 | 0.38 | 1.03× 10−03 | 4.97× 10−01 | 20 |
⁎ GWASの要約データはCOVID-19宿主遺伝学イニシアティブ(6)から提供されたものであり、対照は特に断りのない限り集団である。肺(15)のeQTLデータは cnsgenomics.com/data/SMR/#eQTLsummarydata でできる。
#サマリーデータはSevere COVID-19 GWASグループ(5)から提供された。
¶対照は非予測、非自己申告のCOVID-19である。
§対照は入院なしのCOVID-19であり、検査室で確認されたCOVID-19または自己報告されたCOVID-19を含む。
PeQTLはeQTL解析における上位関連cis-eQTLのP値、PGWASはGWAS解析における上位関連cis-eQTLのP値。ベータはSMR解析における推定効果量、SEは対応する標準誤差、PSMRはSMR解析のP値、PHEIDIはHEIDI検定のP値、NsnpはHEIDI検定に関与するSNPの数である。
CHR、染色体;COVID-19,コロナウイルス病2019;HEIDI、依存性尺度における異質性;SNP、一塩基多型;SMR、要約データに基づくメンデル無作為化;QTL、定量的形質遺伝子座
上位 5 つのプローブでタグ付けされた遺伝子は、刺激性 C 型レクチン受容体シグナル伝達経路(GO: 0002223)と免疫応答に関与する白血球活性化(GO: 0002366)を含む 2 つの GO 用語に関与していた。ARSA、FCER1G、XOSC6,PSD13を含む上位5つのプローブでタグ付けされた複数の遺伝子は、炎症または抗ウイルス免疫に関与していた(表S3)。
考察
本研究では、GWASとeQTLデータをMR解析に統合し、COVID-19の感受性・予後と多元的な関連を示す遺伝子の探索を行った。その結果、IFNAR2をタグ付けした2つのプローブが、入院したCOVID-19と血液中のCOVID-19との間に多相性を示すことを確認した。上位5つのプローブでタグ付けされた複数の遺伝子は、血液と肺の両方で炎症と抗ウイルス免疫に関与していた。上位5つのプローブでタグ付けされた複数の遺伝子は血液凝固に関与していた。我々の知見は、COVID-19におけるサイトカインストームと静脈血栓塞栓症のメカニズムをよりよく理解するための重要な手がかりを提供し、COVID-19の効果的な治療のための潜在的な治療標的を明らかにした。
インターフェロン(IFN)は、ウイルスの侵入に応答して宿主細胞によって作られ、放出されるシグナル伝達タンパク質のグループを指す(17)。IFNには3つのタイプがある:I型IFN(IFN-α/β)III型IFN(IFN-γ)およびIII型IFN(IFN-λ)(18,19,20)。21q22.11に位置するIFNAR2(インターフェロンαおよびβ受容体サブユニット2)は、2つのタイプIFNのうちの1つをコードしている(21)。コロナウイルスに対する宿主の応答のカスケードにおいて、IFNは、抗ウイルス状態の確立および抗ウイルス応答の増強において重要な役割を果たしている(22)。IFNはSARS-Cov-2の複製に有益な効果と有害な効果の両方をもたらすことが明らかになった(23)。77人の成人を対象とした最近のレトロスペクティブ研究では、アルビドールの有無にかかわらずIFN-α2b治療を行うと、COVID-19患者の上気道で検出可能なウイルスの持続時間が有意に減少することが明らかになった(24)。しかしながら、COVID-19治療ガイドラインパネルは、インターフェロンの有益または有害な効果を裏付けるデータが不十分であるため、臨床試験を除き、重症および重症のCOVID-19患者の治療にインターフェロンを使用しないことを推奨している(25)。IFNAR2は複数の表現型のトップ5プローブでタグ付けされていたため、COVID-19の重症度の決定に関与している可能性が高く、COVID-19の治療標的となり得ると考えられる。SARS-Cov-2の間のIFNの二重の役割の根底にあるメカニズムと、IFNAR2がこのプロセスに関与しているかどうか/どのように関与しているかを解明するためには、より多くの研究が必要である。
我々は、トップ5のプローブによってタグ付けされた複数の遺伝子が炎症に関与していることを発見した。ATF4(活性化転写因子4)は小胞体ストレスセンサーであり、ヘムオキシゲナーゼ1の誘導を介して肺を防御する(26)。活動性クローン病や活動性潰瘍性大腸炎患者の炎症性腸管粘膜ではATF4が減少しており、ATF4欠損症はマウスの腸管炎症を促進していた(27)。ATF4は、高齢者の肺胞Ⅱ型細胞において、若年者と比較してダウンレギュレーションされていた(28)。ATF4は20以上の候補ダウンストリーム因子を有しており、その大部分は小胞体ストレスに応答するためのATF4依存性の能力が低下していることを示した高齢者において有意にダウンレギュレーションされていた。ATF4関連の抗酸化物質のラング特異的送達は、高齢者におけるCOVID-19の転帰をさらに改善するために、有望な抗ウイルス薬との相乗効果を発揮する可能性がある(28)。TRIM5,NLRC5,MCTP1,PTPN1,ARSAおよびFCER1Gなどのタグ付けされた遺伝子のいくつかもまた、抗ウイルス免疫に関与している(29,30,31,32,33,34,35)。これらの遺伝子がCOVID-19と関連していることは、これまでの研究では報告されていない。
COVID-19患者の約40%が静脈血栓塞栓症リスクが高いとされ、そのうち11%が予防薬なしで静脈血栓塞栓症を発症したと推定されている(36)。COVID-19患者の凝固異常は死亡リスクの増加と関連していた(37)。最近の研究では、SARS-CoV-2感染症では、血小板と好中球の調節異常が協力して全身性の血栓塞栓状態を誘導していることが示された(38)。しかし、一般的なメカニズムと、特に遺伝的な寄与については、さらに研究が進められている。我々の研究では、トップ5のプローブでタグ付けされた2つの遺伝子が血液凝固に関与していることを発見した。入院を伴わないCOVID-19の血中分析(表2)および重度のCOVID-19の肺分析(表3)において、それぞれPON2およびHPS5が検出された。PON2(パラオキソナーゼ2)は、パラオキソナーゼ遺伝子ファミリーのメンバーをコードし、細胞の抗酸化物質として作用し、酸化ストレスから細胞を保護する(39)。PON2欠損症では、レドックス制御が緩和されると、血管炎症や血液凝固異常を引き起こすことが示されている(40)。HPS5は、メラノソームや血小板緻密顆粒に関連したオルガネラ生合成に関与するタンパク質をコードしている(41)。HPS5の変異は、眼球皮アルビニズムおよび長引く出血を特徴とする一連の障害の亜型であるハーマンスキー・プドラック症候群5型(Hermansky-Pudlak syndrome type 5:HPS5)と関連している(42)。多重検定補正後のSMR解析では、多面的な関連性は統計的に有意ではなかったが(PON2ではP=7.56×10-4,HPS5では4.21×10-4)COVID-19患者の静脈血栓塞栓症の病態にこれらの遺伝子が関与している可能性が示唆された。SARS-CoV-2感染に対するこれら2つの遺伝子の機能を調べるためには、さらなる研究が必要である。
我々の研究にはいくつかの限界がある。GWAS解析では、転帰に影響を及ぼす可能性のある交絡因子をコントロールしていない。また、GWAS研究における対象者の選択が集団全体の曝露-転帰分布を代表するものであったかどうかも不明であり、推定精度に影響を及ぼす可能性のある選択バイアスの可能性を排除することはできなかった。GWAS試験では、COVID-19パンデミックの期間が限られていたため、COVID-19の短期的な効果のみを検討しており、COVID-19の長期的なアウトカム/リンゲル効果を評価することはできなかった。同様に、対応するGWAS要約データがないため、小児/10代、成人および高齢者患者の疾患行動の違い、および無症候性COVID-19のような他の興味深い表現型の遺伝的寄与を解析することができなかった。我々は血液と肺のeQTLデータを用いた解析のみを行ったが、COVID-19感染に対する宿主応答に関与する組織・細胞型特異的遺伝子を探索するためには、より多くの研究が必要である。個々のeQTLデータが不足しているため、COVID-19感染患者における遺伝子発現の変化をコントロールと比較して定量化することはできなかった。
結論
また、IFNAR2をはじめとするCOVID-19の感受性・予後に関与する可能性のある遺伝子を同定した。これらの知見は、COVID-19におけるサイトカインストームのメカニズムをよりよく理解するための重要な手がかりを提供し、COVID-19の効果的な治療のための潜在的な治療標的を明らかにした。
