Contents
Identification of 3-chymotrypsin like protease (3CLPro) inhibitors as potential anti-SARS-CoV-2 agents
2021年1月20日
pubmed.ncbi.nlm.nih.gov/33473151/
ヴィッキー・モディ、ジョアンナ・ホー、サバンナ・ウィルズ、アーメド・マウリ、ラターシャ・ローソン、マクシミリアン・C.
要旨
重症急性呼吸器症候群コロナウイルス2型(SARS-CoV-2)感染症の発生は,公衆衛生にとって大きな脅威である.SARS-CoV-2に特異的な薬剤が不足しているため、罹患率が増加している。ここでは、SARS-CoV-2の複製に重要な役割を果たす主要プロテアーゼである3-キモトリプシン様プロテアーゼ(3CLpro)を標的とした薬剤を同定した。
計算分子モデリングを用いてFDA(アメリカ食品医薬局)承認薬3987種をスクリーニングし、47種の薬剤を選択して、SARS-CoV-2特異的な3CLpro酵素に対する阻害効果を試験管内試験で検討した。その結果、ボセプレビル、オンビタスビル、パリタプレビル、ティプラナビル、イベルメクチン、ミカファンギンが3CLpro酵素活性を阻害する効果を示した。また,100nsの分子動力学シミュレーションにより,イベルメクチンの阻害作用には3CLpro酵素のホモ二量体が必要であることが示唆された。
以上のことから,これらの分子は,SARS-CoV-2 ウイルスの複製を阻害するために,単独で,あるいは他の SARS-CoV-2 ウイルスに特異的な薬剤と組み合わせて,特異性の高い治療可能な薬剤を開発するために有用であると考えられる。
序論
重症急性呼吸器症候群コロナウイルス2型(SARS-CoV-2)による21世紀の大パンデミックは、死亡に至る感染率の高さから世界的な公衆衛生上の脅威となっている。2020年12月24日現在、米国だけでCOVID-19陽性患者数は13,881,620人、死亡者数は272,820人となっており、世界全体では64,326,880人の確定症例と1,488,992人の死亡者が確認されている(https://coronavirus.jhu.edu/)。COVID-19に特異的な薬剤やワクチンがないため、死亡者数は驚くべき速さで増加している。COVID-19特異的薬剤の開発、バリデーション、承認には何年もかかる1。そのため、リパーポージングとも呼ばれる薬剤のリポジショニングの考え方は、急速に拡散する生命を脅かす感染症の突然の発生を抑制するための重要な戦略である。FDA(アメリカ食品医薬局)承認の抗ウイルス薬は、ヒトでの使用が安全であることが知られている2が、SARS-CoV-2に対する有効性は実験的に検証する必要がある。FDA(アメリカ食品医薬局)承認の抗ウイルス薬としては、ファビピラビル、ダノプレビル、ダルナビル、ロピナビル、オセルタミビル、リトナビル、レムデシビル、ユミフェノビルなどがあり、抗COVID-19活性を調べるために臨床試験が行われている3。しかし、これらの薬剤のCOVID-19の症状の予防や重症化の軽減に対する有効性はまだ完全には確立されていない。したがって、COVID-19の治療効果を高めるために、異なるSARS-CoV-2タンパク質を標的とする追加の薬剤候補を同定することが急務となっている。
最近、Wuら4は、SARS-CoV-2のゲノムを他のコロナウイルス(CoV)と比較し、新規SARS-CoV-2がβ-CoVに属することを確認した。CoVsは、ポジティブセンス一本鎖RNA(+ssRNA)を遺伝物質とするRNAウイルスであり5,最近の研究により、SARS-CoV-2は他のSARS-CoVs4と89%程度の配列類似性を有することが明らかになっている。さらに、SARS-CoV-2は、他のSARS-CoVと同様の遺伝的組織を有しており、5′の非翻訳領域に続いて16の非構造タンパク質(オープンリーディングフレーム;ORF1aとORF1b複合体)は、レプリカーゼ複合体とも呼ばれ、スパイク(S)エンベロープ(E)膜(M)およびヌクレオカスピド(N)タンパク質などの構造タンパク質は、3′末端に向かって存在する他の付属タンパク質と一緒に6. ウイルスのライフサイクルは、宿主細胞、ACE2上のその受容体へのウイルスのSタンパク質の結合から始まる。結合は、その後、宿主細胞膜と細胞質7にウイルスゲノムのリリースとウイルスエンベロープの融合が続いている。ウイルスゲノム(+ssRNA)は、宿主のリボソームをハイジャックし、800KDaの大型ポリペプチド(PP)鎖に翻訳される。新たに生成されたPP鎖は、ウイルスゲノムにコードされているパパイン様プロテアーゼ(PLpro)と3-キヨモトリプシン様プロテアーゼ(3CLpro)のような2つのプロテアーゼによって自己保護的に切断され、ウイルスの複製に必要ないくつかの非構造タンパク質(NSP)を生成する。3CLproはメインプロテアーゼ(Mpro)とも呼ばれ、ウイルス複製に大きな役割を果たしている。PLproと3CLproはPP鎖を16個のNSPに切断し、生成された16個のNSPのうち、11個のNSPが3CLproによって生成されることから、このプロテアーゼは抗SARS-CoV薬開発の主要なターゲットの一つとなっている8,9。一方、構造タンパク質やその他のアクセスタンパク質は、サブゲノム(Sg)翻訳と呼ばれる独自のメカニズムで生成される。SgはアンチセンスウイルスRNAの5′末端10,11,12,13からの不連続転写によって産生される。ゲノムの複製と翻訳に成功した後、NSP、構造タンパク質とアクセサリータンパク質は、ポジティブセンスウイルスRNAゲノムとともに集合し、新しいウイルスを形成する。CoVsゲノムとPLproおよび3CLproによるタンパク質分解開裂を図1に示する。
図1 SARS-CoV-2のゲノム配列とプロテアーゼ切断部位を表した図。
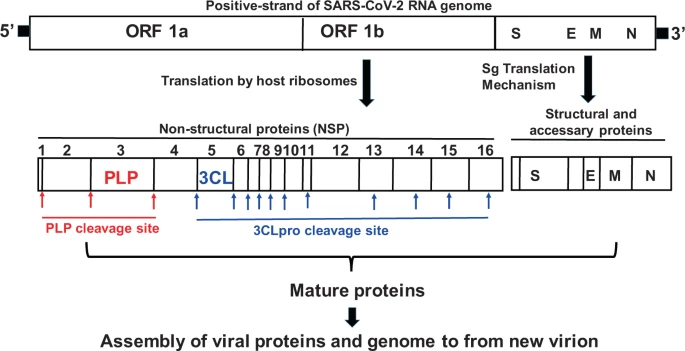
SARS-CoV-2の遺伝物質(RNA)は宿主細胞に入り、リボソームを借りて、PLproと3CLproの酵素で自己増殖的に切断されて16種類の非構造タンパク質に翻訳される。構造タンパク質(S, E, M, N)はsg翻訳機構により翻訳される。すべての成熟タンパク質は、ポジティブセンス一本鎖RNAゲノムと一緒に集合し、新しいウイルスを形成する。矢印はプロテアーゼ切断部位を示す。ORFオープンリーディングフレーム、PLPパパイン様プロテアーゼ、3CLP3-キモトリプシン様プロテアーゼ、Sスパイク蛋白質、Eエンベロープ蛋白質、M膜蛋白質、Nヌクレオカスピド蛋白質。
SARS-CoV-2のゲノムおよびタンパク質配列はNIH(アメリカ国立衛生研究所)遺伝子データバンクから公開されている4,6,14。ここでは、SARS-CoV-2の3CLproを標的として、このプロテアーゼがウイルスの複製に不可欠であることから、優れた創薬ターゲットとなることを明らかにした9。SARS-CoV-2の3CLproタンパク質と阻害剤N3との複合体の構造がPDBデータベースに掲載されている(ID: 6LU7)。3CLproの阻害剤としてFDA(アメリカ食品医薬局)に承認されている薬剤を特定するために、in silico薬物スクリーニング試験を実施した。全3987種類のFDA(アメリカ食品医薬局)承認薬(SuperDrugs2データベース)をウイルスプロテアーゼ阻害剤(PI)ウイルス非プロテアーゼ阻害剤(VNI)オフターゲット薬(OTD)に分類し、Molecular Operating Environment(MOE)ソフトウェアを用いて抗3CLpro活性をスクリーニングした。計算手法を用いたタンパク質構造に基づくドラッグデザインは、COVID-1915,16,17のような新興感染症の治療薬候補を迅速に同定するために、現在承認されている薬剤のスクリーニングに代わるものである。しかし、計算モデルによる偽陽性の可能性は、ドッキング研究の最も一般的な限界の一つである18。そこで、我々は、市販の3CLproプロテアーゼ阻害剤スクリーニングアッセイキットを用いて、選択された薬剤のSARS-CoV-2 3CLpro酵素アッセイを確立し、薬剤の試験管内試験阻害活性を評価し、計算上の結合スコアと試験管内試験阻害活性との間に相関関係が存在するかどうかを調査した。本報告では、FDA(アメリカ食品医薬局)承認薬3987品目の中から、結合スコア、副作用、半減期、活性型、免疫抑制性、自家蛍光性、試験管内試験 3CLpro酵素阻害剤スクリーニングアッセイ用の入手可能性に基づいて47品目を選択し、その中から3CLpro酵素阻害剤スクリーニングアッセイ用のキットを作成した。我々は、ボセプレビル、オンビタスビル、パリタプレビル、ティプラナビル、ミカファンギンが部分的に阻害効果を示したのに対し、イベルメクチンはSARS-CoV-2の3CLpro活性の85%以上を阻害したことを確認した。イベルメクチンの抗ウイルス作用はα/β1インポーチン19,20,21,22,23の阻害を介して確立されているが,ここではSARS-CoV-2の3CLpro酵素に対するイベルメクチンの阻害作用を報告し,SARS-CoV-2に対するイベルメクチンのさらなる抗ウイルス機序が示唆された。
研究結果
SARS-CoV-2 3CLpro酵素に結合する可能性のあるFDA(アメリカ食品医薬局)承認薬のin silicoスクリーニング
FDA(アメリカ食品医薬局)承認の全3987種類の医薬品(SuperDrugs2から)をPI、VNI、OTDとしてソートし、Molecular Operating Environment(MOE)ソフトウェアを使用して3CLproタンパク質のモノメリックフォームとドッキングした。薬物は、最高の結合エネルギー(Sスコア)に応じてランク付けされた。S スコアは、配置のための London dG スコアとポーズの洗練のための GBVI/WSA dG スコアを使用して計算された。≦-6.5 kcal/mol(Sスコア)の結合エネルギーを、最高の結合親和性を持つ薬剤をランク付けするためのカットオフとして選択した16。このリストは、「方法」の項に記載されている基準に基づいてさらに絞り込まれた。その結果、56の薬剤が3CLpro酵素に対する結合親和性が<-6.50 kcal/molであることがわかった(表1)。計算研究により、同定された薬剤のリストは、ウイルスの3CLプロテアーゼを標的とすることにより、SARS-CoV-2ウイルスの複製を阻害する可能性があることが示唆された。しかし、予測された結合スコアと偽陽性の可能性は、ドッキング研究24,25の最も一般的な制限の一つである。そこで、偽陽性予測の曖昧さを排除するために、我々は、潜在的な治療法を同定し、結合スコアと試験管内試験活性との相関を調査するために試験管内試験酵素アッセイを実施した。今回の研究では、選択された薬剤の試験管内試験結果と計算上の阻害定数との間に相関関係は見られなかった。初期の蛋白質-薬物相互作用を予測するために計算科学的な研究が広く行われているとはいえ、薬物の阻害活性を確認するためには試験管内試験でのスクリーニングが必要であると考えられる。
表1 計算科学的研究により算出された3CLプロテアーゼのSスコアが最も高かった薬剤の一覧(Sスコア)
| SL。番号。 | 薬 | 3CLproへの結合親和性(Kcal / mol) |
|---|---|---|
| 1 | アバカビル | −6.6178 |
| 2 | アンプレナビル | −8.29197 |
| 3 | アスナプレビル | −9.64249 |
| 4 | アタザナビル | −8.51517 |
| 5 | アトルバスタチン | −7.98771 |
| 6 | ベクラブビル | −8.45563 |
| 7 | ボセプレビル | −8.4209 |
| 8 | カモスタット | −7.66508 |
| 9 | カンジシジン | −9.05496 |
| 10 | クロロキン | −6.7514 |
| 11 | ダクラタスビル | −8.47852 |
| 12 | ダノプレビル | −9.26908 |
| 13 | ダルナビル | −8.59002 |
| 14 | デラビルジン(メシル酸塩) | −7.3092 |
| 15 | エルバスビル | −8.77218 |
| 16 | Elvitegravir | −7.48581 |
| 17 | エトラビリン | −6.91494 |
| 18 | ファビピラビル | −4.23592 |
| 19 | フマギリン | −7.25118 |
| 20 | Gabexate | −7.24702 |
| 21 | グレカプレビル | −8.70897 |
| 22 | グラゾプレビル | −8.85946 |
| 23 | ヒドロキシクロロキン | −7.03572 |
| 24 | インジナビル | −8.39968 |
| 25 | イトラコナゾール | −7.99098 |
| 26 | イベルメクチン | −7.74053 |
| 27 | レジパスビル | −9.52825 |
| 28 | ロピナビル | −9.10074 |
| 29 | マラビロック | −8.19033 |
| 30 | メチルプレドニゾロン | −6.42667 |
| 31 | ミカファンギン | −9.60339 |
| 32 | ネルフィナビル | −8.55575 |
| 33 | オムビタスビル | −8.96571 |
| 34 | Oseltamvir Phosphate | −7.04549 |
| 35 | パリタプレビル | −7.43470 |
| 36 | ペラミビル | −6.7811 |
| 37 | ピブレンタスビル | −9.87502 |
| 38 | ピモディビル | −6.79098 |
| 39 | プレコナリル | −7.16664 |
| 40 | ポサコナゾール | −7.95234 |
| 41 | キニーネ | −6.67637 |
| 42 | ラルテグラビル | −7.76854 |
| 43 | レムデシビル | −5.9488 |
| 44 | リバビリン | −6.3251 |
| 45 | リボスタマイシン | −7.28802 |
| 46 | リルピビリン | −7.09415 |
| 47 | リトナビル | −9.94965 |
| 48 | サキナビル | −9.25575 |
| 49 | シメプレビル | −9.50916 |
| 50 | ソフォスブビル | −8.4513 |
| 51 | テラプレビル | −8.96481 |
| 52 | テムサビル | −8.33862 |
| 53 | テノホビル二リン酸 | −6.557 |
| 54 | チプラナビル | −7.47819 |
| 55 | ウミフェノビル | −7.23012 |
| 56 | ベルパタスビル | −9.33025 |
選択された薬剤候補によるSARS-CoV-2 3CLpro酵素活性阻害作用
選択された化合物のSARS-CoV-2 3CLpro阻害活性を計算上の研究からさらに検証するために、市販のアッセイキットを用いて試験管内試験酵素阻害アッセイを行った。選択された56化合物のバックグラウンド自家蛍光を360/460 nmの励起/発光波長で測定した。56剤中9剤が極めて高い自家蛍光を示した(補足図2)ため、試験管内試験プロテアーゼ阻害酵素アッセイから除外した。残りの47剤をPI、VNI、OTDに分類した。具体的な作用機序およびそれらの臨床的用途は、補足表1に記載した。3CLpro阻害活性は、これらの薬剤の50μM濃度でスクリーニングした。スクリーンされた17種類のPIのうち、ボセプレビル、パリタプレビル、ティプラナビルは、それぞれ45%、70%、64%の3CLpro酵素活性を有意に阻害した(図2)。また、17種類のVNIsのうち、3CLpro酵素活性を部分的に(65%)阻害できたのはオンビタスビルのみであった(図3)。興味深いことに,図4に示すように,13種類のOTDのうち,イベルメクチンのみが50μMの濃度で3CLpro活性を完全にブロックした(80%以上)。また,ミカファンギンはSARS-CoV-2の3CLproに対して57%の部分的な阻害活性を示した。
図2:プロテアーゼ阻害剤であるボセプレビル、パリタプラビル、ティプラナビルは、SARS-CoV-2の3CLpro酵素に対して部分的な阻害活性を示した。
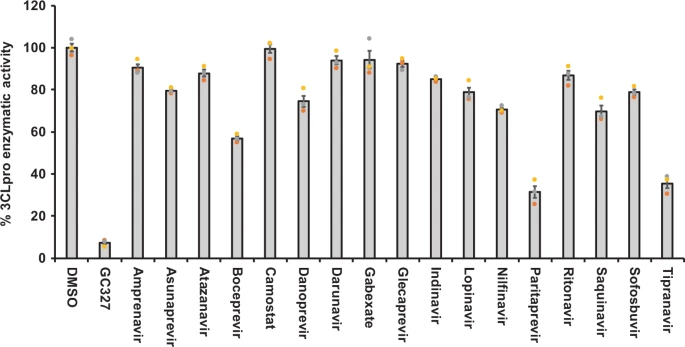
選択されたFDA(アメリカ食品医薬局)承認のウイルスプロテアーゼ阻害剤を、方法の項に記載されているように、SARS-CoV-2 3CLpro酵素に対する阻害活性についてスクリーニングした。蛍光単位は、DMSO処理した対照を100%の活性として考慮して、酵素活性パーセントに換算した。ブランク値は、パーセント活性を計算する前に、すべての測定値から差し引いた。トリプリケート値を持つ3つの個々の実験の代表は、グラフで示された(n = 3)。P値< 0.001は、統計的に有意であると考えられる。Dunnettの多重比較ポストホック検定を用いた一方向ANOVAは、統計的有意性を計算するために使用された。
図3:非プロテアーゼ阻害剤オンビタスビルがSARS-CoV-2 3CLpro活性を部分的に阻害した
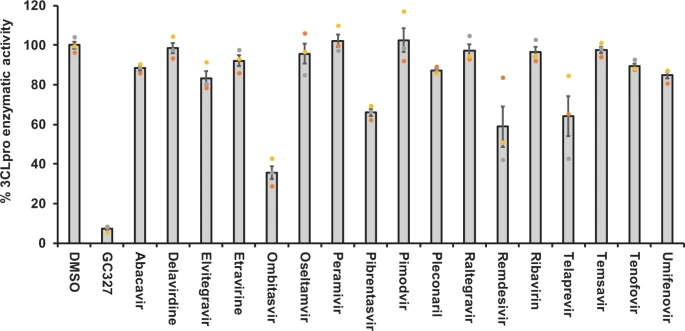
計算研究によって選択された非プロテアーゼ抗ウイルス剤を、方法の項に記載されているように、SARS-CoV-2 3CLpro酵素に対する阻害活性についてスクリーニングした。酵素活性のパーセントは、図1の凡例に記載されているように計算した。ブランク値は、パーセント活性を計算する前に、すべての測定値から差し引いた。トリプリケート値を持つ3つの個々の実験の代表は、グラフで示された(n = 3)。P値< 0.001は、統計的に有意であると考えられる。統計的有意性を計算するために使用されるDunnettの多重比較ポストホック検定との一方向ANOVA。
図4:イベルメクチンはSARS-CoV-2 3CLpro酵素活性の完全な阻害を示したが、ミカファンギンは部分的に阻害した
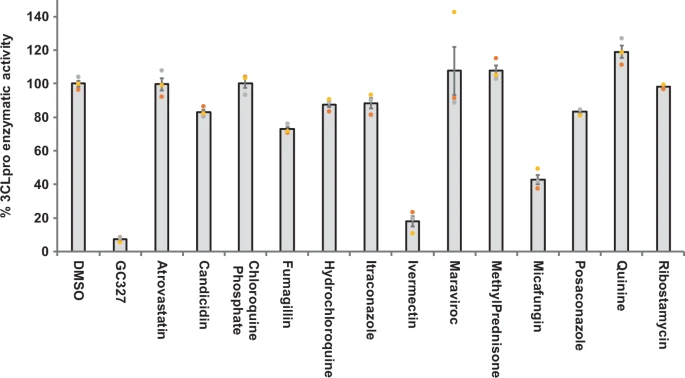
in silico試験により選択された非ウイルス性疾患の治療に使用されているオフターゲット薬剤を、Methodsの項に記載されているように、SARS-CoV-2 3CLpro酵素に対する阻害活性についてスクリーニングした。酵素活性のパーセントは、図1の凡例に記載されているように計算した。ブランク値は、パーセント活性を計算する前に、すべての測定値から差し引いた。トリプリケート値を持つ3つの個々の実験の代表は、グラフで示された(n = 3)。P値< 0.001は、統計的に有意であると考えられる。統計的有意性を計算するために使用されるDunnettの多重比較ポストホック検定との一方向ANOVA。
50%以上の阻害活性を示した化合物は、その後、用量依存性試験を行い、3CLpro酵素活性の50%を阻害するのに必要な濃度(IC50)を算出した。ボセプレビル、イベルメクチン、ミカファンギン、オンビタスビル、パリタプレビル、およびティプラナビルについては、用量依存的な阻害活性試験を行った。図5に示すように、イベルメクチンは50μM濃度で85%以上の酵素活性を阻害したのに対し、ミカファンギンおよびパリタプレビルは100μM濃度で約80%の酵素活性を阻害した。また,ティプラナビルとオンビタスビルは,100 μM 濃度でも酵素活性の 50%程度しか阻害されなかった(図 5)。酵素活性の対数濃度に対するパーセンテージを用いて、「方法」の項で述べた非線形カーブフィットモデルを用いてIC50値を算出した。その結果,イベルメクチン,ティプラナビル,ボセプレビル,ミカファンギン,パリタプレビル,オンビタスビルのIC50値は,それぞれ21.5,27.7,31.4,47.6,73.4,75.5μMであった(表2).以上のことから,上記の分子はSARS-CoV-2の3CLpro酵素に対して阻害活性を示すことが示唆された。
図5:選択されたPI、VNI、およびOTDによるSARS-CoV-2の3CLpro活性の用量依存的阻害
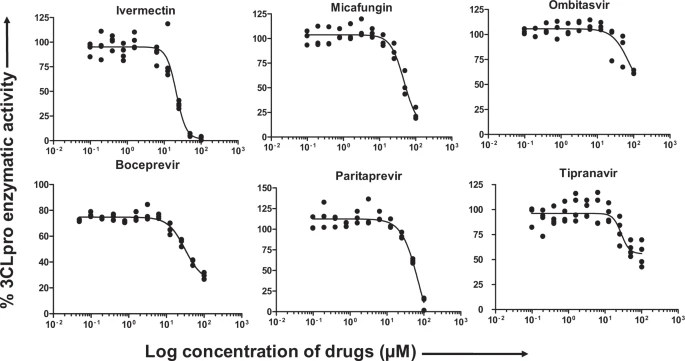
50μMの濃度で50%以上の阻害活性を示した薬剤候補を、用量依存性およびIC50計算試験のために選択した。0〜100μMの範囲の薬剤をアッセイバッファー中で連続希釈したものを使用した。パーセントの活性を、図1の凡例に記載されているように計算した。トリプリケート値を持つ3つの個々の実験の代表をグラフ化して示した(n = 3)。4つの変数の用量対阻害を持つ非線形回帰(カーブフィット)は、GraphPad Prismを使用してIC50値を計算するために使用された。
表2 SARS-CoV-2 3CLプロテアーゼの非ウイルス性プロテアーゼ阻害剤のIC50値
| SL。番号。 | 薬名 | IC 50値 |
|---|---|---|
| 1 | イベルメクチン | 21.53 |
| 2 | チプラナビル | 27.66 |
| 3 | ボセプレビル | 31.36 |
| 4 | ミカファンギン | 47.63 |
| 5 | パリタプレビル | 73.38 |
| 6 | オムビタスビル | 75.49 |
ボセプレビル、パリタプレビル、オンビタスビル、ティプラナビル、イベルメクチン、ミカファンギンの3CLproの酵素活性部位との構造的相互作用
3CLproにおける型破りな触媒的システイン残基の存在は、酵素や他のSer(またはCys)ヒドロラーゼ26のような他のキモトリプシンと比較してユニークなものになる。さらに、3CLproは、標準的なSer(Cys)-His-Asp(Glu)トライアッド27の代わりに、触媒的なCys145-His41ダイアドで構成されている。この触媒的なダイアドは、Leu-Gln↓Ser-Ala-Gly(↓は開裂部位をマーク)26を含む基質の存在下で活性化される。構造的には、3CLproの触媒残基Cys145とHis41は、タンパク質の表面に位置する活性部位の空洞に埋もれている(補足図1)と、両方の残基は3.8Åの距離にある26,28。この距離は、生理的なpHでCys145とHis41の間に分子間水素結合が形成されないようにするのに十分な長さであると考えられる。また、Cys145は生理的pHではプロトン化されており、His41は中性状態で存在する。基質結合は、基質ペプチド結合27の(C=O)上にCys145-硫黄の攻撃を介してトリガされたHis41にCys145から分子内プロトン移動をトリガする。触媒活性の間に、プロトン化されたHis41は、His41の近くにH2O分子の存在によって安定化される。したがって、Cys145とHis41の両方の残基またはこれらの残基のいずれかで水素結合を形成する能力は、良い阻害剤の特性である可能性がある。
計算モデルを用いて、ボセプレビル、パリタプレビル、オンビタスビル、ティプラナビル、イベルメクチン、ミカファンギンの3CLpro SARS-CoV-2酵素との構造的相互作用を調べた。その結果、イベルメクチンとボセプレビルのカルボニル(C=O)基がCys145と水素結合を形成していることがわかった(図6a,e、パネルIとII)。また、パリタプレビルのスルホンアミドに隣接するカルボニル基(C=O)とCys145の間にも同様の水素結合が観察され、ティプラナビルのスルホンアミド基とCys145の間にも同様の水素結合が観察された(図6b, c:パネルIおよびII)。また、パリタパビルの同じカルボニル(C=O)基もHis41と水素結合相互作用を示している(図6b;パネルIおよびII)。しかし、オンビタパビルとミカファンギンはGlu166と水素結合相互作用を示した(図6d、f、パネルI、II)。オンビタサビルおよびミカファンギンとGlu166との水素結合相互作用は、Glu166がSARS-CoV-228における3CLproのホモ二量体の形成に関与していることから、非常に重要である。この二量体の形態は、その酵素活性にとって重要であり、Glu166との任意の相互作用は、3CLpro28の酵素活性を阻害する不活性モノマーの形成をもたらす可能性がある。パネルIIIは、3CLpro酵素部位の親油性ポケットと薬物の相互作用を示している。
図6:潜在的な阻害剤のSARS-CoV-2 3CLpro酵素との構造解析および相互作用を示す
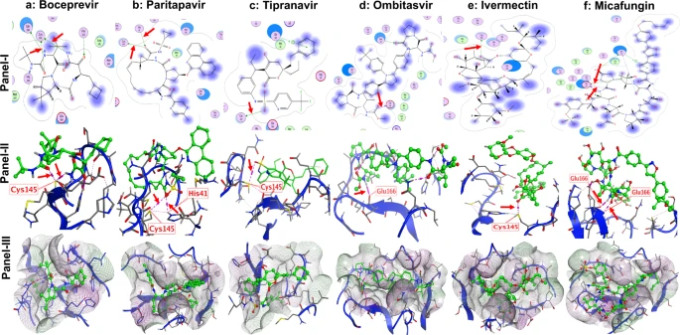
インヒビターは3CLproタンパク質の活性部位とドッキングした。パネル-I:リガンド相互作用マップ。パネルII:阻害剤の活性部位における3CLproの特定のアミノ酸との相互作用。パネル-III:特定のアミノ酸と相互作用する薬剤と活性部位の親油性空洞。比較のために、ボセプレビル(a)パルタプラビル(b)ティプラナビル(c)オンビタスビル(d)イベルメクチン(e)ミカファンギン(f)を列に並べた。薬剤は緑色でボールとスティックモデルで表現している。矢印は薬剤間のC-H、N-H、C-O結合を示し、Cys145,His41,Glu166残基は3CLpro酵素の酵素活性に必須であることから、薬剤間のC-H、N-H、C-O結合を示している。しかし、薬剤が隣接するアミノ酸残基と相互作用していることも観察された。
100 ナノ秒 (ns) 分子動力学シミュレーションによるミカファンギンとイベルメクチンの解析
これらのドッキングポーズの安定性を調べるために、イベルメクチンとミカファンギンの2つの化合物について100 nsの分子動力学(MD)シミュレーションを行った。図7aパネルI-IIIは、3CLproのモノマー形態でのミカファンギンのMDシミュレーションを示している。MDシミュレーションデータは、ミカフンジンが安定であり、100nsのシミュレーション時間を通して活性部位ポケットに結合したままであることを示唆している(図7aパネルI-IIIを比較)。モノメリック3CLpro酵素とミカファンギンの間のタンパク質-リガンド相互作用フィンガープリントの解析(図7a、パネル-IV)は、ミカファンギンがGlu166との優勢な相互作用を有することを示している。これは、ミカファンギンがGlu166との水素結合を介して100nsの軌跡の全長にわたって3CLproのモノメリック形態のポケットに結合したままであった可能性がある。前述のように、Glu166との相互作用は、SARS-CoV-2の3CLproの二量体化を阻害することを示唆している28。補足動画1は、モノマーの3CLproの活性部位とミカファンギンのリアルタイム相互作用を示しており、モノマーの触媒ポケット内でのミカファンギンの安定性を示している。
図7:イベルメクチンとミカファンギンのMD100nsシミュレーション研究
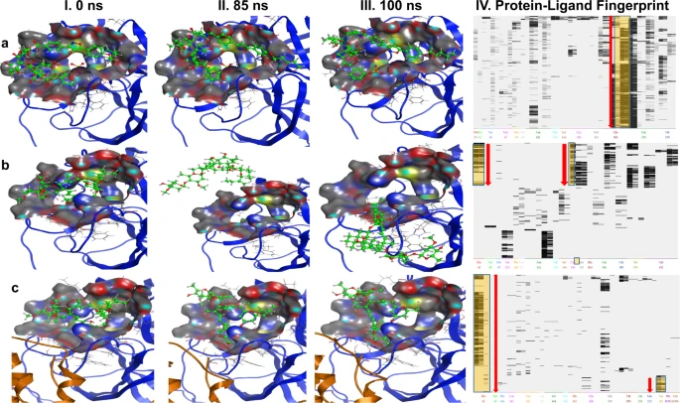
MD シミュレーションは、Methods の項に記載されているように実施した。ミカファンギン(a)、イベルメクチン(b)と3CLpro酵素のモノマーとの相互作用 c イベルメクチンと3CLproホモダイマーの活性部位との相互作用 パネルI-IIIは、ナノ秒(ns)での異なる時間ポイントでのリガンドの相互作用を示す。パネルIVは、3CLpro酵素の特定のアミノ酸とミカファンギンおよびイベルメクチンのリガンド結合フィンガープリントを示す。
イベルメクチンの MD シミュレーションを図 7b(パネル II-III)に示すが、イベルメクチンは 85 ns 後に 3CLpro モノマーの触媒ポケットから拡散する。イベルメクチンは、約14 nsの間、3CLproモノマーのCys145およびHis41の両方と相互作用していることは、タンパク質-リガンドフィンガープリントマップから明らかである(図7b、パネル-IV)。その後、イベルメクチンはHis41との相互作用を失い、関心のあるアミノ酸(Cys145,His41,およびGlu166)との相互作用を示さず、最終的には85 nsでポケットの外に拡散する(図7b、パネル-II)。補足動画2は、3CLproのモノマー形態の触媒ポケット内のイベルメクチンの不安定性を示す。ホモ二量体は3CLpro酵素28の活性形態であるので、我々は、3CLproのホモ二量体形態が触媒ポケット内のイベルメクチンを安定化するために必要であり、それゆえにイベルメクチンの阻害活性に責任があるという仮説を立てた。この仮説を検証するために、3CLproのホモ二量体を用いたイベルメクチンのMDシミュレーションを行った。興味深いことに、イベルメクチンがホモ二量体の触媒ポケットに結合したままであることが観察された(図 7c、パネル I-III 参照)。ホモダイマー3CLpro-イベルメクチンフィンガープリント領域の詳細な解析(図7c、パネル-IV)は、イベルメクチンが2 nsの間、Cys145およびHis41の両方と相互作用し、その後、His41と相互作用することを示している。85 ns後、イベルメクチンは、隣接するモノマーのSer1と接触し、このアミノ酸残基が触媒結合ポケット内のイベルメクチンの安定化を助けることを示唆している(図7c、パネルIV)。補足動画3は、リアルタイムMDシミュレーションの相互作用と3CLproのホモダイマーとの触媒ポケット内でのイベルメクチンの0-100nsからの安定性を示している。
また、補足図3には、3CLproのモノマー型とホモダイマー型のイベルメクチンと、3CLproのモノマー型のミカファンギンのMDシミュレーションの過程での結合親和性(S-score)を示した。ミカフンジンのSスコアは計算期間中安定していたが、3CLproのモノマー型を用いたイベルメクチンは-9.64~-2.2 kcal/molの範囲で変動していた。補足図 3 に示すように,イベルメクチンは活性部位を離れてから ~85 ns で S スコアが増加した(~-2.0 kcal/mol).これは、3CLproのホモ二量体との複合体におけるイベルメクチンのSスコアとは対照的であり、平均-5.64 kcal/molとシミュレーションを通して安定していた。以上のことから、この計算モデルは、これらの阻害剤と3CLproとの相互作用の可能性の枠組みを提供している。しかし、これらの阻害剤とSARS-CoV-2 3CLproの構造的相互作用については、X線結晶構造解析による検証が必要である。
考察
COVID-19は、SARS-CoV-2によって引き起こされる疾患であり、その感染率と死亡率の高さから、世界的に公衆衛生に対する大きな脅威となっている。COVID-19の病気の進行を防ぐ特異的なワクチンや薬剤がないため、罹患率と死亡率は上昇し続けている。この疾患に対する潜在的な治療法を特定し、試験することが急務となっている。治療法の選択肢をより迅速に増やすことにつながる可能性のある一つのアプローチは、現在承認されているFDA(アメリカ食品医薬局)の薬剤を、ウイルスの拡散やCOVID-19病因の重症度を予防または軽減する能力のために再利用することである。FDA(アメリカ食品医薬局)で承認された薬剤の多くは、COVID-19を治療するために臨床で再利用されている。しかしながら、これらの薬剤の有効性およびCOVID-19の症状の予防または重症度の低下に対する特定の標的は、まだ完全には確立されていない2,29,30,31。そのため、いくつかの研究室では、SARS-CoV-2の多くの標的に対する特異的な薬剤を同定している。ここでは、ウイルス複製のための主要な酵素であり、COVID-1932の好ましい薬物標的であるSARS-CoV-2 3CLpro酵素活性を阻害するFDA(アメリカ食品医薬局)承認の47の薬剤を調査した。我々は、3CLproに高い親和性を有する薬剤を選定するためにMOE計算研究を用いて初期スクリーニングを行い、選定された47薬剤の機能阻害活性を試験管内試験酵素アッセイにより確認した。先行研究24,25 で指摘されているように、我々のデータは、S スコアによって定義される計算上のスクリーニングから予測される薬剤の阻害効果が、我々の試験管内試験での実験的研究と一致しないことを示唆している。したがって、すべての薬剤について試験管内試験での追加スクリーニングが必要である。
ここで試験した17種類のPIのうち、ボセプレビル、パリタプレビル、ティプラナビルは、50μMの薬剤濃度で3CLpro酵素活性を部分的に阻害することができた(図2)。ボセプレビルおよびパリタプレビルは、非構造タンパク質3および4A(NS3/4A)のプロテアーゼ活性を阻害することにより、C型肝炎ウイルスを阻害することが示されている33,34。ティプラナビルは、HIVプロテアーゼの活性部位に結合し、それによってウイルスの複製を阻害する成熟した機能性タンパク質に前駆体ポリタンパク質のタンパク質分解的切断を防ぐレトロウイルスプロテアーゼ阻害剤です35。これらの薬剤とは別に、既知のPIであるロピナビルとリトナビルは3CLpro阻害活性を示さなかった(図2);したがって、COVID-19治療の臨床試験でこれらの薬剤の効果がないことを説明する31。また、ロピナビルやリトナビルについては、Maら34と同様の結果が得られた。また、C型肝炎ウイルスの非構造タンパク質(NS5A)を標的にしてウイルスの複製・集合を阻害するombitasvirなどのVNI剤36も、試験管内試験で3CLpro酵素活性を部分的に阻害することができた。抗ウイルス薬に加えて、試験したOTDの一つであるミカファンギンも3CLpro活性の66%を阻害した。ミカファンギンはエキノカンジンのクラスに属し、真菌のβ-1-3グルカン合成酵素37を標的として作用する広汎な抗真菌薬である。
boceprevir、ombitasvir、paritaprevir、tipranavir、およびmicafunginの3CLproに対する部分的な阻害効果の理由は明確には解明されていないが、これらの薬剤と3CLproのCys145/His41/Glu166との間の水素結合の強さが阻害効果の違いを説明している可能性がある。しかし、我々の研究は、これらの分子の足場を利用して、SARS-CoV-2の3CLpro酵素に対して非常に強力で特異的な阻害剤を設計する方法を提供している。
興味深いことに、OTDの一つであるイベルメクチンは、我々の試験管内試験酵素アッセイにおいて、3CLpro活性の85%以上(ほぼ完全に)を阻害することができた(IC50値は21 µM)。これらの知見は、イベルメクチンが SARS-CoV-2 の複製を阻害する可能性を示唆している。これを裏付ける最近の知見として、イベルメクチン(5 µM)がオーストラリアから分離された生きた SARS-CoV-2(VIo1/2020)の Vero/hSLAM 細胞での複製を阻害することが示唆された23。彼らは、DMSO処理した感染細胞と比較して、培養上清(新しいウイルスの放出:93%)だけでなく、細胞内の両方で48時間で> 5000倍のウイルス数が減少したことを発見した(未放出および未アセンブルウイルス:99.8%)。興味深いことに、本研究ではイベルメクチンのIC50値が2.5μM23と報告されているのに対し、我々は21μM(10倍)のIC50値を観測した。報告された IC50 値のばらつきは、ライブウイルスを使用した場合と精製 3CLpro タンパク質を使用した酵素法を使用した場合のようなアッセイ条件の違いに起因している可能性がある。さらに、イベルメクチンの生体内試験での阻害活性とIC50値を検証するために、前臨床試験を実施する必要がある。
イベルメクチンは、ジカ、デング熱、黄熱病、西ナイル、ベネズエラ馬脳炎、チクングニヤ、セムリキ森林、シンドビス、ロクシン生殖呼吸器症候群、ヒト免疫不全症-1ウイルスなどの多くの陽性センス一本鎖RNAウイルスに対して有効であることが知られている38。他のRNAおよびDNAウイルスに対するイベルメクチンの抗ウイルス効果のリストは、最近のレビューにまとめられている38。これまでの研究では、イベルメクチンの抗ウイルス作用のメカニズムは、ウイルスタンパク質とα/β1インポーチン・ヘテロダイマーとの相互作用を阻害することで、ウイルスタンパク質の核への輸送を阻害することであることが示されている19,20,21,22,23。しかし、本研究では、イベルメクチンがSARS-CoV-2 3CLproの酵素活性を阻害し、SARS-CoV-2を含むRNAウイルスの複製を阻害する可能性があることを報告している。これらの研究から、イベルメクチンがSARS-CoV-2の複製を阻害する可能性のある薬剤候補であることが示唆され、図8に示されたイベルメクチンの提案された抗ウイルス機構と、COVID-19に対するイベルメクチンの生体内試験での有効性は、現在臨床試験で評価されている(ClinicalTrials.gov Identifier: NCT04438850)。
図8:イベルメクチンの抗ウイルス機序を模式的に示した図
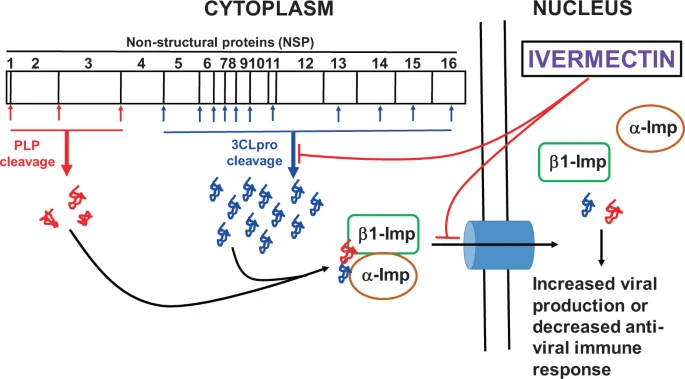
イベルメクチンによるSARS-CoV-2複製の阻害を示す仮説モデル。
結論として,SARS-CoV-2特異的な3CLpro酵素は,ウイルスの複製に大きな役割を果たしていることから,3CLproに対する高い結合親和性を有する薬剤の候補をスクリーニングするためのターゲットとして使用された。その結果、ボセプレビル、ミカファンギン、オンビタスビル、パリタプレビル、ティプラナビルは部分的な阻害効果を示したが、イベルメクチンは試験した用量で試験管内試験でSARS-CoV-2の3CLpro酵素活性を完全に阻害することができた。100nsのMDシミュレーションにより,イベルメクチンの阻害作用には3CLpro酵素のホモ二量体が必要であることが示唆された。これは、隣接するモノマーからのアミノ酸残基Ser1の相互作用によるものと考えられる。一方、ミカファンギンはシミュレーション期間中ずっと3CLproのモノマー型の触媒ポケットに結合したままであった。本研究で報告された薬剤のリストは、SARS-Cov-2の病原体を標的とした臨床試験の前臨床試験の後に、これらの潜在的な薬剤を優先的に試験するための根拠を提供するものである。
方法
試薬・薬剤
分子生物学グレードのDMSOはSigma Aldrich (St. Louis, MO, USA)から、滅菌PBSはThermoFisher Scientific (Waltham, MA, USA)から購入した。3CLpro阻害剤スクリーニング酵素アッセイキット(カタログ番号79955-1)は、BPS Biosciences(サンディエゴ、カリフォルニア州、米国)から購入した。アムプレナビル、アタザナビル硫酸塩、カンジシン、クロロキンリン酸塩、ヒドロロイクロルキン硫酸塩、およびロピナビルは、シグマ・アルドリッチ(ミズーリ州セントルイス)から購入した。ベクラブビル、テムサビルはメドケムエクスプレス(モンマスジャンクション、ニュージャージー州)から購入した。アバカビル(硫酸塩)アルビドール塩酸塩、アスナプレビル、アトロバスタチン、ボセプレビル、ダクラタスビル、ダノプレビル、ダルナビル、デラビルジン(メシル酸塩)エドクスジン、エルバスビル、エルビテグラビル、エトラビリン。ファビピラビル、フマギリン、グレカプレビル、グラゾプレビル、硫酸インジナビル、イトラコナゾール、イベルメクチン、レディパスビル(G-5885)マラビロク、メチルプレドニゾロン、ミカファンギンナトリウム、オンビタスビル。オセルタミビルリン酸塩、パリタプレビル、ペラミビル、ピブレンタスビル、ピモジビル、プレコナリル、ポサコナゾール、キニーネ、ラルテグラビル(カリウム塩)レムデシビル、リバビリン5′-一リン酸塩(リチウム塩)。リボスタマイシン硫酸塩、リルピビリン、サキナビル、テラプレビル、テノホビル二リン酸塩(ナトリウム塩)およびベルパタスビルは、Cayman Chemicals (Ann Arbor, MI)から購入した。
リガンドの作業溶液の調製
薬剤1mgを溶媒としてDMSOまたはPBSのいずれかを用いて8mMのストック溶液を調製した。このストック溶液を用いて、PBS中で250μMおよび500μMの薬剤の作業溶液を調製した。
3CLproタンパク質の調製
COVID-19メインプロテアーゼの結晶構造をタンパク質データバンク(www.rcsb.org)からPDB形式で検索した(ID: 6LU7)。タンパク質に構造上の問題がある場合は、MOEのQuickPrepオプションを使用して修正した。QuickPrepオプションはプロトン化を行い、タンパク質の最小エネルギーコンフォメーションを計算する。QuickPrep機能には、MOEソフトウェアのデフォルトパラメータを使用した。
リガンドの調製
FDA(アメリカ食品医薬局)に承認された3987種類の医薬品分子と有効成分のリストを、SUPERDRUGS2データベースからsdf形式でした。これらの3987種類の薬剤のデータベースをMolecular Operating Environment MOEにインポートし、MOEの洗浄機能を用いて洗浄した。この機能は、プロトン化状態をリバランスし、3次元座標を最小エネルギーのコンフォメーションに再生するものである。洗浄機能にはMOEソフトウェアのデフォルトパラメータを使用した。
タンパク質:薬物ドッキング研究
統合型コンピュータ支援分子設計計算法 Molecular Operating Environment (MOE) ソフトウェアを用いて、3CLpro タンパク質と薬剤をドッキングさせた。簡単に言えば、3987種の薬剤の洗浄済みデータセットを用いて、6LU7プロテアーゼのリガンドサイト上でドッキングを行った。London dGの30ポーズとGBVI/WSA dGの20ポーズを最終的なドッキングに使用した。すべてのドッキング結果は、S Score関数を用いて結合エネルギーでソートし、MOEのリガンド相互作用関数を用いて精度を確認した。結合スコア(S-Score)が 6.5 以下の薬剤を検討対象とした。(1)体内で免疫反応を誘発する傾向があるため、ペプチド模倣薬はすべて除外し、(2)免疫抑制剤として使用される薬剤や心血管疾患の治療に使用される薬剤はリスクが高いため除外した。(3)当グループの試験管内試験アッセイはSARS-COV-2 3CLproに特異的であり、試験管内試験で薬剤を活性化するオプションを提供していないため、活性型が容易に入手可能な場合を除き、すべてのプロドラッグを除外した;(4)半減期が30分未満の短い薬剤も除外した。さらに、現在行われている第I相、第II相、第III相臨床試験の一部である低分子は、S-Scoreに関係なく本試験に含まれていることを確認した。例えば、favipiravirのSスコアは-4.23であり、COVID-19の臨床試験中であるため、favipiravirを本試験に含めることにした。このスクリーニングの結果、56剤をデータベース化し、試験管内試験 3CLpro酵素アッセイを用いてSARS-CoV-2特異的な3CLpro酵素に対する阻害効果を検討した。
3CLpro酵素アッセイ
SARS-CoV-2特異的3CLproアッセイキットをBPS Biosciences (CA)から購入し、96ウェルプレートを用いて製造業者のプロトコルに従って酵素アッセイを行った。簡潔に言うと、30μlのアッセイバッファー中の4 ngの3CLpro-MBPタグ付き酵素を10μlの(250μM)薬剤と1時間プレインキュベートし、10μlの(250μM)蛍光基質を添加することにより酵素反応を開始した。アッセイ試料の最終量は50μlであった。反応混合物中の薬物および基質の最終濃度は50μMであった。インキュベーションは室温で16~18時間継続した。Synergy HT蛍光プレートリーダーを用いて、360/40励起および460/40nm発光で蛍光測定を行った。IC50計算のために、薬物を0〜100μMの用量範囲でスクリーニングした。4 ngの酵素と50 μMの基質を含む1% DMSOを添加したウェルは、酵素阻害を伴わないポジティブコントロールとして機能した。GC367化合物(BPS Biosciences社提供)を50μM添加したウェルは、標準阻害剤およびネガティブコントロールとして機能した。酵素を含まない基質50μMの1%DMSOを添加したウェルはブランクとした。全ての値はブランク値から差し引いた。
分子動力学シミュレーション研究
COVID-19メインプロテアーゼ6LU7 (PDB ID: 6LU7)の構造を、デフォルトのパラメータを使用してMOE2019のQuickPrepアプリケーションを使用して、その単量体および機能的な二量体の形態で調製した。この原子論的モデルは、すべてのMDシミュレーションの入力ファイルを生成するために使用された。
ミカフンジンの最良のドックポーズは、準備されたモノマー3CLプロテアーゼ構造の結合ポケットに配置された。イベルメクチンの最良のポーズは、阻害剤を安定化できる形態をテストするために、モノメリックおよびダイマープロテアーゼ構造に配置された。シミュレーションセルとNAMD 2.1439入力ファイルは、MOE2019を使用して生成した。結晶化水分子は、溶媒和の前に除去した。次に、タンパク質/リガンド複合体を、境界とタンパク質との間に10Åの距離を保ちながら、立方周期の境界条件を有するTIP3P水ボックスに埋め込んだ。タンパク質の正味電荷は100mM NaClで中和した。エネルギー最小化とMDシミュレーションのために、AMBER10:EHT力場を使用し、静電相互作用は粒子メッシュEwald法で評価した。各システムは、急峻降下法と共役勾配法を用いて5000ステップのエネルギー最小化を行った。次に、300Kで100psのNVTアンサンブルを生成し、300Kで200ps、1barのNPTアンサンブルを生成した。次に、各複合体について、さらなる解析のために100nsの生成軌跡を生成した。軌跡の解析は、CCGサポートグループで共有されているスクリプトを用いて行った。
リガンド結合モードの同定
MOE2019のタンパク質リガンド相互作用フィンガープリントアプリケーションを使用して、6LU7のモノマーまたはダイマーに結合した各インヒビターの平均結合モードを調べた。計算は、記録されたMD軌道上のデフォルトパラメータを使用した。
統計と再現性
統計的有意性を比較するために、Dunnettの多重比較ポストホック検定を用いた一方向分散分析(ANOVA)を99.9%の信頼区間で実施し、平均値±SEMとして表した。P値<0.001は統計的に有意と考えられた。IC50値の算出には、4変数の用量対阻害率を用いた非線形回帰(カーブフィット)を用いた。統計解析は、GraphPad Prism(バージョン6.07; La Jolla, CA, USA)を用いて行った。すべての実験は、再現性のためにトリプリケートを用いて最低3回実施し、3回の個別実験の代表を本報告書に記載した。異なる時点で生成されたデータを組み合わせて最終的なグラフを作成した。アッセイを実施した研究者は、アッセイで試験された薬剤の盲検化を行った。
報告書の要約
研究デザインに関する詳細な情報は、この記事にリンクされているNature Research Reporting Summaryに掲載されている。
