Contents
Antigenic Evolution on a Global Scale Reveals the Potential Natural Selection of Severe Acute Respiratory Syndrome-Coronavirus 2 by Pre-existing Cross-Reactive T-Cell Immunity
www.ncbi.nlm.nih.gov/pmc/articles/PMC8169977/
オンラインで2021年5月18日に公開
概要
重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)の突然変異パターンは,世界的な地域感染の中で常に変化している.しかし,変異パターンの変化の理由はまだ明らかになっていない.そこで本研究では、4,420個のアミノ酸変異を有する13,432株のSARS-CoV-2由来の3億個以上のペプチドを包括的に解析し、宿主免疫系の潜在的な選択圧を分析して、循環するSARS-CoV-2分離株の変異のドライバーを明らかにした。
その結果、非構造タンパク質であるORF1abと構造タンパク質であるSpikeが最も変異を受けやすいことがわかった。さらに、SARS-CoV-2と季節性ヒトコロナウイルスとの間の交差反応性T細胞エピトープの変異は、長期的かつ大規模な地域社会での感染下で、SARS-CoV-2が細胞性免疫から逃れるのに役立っている可能性がある。さらに、ホモロジーモデリングとタンパク質ドッキングにより、Spikeタンパク質の変異は、SARS-CoV-2が宿主細胞に侵入し、抗体を介したB細胞免疫から逃れる能力を高める可能性がある。
今回の研究は、自然淘汰されるSARS-CoV-2の潜在的な変異パターンについての洞察を提供し、ウイルスの進化についての理解を深めるとともに、潜在的なワクチン設計のための重要な指針を確立した。
キーワード
重症急性呼吸器症候群-新型コロナウイルス,既存の交差反応性T細胞免疫、ワクチンデザイン、抗原進化、季節性ヒトコロナウイルス
はじめに
重症急性呼吸器症候群コロナウイルス2(SARS-CoV-2)によるコロナウイルス感染症2019(COVID-19)のパンデミックは、今や世界的なパンデミックとなっている(Lu et al 2020; Tang er al)。 SARS-CoV-2の全ゲノム配列は 2020年1月に初めて公開され(Wu et al 2020; Zhou et al 2020)その後、ヒト患者から大量に分離された株が検出された(Shu and McCauley, 2017)。全ゲノム配列の解析(Shu and McCauley, 2017; Lu et al 2020)により、SARS-CoV-2の構造タンパク質と非構造タンパク質の両方にすでに変異が存在し、異なるサブタイプの出現(Tang et al 2020)やSARS-CoV-2の病原性に影響を与えていることが明らかになった。しかし、これらの変異の潜在的な推進要因は特定されておらず、循環しているSARS-CoV-2分離株の進化圧力と病原性を分析するためには、包括的な調査が必要である。
ウイルスの進化は、主に宿主集団間の大規模な感染に影響され、体液性免疫と細胞性免疫の両方を含む宿主免疫系による進化圧力を受けて、変異株が選択的に増殖する機会を与えられる可能性がある(Petrova and Russell, 2018)。ウイルスの主抗原タンパク質を主に標的とする体液性免疫とは異なり、T細胞を介した細胞性免疫は、タンパク質全体からの線状エピトープがHLAによって提示され得るヒト白血球抗原(HLA)制限を認識することで、ウイルス感染に反応することができる(Kindt er al 2007)。HLA対立遺伝子は、世界中の集団において、地理的、民族的な違いによって異なる。したがって、HLA対立遺伝子の多様性や、ウイルス感染に対する感受性と頻繁に関連する遺伝子多型があるため(Setiawan et al 2015,Karimzadeh et al 2019年)T細胞は同じ抗原に対して異なる反応を示す可能性がある。また、集団レベルでのHLAの多様性は、HLA制限T細胞免疫の変動に基づいて、過去6カ月間の世界的な広がりの中でSARS-CoV-2の急速な進化を促した可能性がある。
新たに登場したSARS-CoV-2を含め、7種類のヒトコロナウイルス(HCOV)が報告されている。これらのウイルスのうち、HCOV-229E、HCOV-HKU1,HCOV-NL63,およびHCOV-OC43は、長期間にわたって社会に流通している季節性のHCOVであり、軽度の呼吸器症状を引き起こすだけである(Gaunt er al)。 さらに、複数の研究により、循環している季節性HCVとSARS-CoV-2との間の交差反応性T細胞認識の存在が示唆されている(Bert er al 2020,Grifoni er al 2020,Kissler er al 2020)。SARS-CoV-2の世界的な広がりを示すゲノム配列データにアクセスすることで、免疫系を媒介とした選択圧のメカニズムや、SARS-CoV-2の潜在的な進化の方向性を明らかにすることができるかもしれない。
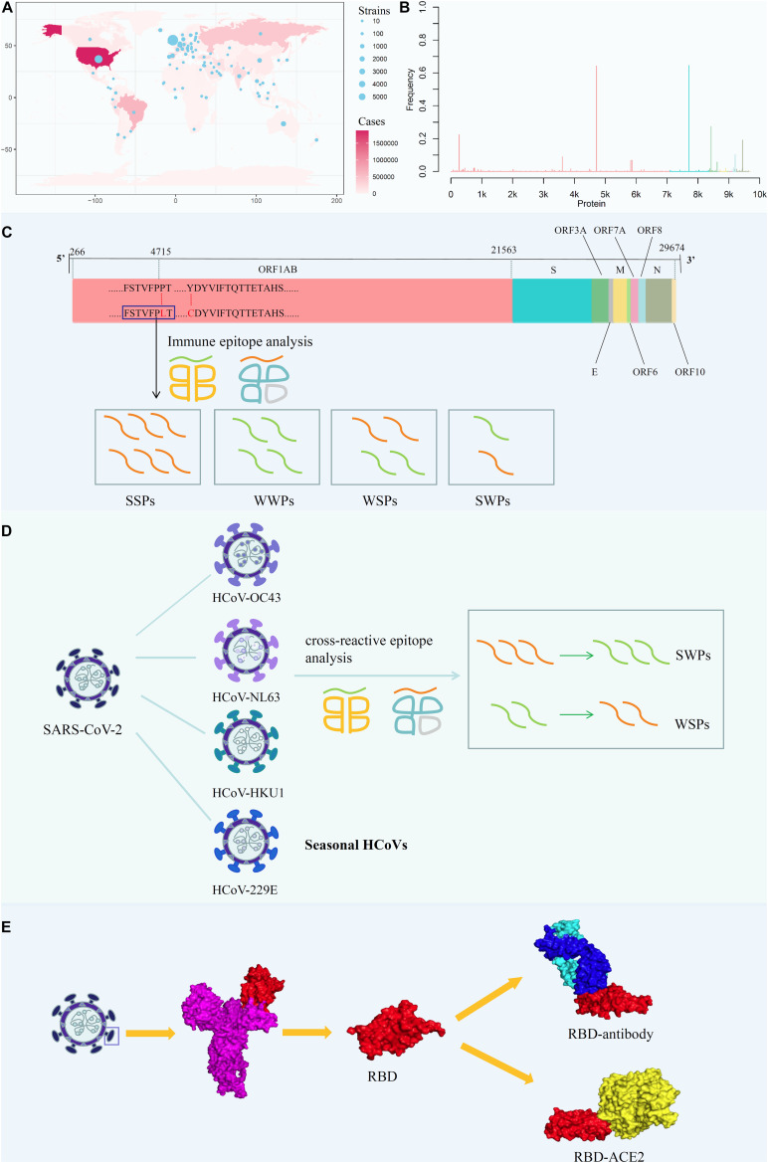
そこで本研究では、SARS-CoV-2の全ゲノム配列上にアミノ酸変異をマッピングし、変異部位を含むすべての潜在的なT細胞エピトープ(PTE)をスクリーニングすること(図1A,B)世界中のウイルスの流通地域と地域の優勢な対立遺伝子に基づいて潜在的なペプチドの免疫原性を分析すること(図1C)など、包括的な分析を行った。季節性HCVとSARS-CoV-2の間の交差反応性エピトープ(CRE)を介したHLAの選択圧の分析(図1D)Sタンパク質変異体のヒトアンジオテンシン変換酵素2(ACE2)と結合抗体に対する結合親和性の評価(図1E)などがある。
図1 SARS-CoV-2全ゲノムの突然変異パターンの解析
(A) 世界規模でSARS-CoV-2の突然変異株を収集。(B) SARS-CoV-2の全ゲノム配列上に突然変異をマッピングした。(C) 全ゲノム配列の異なる構造にある変異を含む潜在的なT細胞エピトープを導き出した。IEDBスタンドアローンツールで予測された結合親和性に応じて、ペプチドを強い結合ペプチドと弱い結合ペプチドに分けた。変異により結合親和性が強いペプチドから弱いペプチドに変化した場合はSWPとカウントし、同様にWSPは弱いペプチドから強いペプチドに変化したことを表する。結合親和性が変わらない場合は、SSPとWWPと表記される。(D) 季節性HCVとSARS-CoV-2の間の交差反応性エピトープの選択圧を、ウイルスの循環領域と地域の優勢な対立遺伝子に応じて明らかにした。ここでは、ローカルブラストを用いて、SARS-CoV-2と他の4つの一般的なHCVの間で同じ配列を持つペプチドを交差反応性エピトープとして導き出した。(E) ヒトACE2と結合抗体CR3022に対するSタンパク質変異体の結合親和性を評価している。2番目のサブグラフは、SARS-CoV-2のSタンパク質の結晶構造である。3番目のサブグラフは、RBD領域の結晶構造である。そして、RBD-ACE2とRBD-mAbの結合複合体も示した。
SARS-CoV-2のORF1abとスパイク(S)タンパク質に頻発する突然変異
その結果、ORF1ab、S、ORF3a、エンベロープ(E)メンブレン(M)ORF6,ORF7a、ORF8,ヌクレオカプシド(N)ORF10の10種類の構造タンパク質および非構造タンパク質に、4,420カ所のアミノ酸の変異が確認された(図2A)。その結果、ORF1ab、S、ORF3a、M、ORF7a、ORF8,Nには、保存領域と非保存領域が存在することがわかった。これらのタンパク質の中でも、ORF1abとSタンパク質は、変異部位の頻度が最も高く、変異頻度が0.6を超えてた(図2A)。各残基の変異頻度とカウント数は、補足表1に記載されている。さらに、大陸別の変異部位をカウントし、S、ORF1ab、N、ORF3aの各タンパク質に発生する頻度の高い変異を特定した。その結果、Sタンパク質のD614Gが9,780回、次いでORF1abのP4715Lが9,745回、Nタンパク質のR203Kが2,866回、ORF1abのP5828Lが1,021回、ORF3aのG251Vが859回となり、上位5つの変異が確認された。上位の変異部位の詳細情報は、補足表2に記載されている。興味深いことに、パンデミックの震源地となったヨーロッパや北アメリカで流通している株から最も高い頻度で変異部位が検出されたことから、免疫圧力がウイルスの進化に寄与している可能性が示唆された。例えば、Sタンパク質のD614G変異とORF1abタンパク質のP4715L変異は、アメリカとイギリスで約3,000回検出された(補足表3)。現在、米国や欧州大陸などの国・地域は、SARS-CoV-2のパンデミックに悩まされており、SARS-CoV-2の高発生地域の一つとなっている。これらの国では、長期にわたる大規模な地域感染が、SARS-CoV-2の全ゲノム上のORF1abとSタンパク質の両方に高い変異頻度をもたらしていることが観察された(Mercatelli and Giorgi, 2020)。Sタンパク質とORF1abに見られる高頻度の変異は、SARS-CoV-2の長期にわたる大規模な地域感染後の体液性免疫と細胞性免疫の選択圧に関係していると考えられる。
図2 SARS-CoV-2の変異プロファイルと潜在的な選択圧の分析
(A)SARS-CoV-2の全ゲノム上の突然変異パターン。棒グラフは、ゲノム配列中の各部位の頻度を示しており、異なる色はゲノム配列の異なる構造を表している。(B) SARS-CoV-2の全ゲノム上のSWPおよびWSPの数。棒グラフは、配列の異なる構造におけるSWPとWSPの数を示している。SWPは強~弱のペプチド、WSPは弱~強のペプチドを表す。(C) Matenusの研究で同定された全てのエピトープの数。棒グラフは、Matenusの研究で同定されたすべてのエピトープの数を示している。ORF1abは多くの非構造タンパク質(NSP1-NSP16)に切断される。S(RBD)はスパイクタンパク質の受容体結合ドメインを表し、S(non-RBD)はスパイクの非RBD部分を表す。N:ヌクレオカプシドタンパク質、E:エンベロープタンパク質、ORF:オープンリーディングフレーム、(D)ORF1ab上の交差反応性のSWPおよびWSPの数。棒グラフは、北米、ヨーロッパ、南アジア、東アジア、東南アジア、オセアニアの6大陸におけるHLA-IとHLA-IIのCREsの数を示している。CREs: cross-reactive epitopes. HLA:human leukocyte antigen (E) Sタンパク質上の変異に対する結合親和性の影響。左のプロットは変異部位の3次元構造の変化を、右の棒グラフはRBDとACE2の間の変異部位のZdockスコアの予測値を示している。
さらに、T細胞クラスI pMHC免疫原性予測ツールを用いて、ORF1abの変異が関与するエピトープを調べた結果を補足表4に示した。その結果、ORF1abの変異を含む86個の9merエピトープのうち、36個のエピトープがポジティブスコアを獲得し、T細胞免疫における免疫原性に影響を与える能力があることが示された。また、我々の研究で予測された変異を含むエピトープを用いて、HLA対立遺伝子DRB1∗01:01およびDRB1∗11:01のT細胞エピトープとしてペプチドLAILTALRLCAYCCNが同定されたことがわかる。この結果は、我々のin silico研究とMatenus Jの最新の実験研究(Mateus er al 2020)の両方と一致しており、変異を伴う予測されたT細胞エピトープが実際にT細胞応答を誘発する可能性を示している。
変異によるT細胞ペプチドの免疫原性の変化
COVID-19の世界的な広がりに伴い、感染者数や罹患国・地域数も増加の一途をたどっている。我々は、Immune Epitope Database(IEDB)が提供するツールに基づいてエピトープ予測を行い、各株の8merから 15merのペプチドを用いてエピトープを選択した。366,286,292個のペプチドが得られ、13,432個のSARS-CoV-2分離株に由来する4,420個のアミノ酸変異が観察された。位置情報を持つ各株について、人口カバー率が支配的なHLA対立遺伝子(対立遺伝子頻度≧0.05)を選択し、IEDBスタンドアローンツールを用いてペプチドの結合親和性を予測した(Moutaftsi et al 2006; Wang et al 2008)。比較のために、SARS-CoV-2の参照配列(MN908947.3)を選択した。その結果、PTE上のほとんどの変異は、予測される結合親和性を変化させないことがわかった(補足表5,6)。結合親和性を変化させる変異については、弱い結合から強い結合へと変化するペプチド(WSP)が、強い結合から弱い結合へと変化するペプチド(SWP)よりも優勢であった。図2Bに示すように、HLA-IとHLA-IIの両方のPTEにおいて、非構造タンパク質と構造タンパク質のほとんどで、WSPがSWPを上回っていた。一方,S proteinでは,HLA-IとHLA-IIのPTEの両方で,SWPがWSPの2.2倍と1.8倍を占めていた。
HLAを介した免疫圧力がSARS-CoV-2の進化を促進した
季節性のHCoVによる感染は、どこにでも存在し、風邪に似た軽い症状を引き起こす(Lidwell and Williams, 1961; Tyrrell, 1965; Gaunt er al)。 疫学的データによると、HCoVは2〜3年ごとに成人に感染する可能性があるとされている(Braun er al 2020)。ヒト集団におけるHCOVの継続的な感染は、免疫記憶の維持を可能にし、HCOVとSARS-CoV-2の間のCREsに対する選択圧を生み出す。HCoVに対するT細胞の記憶免疫がSARS-CoV-2の進化に果たす役割を探るため、COVID-19のパンデミック前にすでに地域社会で流通していた可能性のあるSARS-CoV-2とHCOVの間のCREsを分析した。Matenus Jの最近の研究(Mateus et al 2020)では、交差反応性のSARS-CoV-2 T細胞エピトープが非暴露のヒトでも検出されることが証明されており、風邪の原因となるコロナウイルスに対するT細胞の記憶が多様化していることが、COVID-19疾患で観察された広範な不均一性の少なくとも一部の原因である可能性を示している。ここでは、Matenus Jの研究で得られたT細胞エピトープを全ゲノム上にマッピングし、変異を伴うエピトープの分布を示した(補足表7および図2C)。エピトープは、非RBD領域とRBD領域の両方を含む、SARS-CoV-2ゲノム全体のSタンパク質にほとんど濃縮されていることがわかる。これは、ORF1abの他に、Sタンパク質に最も多くの変異を伴うT細胞エピトープが含まれているという我々の結果と一致する(補足表1)。T細胞エピトープの提示は、HLAタイプと有意に関連している(Hoof et al 2009)。そこで、ペプチドとMHC分子との結合親和性を予測する前に、HLA対立遺伝子をAllele Frequency Net Database (Gonzalez-Galarza et al 2020) から導き出し、異なる地域の主要なHLA対立遺伝子を頻度に基づいてスクリーニングした。各地域では、地域人口の5%以上に記録されたHLA対立遺伝子を導き出し、さらなる分析を行った(補足表8)。オリジナルの感染地域からの情報と優勢なHLA-I/HLA-II対立遺伝子をマッチングさせることにより、7/4大陸の13/11カ国からそれぞれ197/358対立遺伝子と9,327/943ペプチドが得られ、さらなる分析が可能となった。それぞれの CRE について,HLA-I/HLA-II と対応する対立遺伝子との結合親和性を IEBD MHC-I/MHC-II 結合予測ツールで予測した。
ORF1abタンパク質では、変異を含むすべてのペプチドについて、WSPの数はSWPの1.6倍であった(Supplementary Table 9)。しかし、CREsの数を解析すると、逆に、北米、ヨーロッパ、南アジア、東アジア、東南アジア、オセアニアの6つの大陸では、交差反応性のSWPの方が交差反応性のWSPよりも有意に多かった(図2C)。例えば、北米では、HLA-Iの交差反応性SWP(34,800件)は、WSP(1,733件)の20.1倍の頻度で検出された。また、ヨーロッパ、南アジア、東アジア、東南アジア、オセアニアでも同様の結果が得られ、交差反応性のHLA-I SWPの数はWSPの1.75~4倍であった。頻度が10以上のSWPを補足表10に示した。HLA-IIでは、北米、欧州、東アジア、南アジアでのみCREが検出され、交差反応性のHLA-II SWPが2,206個検出され、WSP(546個)の4倍であった。北米・欧州・東アジアでは,クロスリアクティブなHLA-II SWPの数はWSPの数の2.4~19.4倍であった。一方,南アジアのデータでは,クロスリアクティブなHLA-II WSPが13個検出された。これは,南アジアから提出された配列データが少なかったため,SWPの数よりも多かったと考えられる.これらの結果は、既存の交差反応性T細胞免疫による自然な選択圧が、SARS-CoV-2の進化の方向性を後押しし、ウイルスが免疫モニタリングから逃れることを可能にした可能性を示している。さらに、これがランダムな現象であるかどうかを評価するために、全ゲノム中の交差反応性ペプチド(CRP)とCRP以外に出現したすべてのペプチドを用いて統計解析を行った。補足表5,6に示されているように、CRP以外のすべての出現ペプチドにおいて、WSPはSWPよりも有意に高い値を示した。例えば、MHC Iでは13回、MHC IIでは4回となっている。逆に、SWPの数はWSPの数よりも多く、MHC IとMHC IIでそれぞれ約14倍と4倍であった。Pearsonのカイ二乗検定によると、両検定のp値は2.2e-16以下であり、既存の交差反応性T細胞免疫によるSARS-CoV-2の自然選択の可能性を示している。
Sタンパク質の変異により、ACE2との結合容量が増加し、抗体との親和性が低下した
SARS-CoV-2のSタンパク質は体液性免疫の主要な標的であり、Sタンパク質の受容体結合ドメイン(RBD)は、宿主細胞の表面受容体へのウイルスの付着を媒介する(Song et al 2004,Kirchdoerfer et al 2016,Schoeman and Fielding 2019)。また、SARS-CoV-2の主要な抗原性タンパク質であるSタンパク質上の変異を結合解析で評価した。ACE2-RBD結合およびmAb-RBD結合解析の両方が、タンパク質ドッキングアプローチによって提供された。その結果、RBD上の変異は、mAbとRBDの間の結合親和性を低下させ、一方で、ヒトACE2とRBDの間の結合親和性を高める傾向があることが示された。つまり、Sタンパク質上の変異は、免疫系に対する抗原性を低下させるだけでなく、ヒトACE2との結合親和性を高めることで、ウイルスの感染力を増加させる可能性があるということである。同様に、最新の実験では、D614G変異のように、RBD上の変異がSARS-CoV-2の感染力を大幅に増加させる可能性も指摘されている(Li er al 2020)。ここでは、Sタンパク質のACE2結合ドメインおよびエピトープ領域で発生した変異の影響を評価した。ACE2結合ドメインに変異があるR408I、N439K、G476Sなどの3つの変異体(図2D)と、エピトープ領域に変異があるK378R、H519P、H519Q、R408Iなどの4つの変異体(図2E)の立体構造を、ホモロジーモデリング(Webb and Sali, 2008)によって構築した。さらに、対応する変異体とACE2(Lan et al 2020)またはCR3022抗体(Yuan et al 2020)との間の結合親和性を、分子ドッキング(Pierce et al 2014)によって計算した。コントロールとして、SARS-CoV-2分離株(MN908947.3)のRBD領域をリファレンスとして選択した。
変異体R408I,N439K,G476Sは,リファレンスと比較して,RBD領域とACE2の間の結合を促進した(図2E)。アルギニン(R)とリジン(K)はアルカリ性アミノ酸であり、Rをイソロイシン(I)に、アスパラギン(N)をKに変異させると、タンパク質の特性が大きく変化することがわかった。さらに調べてみると、N439Kの潜在的な結合部位は酸性アミノ酸のグルタミン酸(329E)であり、これがイオン結合を形成してRBDとACE2の間の結合能力を高める可能性があることがわかった(図2E)。さらに、R408Iの潜在的な結合部位はアルカリ性アミノ酸のヒスチジン(H)であり、変異R408Iは2つのアルカリ性アミノ酸HとRの間の反発力を低下させる可能性があった(図2E)。
変異K378R、H519P、H519Q、R408Iは、Sタンパク質のCR3022抗体に対する結合親和性を低下させる可能性がある(図2E)。アルカリ性アミノ酸の間に位置する変異K378Rに加えて、他の3つの変異はすべてアルカリ性アミノ酸が非電荷残基に変異したものであった。注目すべきは、408RがH鎖の54Dにあるアスパラギン酸(D)とイオン結合を形成していることである。RをIに変異させると、このイオン結合が切れて、抗体への結合親和性が低下する。以上の結果から、Sタンパク質のRBD上の変異は、ウイルスがACE2受容体を標的とする能力を高め、宿主細胞への侵入能力をさらに促進する可能性があることがわかった。さらに、RBD上の変異は、抗体との結合親和性を低下させ、ウイルスの免疫逃避を引き起こす可能性がある。
さらに、最近の複数の研究で、RBDを利用したワクチンがT細胞応答を誘発することが示されている。例えば、Ugur Sahin氏の研究(Sahin et al 2020)では、nRBDをベースにしたCOVID-19ワクチンによって、ウイルス特異的なCD4+ T細胞とCD8+ T細胞の活性化が観察された。また、別の研究では、スパイクタンパクに対する6ヶ月後のT細胞ELISpot応答の大きさは、スパイクタンパクとRBDドメインの両方に対するピーク抗体レベルの大きさと強い相関があることが示された(Zuo et al 2021)。このように、RBDドメインの線状エピトープは、T細胞免疫を誘導する可能性があり、また、RBDの変異は、T細胞免疫を誘導する能力を変化させ、T細胞応答を変化させる可能性がある。
考察
結論として、我々は、ウイルスの自然選択の可能性と関連する結果を分析するために、循環地域内の各SARS-CoV-2分離株の全ゲノム上に観察されたすべての変異をマッピングした。その結果、北米やヨーロッパなどの大陸で長期的かつ大規模な地域感染が起きていると、ORF1abとSタンパク質の両方に高い変異頻度が生じることがわかった。したがって、循環しているSARS-CoV-2は、宿主の免疫系から強い選択圧を受けている可能性があり、突然変異によって全ゲノム上のSWPの数に比べてWSPの数が増加する可能性がある。しかし、季節性HCVとSARS-CoV-2の間のCREについては、ORF1abタンパク質上のWSPの数よりもSWPの数が有意に多く、SARS-CoV-2が既存の交差反応性T細胞免疫によって自然淘汰される可能性を示していた。最後に、SARS-CoV-2のSタンパク質は、N439Kなどの変異により新たなイオン結合を形成してヒトの受容体であるACE2との結合親和性を高めるだけでなく、R408Iの変異によりイオン結合を破壊してCR3022抗体との結合親和性を低下させる可能性があることを発見した。この結果から、Sタンパク質は、抗体を介したB細胞の免疫圧力下で、感染力を高め、免疫モニタリングから逃れる進化的傾向を示している可能性が示唆された。最近の研究(Kissler et al 2020)によると、SARS-CoV-2は予想よりもはるかに長く拡散する可能性が高いという。
最近の研究では、突然変異がSARS-CoV-2に対するワクチンの効果に大きく影響する可能性が指摘されている。Sタンパク質上のウイルスの突然変異や組み換えイベントは、第一世代のワクチンの効果を低下させたり否定したりする可能性がある(Poland er al)。 さらに、Sタンパク質上の変異が免疫を回避することができれば、ワクチン開発が妨害される可能性がある(Poland er al 2020b)。さらに、南アフリカの変異株のような証拠があれば、ワクチンを接種したヒトの防御効率が低下する可能性がある(Wang et al 2021)。以上の結果から、SARS-CoV-2の変異、特にSタンパク質の変異は、現在開発されているワクチンの効率を低下させる可能性があることがわかった。SARS-CoV-2とインフルエンザウイルスはともにRNAウイルスであり、タンパク質の発現はウイルスのRNAポリメラーゼに依存しているが、SARS-CoV-2には校正機構があるため、インフルエンザウイルスよりも変異速度が遅い。そのため、SARS-CoV2のワクチンや、回復した患者で発達した免疫は、インフルエンザウイルスよりも長期的な保護を提供できる可能性がある(Manzanares-Meza and Medina-Contreras, 2020)。しかし、現在の最良の知見によれば、さらなる変異や、さらには組み換え事象が確認されることは事実上確実であり(Poland er al 2020a)防御的なSARS-CoV-2ワクチンを開発することは依然として大きな課題であるか、あるいは、インフルエンザウイルスのように定期的にSARS-CoV-2ワクチンを更新する必要があるかもしれない。
この研究は、もし免疫記憶が短期的にしか有効でないならば、季節性HCVと同様に、2~3年ごとに毎年または周期的に発生するリスクがあることを示唆している(Kissler er al)。 これと一致するように、我々の研究では、SARS-CoV-2の急速な変異は、免疫システムのモニタリングや認識から逃れる傾向があることが示された。このことは、仮に現在流通しているSARS-CoV-2に対して有効なワクチンが開発できたとしても、急速な免疫逃避を促す変異によって、短期間でワクチンが効かなくなる可能性があることを示唆している。したがって、SARS-CoV-2のワクチン開発は、インフルエンザウイルスのように周期的に行われる可能性があることを示唆している。このような状況下では、SARS-CoV-2の突然変異と抗原の進化をモニタリングする必要がある。